+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1015 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
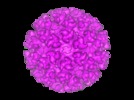
| タイトル | Cryo-electron microscopy reveals the functional organization of an enveloped virus, Semliki Forest virus. | |||||||||
 マップデータ マップデータ | Semliki Forest virus. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報togavirin / T=4 icosahedral viral capsid / virion assembly / small molecule binding / host cell endosome / clathrin-dependent endocytosis of virus by host cell / symbiont-mediated suppression of host toll-like receptor signaling pathway / host cell cytoplasm / viral translational frameshifting / serine-type endopeptidase activity ...togavirin / T=4 icosahedral viral capsid / virion assembly / small molecule binding / host cell endosome / clathrin-dependent endocytosis of virus by host cell / symbiont-mediated suppression of host toll-like receptor signaling pathway / host cell cytoplasm / viral translational frameshifting / serine-type endopeptidase activity / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / host cell nucleus / host cell plasma membrane / virion membrane / structural molecule activity / proteolysis / RNA binding / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Semliki forest virus (セムリキ森林ウイルス) Semliki forest virus (セムリキ森林ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.0 Å | |||||||||
 データ登録者 データ登録者 | Mancini EJ / Clarke M / Gowen B / Rutten T / Fuller SD | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2000 ジャーナル: Mol Cell / 年: 2000タイトル: Cryo-electron microscopy reveals the functional organization of an enveloped virus, Semliki Forest virus. 著者: E J Mancini / M Clarke / B E Gowen / T Rutten / S D Fuller /  要旨: Semliki Forest virus serves as a paradigm for membrane fusion and assembly. Our icosahedral reconstruction combined 5276 particle images from 48 cryo-electron micrographs and determined the virion ...Semliki Forest virus serves as a paradigm for membrane fusion and assembly. Our icosahedral reconstruction combined 5276 particle images from 48 cryo-electron micrographs and determined the virion structure to 9 A resolution. The improved resolution of this map reveals an N-terminal arm linking capsid subunits and defines the spike-capsid interaction sites. It illustrates the paired helical nature of the transmembrane segments and the elongated structures connecting them to the spike projecting domains. A 10 A diameter density in the fusion protein lines the cavity at the center of the spike. These clearly visible features combine with the variation in order between the layers to provide a framework for understanding the structural changes during the life cycle of an enveloped virus. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1015.map.gz emd_1015.map.gz | 26.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1015-v30.xml emd-1015-v30.xml emd-1015.xml emd-1015.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1015.gif 1015.gif | 37.3 KB | ||
| その他 |  emd_1015_additional_1.map emd_1015_additional_1.map emd_1015_additional_2.map emd_1015_additional_2.map emd_1015_additional_3.map emd_1015_additional_3.map | 354.9 KB 354.9 KB 354.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1015 http://ftp.pdbj.org/pub/emdb/structures/EMD-1015 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1015 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1015 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1015.map.gz / 形式: CCP4 / 大きさ: 50.8 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) ダウンロード / ファイル: emd_1015.map.gz / 形式: CCP4 / 大きさ: 50.8 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Semliki Forest virus. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.52 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 1015 additional 1.map
| ファイル | emd_1015_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-添付マップデータ: emd 1015 additional 2.map
| ファイル | emd_1015_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-添付マップデータ: emd 1015 additional 3.map
| ファイル | emd_1015_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Semliki Forest Virus
| 全体 | 名称:   Semliki Forest Virus (セムリキ森林ウイルス) Semliki Forest Virus (セムリキ森林ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1000: Semliki Forest Virus
| 超分子 | 名称: Semliki Forest Virus / タイプ: sample / ID: 1000 / 集合状態: T=4 membraneous particle / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 50 MDa / 手法: theoretical |
-超分子 #1: Semliki forest virus
| 超分子 | 名称: Semliki forest virus / タイプ: virus / ID: 1 / Name.synonym: SFV / NCBI-ID: 11033 / 生物種: Semliki forest virus / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No / Syn species name: SFV |
|---|---|
| 宿主 | 生物種: baby hamster kidney 21 cells (unknown) / 別称: VERTEBRATES |
| ウイルス殻 | Shell ID: 1 / 名称: envelope / T番号(三角分割数): 4 |
| ウイルス殻 | Shell ID: 2 / 名称: nucleocapsid / T番号(三角分割数): 4 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: Tris (10mM) NaCl (100 mM) ph 7.4 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 37 K / 装置: HOMEMADE PLUNGER 詳細: Vitrification instrument: EMBL plunger with warm humid air spray. plunging at ambient temperature and humidity 手法: Blot for 2 sec Graticule grids were used to maintain flatness |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG/ST |
|---|---|
| 温度 | 平均: 105 K |
| 日付 | 1995年1月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 48 / 平均電子線量: 8 e/Å2 / カメラ長: 44 / Od range: 1 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 7.628 µm / 最小 デフォーカス(公称値): 0.975 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: ctf multiplication and summation of normalized reconstructions |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 9.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMBL-ICOS 詳細: final maps were calculated by making a normalized sum of seperate ctf multiplied maps: Baker, T. S., Olson, N. H., and Fuller, S. D. (1999). Adding the third dimension to virus life cycles: ...詳細: final maps were calculated by making a normalized sum of seperate ctf multiplied maps: Baker, T. S., Olson, N. H., and Fuller, S. D. (1999). Adding the third dimension to virus life cycles: Three-Dimensional Reconstruction of Icosahedral Viruses from Cryo-Electron Micrographs. Microbiology and Molecular Biology Reviews 63, 862-922. Fuller, S. D., Butcher, S. J., Cheng, R. H., and Baker, T. S. (1996). Three-dimensional reconstruction of icosahedral particles-the uncommon line. J Struct Biol 116, 48-55. Mancini, E. J., Clarke, M., Gowen, B., Rutten, T., and Fuller, S. D. (2000). Cryo-electron microscopy reveals the functional organization of an enveloped virus, Semliki Forest virus. Molecular Cell 5, 255-266. Mancini, E. J., de Haas, F., and Fuller, S. D. (1997). High-resolution icosahedral reconstruction: fulfilling the promise of cryo-electron microscopy. Structure 5, 741-750. 使用した粒子像数: 6000 |
| 最終 角度割当 | 詳細: sufficient to give maximum inverse eigenvalue of 0.1 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D |
|---|---|
| ソフトウェア | 名称: emfit (Cheng et al 1995) |
| 詳細 | Protocol: rigid body. The capsid protein used for the rigid body refinement was PDB entry 1VCQ |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: R factor and clashes |
| 得られたモデル |  PDB-1dyl:  PDB-2xfc: |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)













































