+Search query
-Structure paper
| Title | Molecular basis for glycan recognition and reaction priming of eukaryotic oligosaccharyltransferase. |
|---|---|
| Journal, issue, pages | Nat Commun, Vol. 13, Issue 1, Page 7296, Year 2022 |
| Publish date | Nov 26, 2022 |
 Authors Authors | Ana S Ramírez / Mario de Capitani / Giorgio Pesciullesi / Julia Kowal / Joël S Bloch / Rossitza N Irobalieva / Jean-Louis Reymond / Markus Aebi / Kaspar P Locher /  |
| PubMed Abstract | Oligosaccharyltransferase (OST) is the central enzyme of N-linked protein glycosylation. It catalyzes the transfer of a pre-assembled glycan, GlcNAcManGlc, from a dolichyl-pyrophosphate donor to ...Oligosaccharyltransferase (OST) is the central enzyme of N-linked protein glycosylation. It catalyzes the transfer of a pre-assembled glycan, GlcNAcManGlc, from a dolichyl-pyrophosphate donor to acceptor sites in secretory proteins in the lumen of the endoplasmic reticulum. Precise recognition of the fully assembled glycan by OST is essential for the subsequent quality control steps of glycoprotein biosynthesis. However, the molecular basis of the OST-donor glycan interaction is unknown. Here we present cryo-EM structures of S. cerevisiae OST in distinct functional states. Our findings reveal that the terminal glucoses (Glc) of a chemo-enzymatically generated donor glycan analog bind to a pocket formed by the non-catalytic subunits WBP1 and OST2. We further find that binding either donor or acceptor substrate leads to distinct primed states of OST, where subsequent binding of the other substrate triggers conformational changes required for catalysis. This alternate priming allows OST to efficiently process closely spaced N-glycosylation sites. |
 External links External links |  Nat Commun / Nat Commun /  PubMed:36435935 / PubMed:36435935 /  PubMed Central PubMed Central |
| Methods | EM (single particle) |
| Resolution | 2.8 - 3.1 Å |
| Structure data | EMDB-15419, PDB-8agb: EMDB-15420, PDB-8agc: EMDB-15421, PDB-8age: |
| Chemicals |  ChemComp-CPL:  ChemComp-PTY:  ChemComp-MN: 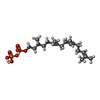 ChemComp-ELU:  ChemComp-NAG: 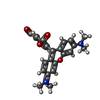 ChemComp-323: 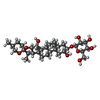 ChemComp-KZB:  ChemComp-HOH: |
| Source |
|
 Keywords Keywords | TRANSFERASE / N-glycosylation OST complex |
 Movie
Movie Controller
Controller Structure viewers
Structure viewers About Yorodumi Papers
About Yorodumi Papers










