| タイトル | Bifurcated assembly pathway and dual function of a Lon-like protease revealed by cryo-EM Analysis |
|---|
| ジャーナル・号・ページ | Fundam Res, Year 2024 |
|---|
| 掲載日 | 2023年7月31日 (構造データの登録日) |
|---|
 著者 著者 | Li M / Liu H / Hsieh KY / Zhang S / Gao Y / Gong Q / Zhang K / Chang CI / Li S |
|---|
 リンク リンク |  Fundam Res / PubMedで検索 Fundam Res / PubMedで検索 |
|---|
| 手法 | EM (単粒子) |
|---|
| 解像度 | 2.54 - 4.5 Å |
|---|
| 構造データ | EMDB-36965, PDB-8k8v:
CryoEM structure of LonC protease hepatmer, apo state
手法: EM (単粒子) / 解像度: 2.75 Å EMDB-36966, PDB-8k8w:
CryoEM structure of LonC protease open hexamer, apo state
手法: EM (単粒子) / 解像度: 3.46 Å EMDB-36968, PDB-8k8y:
CryoEM structure of LonC heptamer in presence of AGS
手法: EM (単粒子) / 解像度: 2.54 Å EMDB-36969, PDB-8k8z:
CryoEM structure of LonC protease hexamer in presence of AGS
手法: EM (単粒子) / 解像度: 2.83 Å EMDB-36970, PDB-8k90:
CryoEM structure of LonC protease open pentamer in presence of AGS
手法: EM (単粒子) / 解像度: 3.43 Å EMDB-36971, PDB-8k91:
CryoEM structure of LonC S582A hepatmer with Lysozyme
手法: EM (単粒子) / 解像度: 3.03 Å EMDB-36972, PDB-8k92:
CryoEM structure of LonC S582A hexamer with Lysozyme
手法: EM (単粒子) / 解像度: 3.31 Å EMDB-36973, PDB-8k93:
CryoEM structure of LonC protease S582A open hexamer with lysozyme
手法: EM (単粒子) / 解像度: 3.23 Å EMDB-36974, PDB-8k94:
CryoEM structure of LonC protease S582A open pentamer with lysozyme
手法: EM (単粒子) / 解像度: 3.4 Å EMDB-36975, PDB-8k95:
CryoEM structure of LonC protease open Hexamer, AGS
手法: EM (単粒子) / 解像度: 3.05 Å EMDB-36976, PDB-8k96:
CryoEM structure of LonC protease hepatmer with Bortezomib
手法: EM (単粒子) / 解像度: 2.89 Å EMDB-36977, PDB-8k97:
CryoEM structure of LonC protease hexamer with Bortezomib
手法: EM (単粒子) / 解像度: 2.73 Å EMDB-36978: CryoEM map of LonC protease open hexamer with Bortezomib
手法: EM (単粒子) / 解像度: 4.5 Å EMDB-36979: CryoEM map of LonC protease heptamer (heated at 343K)
手法: EM (単粒子) / 解像度: 3.65 Å |
|---|
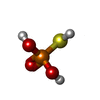
| 化合物 | ChemComp-BO2:
N-[(1R)-1-(DIHYDROXYBORYL)-3-METHYLBUTYL]-N-(PYRAZIN-2-YLCARBONYL)-L-PHENYLALANINAMIDE / ボルテゾミブ / 薬剤, 抗がん剤*YM
|
|---|
| 由来 |  meiothermus taiwanensis (バクテリア) meiothermus taiwanensis (バクテリア)
|
|---|
 キーワード キーワード | CHAPERONE / Lon proteases / hepatmer / open hexamer / open pentamer / heptamer / AGS / hexatmer / Lysozyme / hexamer / inhibitor |
|---|
 著者
著者 リンク
リンク Fundam Res / PubMedで検索
Fundam Res / PubMedで検索


 キーワード
キーワード ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について





























 meiothermus taiwanensis (バクテリア)
meiothermus taiwanensis (バクテリア)