+検索条件
-Structure paper
| タイトル | Dynamic molecular architecture and substrate recruitment of cullin3-RING E3 ligase CRL3. |
|---|---|
| ジャーナル・号・ページ | Nat Struct Mol Biol, Vol. 31, Issue 2, Page 336-350, Year 2024 |
| 掲載日 | 2024年2月8日 |
 著者 著者 | Yuxia Hu / Zhao Zhang / Qiyu Mao / Xiang Zhang / Aihua Hao / Yu Xun / Yeda Wang / Lin Han / Wuqiang Zhan / Qianying Liu / Yue Yin / Chao Peng / Eva Marie Y Moresco / Zhenguo Chen / Bruce Beutler / Lei Sun /   |
| PubMed 要旨 | Phosphatidylinositol 3-kinase α, a heterodimer of catalytic p110α and one of five regulatory subunits, mediates insulin- and insulin like growth factor-signaling and, frequently, oncogenesis. ...Phosphatidylinositol 3-kinase α, a heterodimer of catalytic p110α and one of five regulatory subunits, mediates insulin- and insulin like growth factor-signaling and, frequently, oncogenesis. Cellular levels of the regulatory p85α subunit are tightly controlled by regulated proteasomal degradation. In adipose tissue and growth plates, failure of K48-linked p85α ubiquitination causes diabetes, lipodystrophy and dwarfism in mice, as in humans with SHORT syndrome. Here we elucidated the structures of the key ubiquitin ligase complexes regulating p85α availability. Specificity is provided by the substrate receptor KBTBD2, which recruits p85α to the cullin3-RING E3 ubiquitin ligase (CRL3). CRL3 forms multimers, which disassemble into dimers upon substrate binding (CRL3-p85α) and/or neddylation by the activator NEDD8 (CRL3~N8), leading to p85α ubiquitination and degradation. Deactivation involves dissociation of NEDD8 mediated by the COP9 signalosome and displacement of KBTBD2 by the inhibitor CAND1. The hereby identified structural basis of p85α regulation opens the way to better understanding disturbances of glucose regulation, growth and cancer. |
 リンク リンク |  Nat Struct Mol Biol / Nat Struct Mol Biol /  PubMed:38332366 PubMed:38332366 |
| 手法 | EM (単粒子) |
| 解像度 | 3.76 - 12.18 Å |
| 構造データ | EMDB-34199, PDB-8gq6: EMDB-34449, PDB-8h33: EMDB-34450, PDB-8h34: EMDB-34451, PDB-8h35: EMDB-34452, PDB-8h36: EMDB-34453, PDB-8h37:  EMDB-34454: local refinement of KBTBD2-p85a: KBTBD2-CRL3-p85a complex EMDB-34455, PDB-8h38:  EMDB-34456: local refinement of CSN1/2/4/5/6-CRL3/CTD-N8: KBTBD2-CRL3-CSN(mutate) complex  EMDB-34457: local refinement of CSN1/3/5/6/7/8: KBTBD2-CRL3-CSN(mutate) complex  EMDB-34458: local refinement of CSN5/6-CRL3/CTD-N8: KBTBD2-CRL3-CSN(mutate) complex 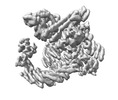 EMDB-34459: local refinement of KBTBD2-CRL3-CSN(mutate) complex  EMDB-34460: Cryo-EM Structure of the KBTBD2-CRL3~N8-CSN(mutate,no CSN3/8) complex  EMDB-34461: Cryo-EM Structure of the KBTBD2-CRL3~N8-CSN(mutate)-CSN(mutate,no CSN3/8) complex EMDB-34462, PDB-8h3a:  EMDB-34463: Cryo-EM Structure of the KBTBD2-CRL3~N8(removed)-CSN(no CSN3/8) complex 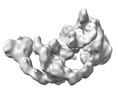 EMDB-34466: Cryo-EM Structure of the KBTBD2-CRL3-CSN(no CSN3/8) complex EMDB-34467, PDB-8h3f: EMDB-34473, PDB-8h3q: EMDB-34474, PDB-8h3r: |
| 化合物 |  ChemComp-ZN: |
| 由来 |
|
 キーワード キーワード |  LIGASE (リガーゼ) / LIGASE (リガーゼ) /  complex complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について