+検索条件
-Structure paper
| タイトル | Human CTP synthase filament structure reveals the active enzyme conformation. |
|---|---|
| ジャーナル・号・ページ | Nat Struct Mol Biol, Vol. 24, Issue 6, Page 507-514, Year 2017 |
| 掲載日 | 2017年5月1日 |
 著者 著者 | Eric M Lynch / Derrick R Hicks / Matthew Shepherd / James A Endrizzi / Allison Maker / Jesse M Hansen / Rachael M Barry / Zemer Gitai / Enoch P Baldwin / Justin M Kollman /   |
| PubMed 要旨 | The universally conserved enzyme CTP synthase (CTPS) forms filaments in bacteria and eukaryotes. In bacteria, polymerization inhibits CTPS activity and is required for nucleotide homeostasis. Here we ...The universally conserved enzyme CTP synthase (CTPS) forms filaments in bacteria and eukaryotes. In bacteria, polymerization inhibits CTPS activity and is required for nucleotide homeostasis. Here we show that for human CTPS, polymerization increases catalytic activity. The cryo-EM structures of bacterial and human CTPS filaments differ considerably in overall architecture and in the conformation of the CTPS protomer, explaining the divergent consequences of polymerization on activity. The structure of human CTPS filament, the first structure of the full-length human enzyme, reveals a novel active conformation. The filament structures elucidate allosteric mechanisms of assembly and regulation that rely on a conserved conformational equilibrium. The findings may provide a mechanism for increasing human CTPS activity in response to metabolic state and challenge the assumption that metabolic filaments are generally storage forms of inactive enzymes. Allosteric regulation of CTPS polymerization by ligands likely represents a fundamental mechanism underlying assembly of other metabolic filaments. |
 リンク リンク |  Nat Struct Mol Biol / Nat Struct Mol Biol /  PubMed:28459447 / PubMed:28459447 /  PubMed Central PubMed Central |
| 手法 | EM (らせん対称) / EM (単粒子) / X線回折 |
| 解像度 | 2.7 - 17.0 Å |
| 構造データ |  EMDB-8476:  EMDB-8490:  EMDB-8491: EMDB-8504, PDB-5u3c: EMDB-8513, PDB-5u6r:  PDB-5tkv: |
| 化合物 | 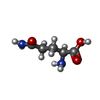 ChemComp-GLN:  ChemComp-SO4:  ChemComp-MPD:  ChemComp-MG:  ChemComp-CTP:  ChemComp-MRD:  ChemComp-HOH:  ChemComp-ATP:  ChemComp-UTP:  ChemComp-ADP: |
| 由来 |
|
 キーワード キーワード |  LYASE (リアーゼ) / LYASE (リアーゼ) /  PYRIMIDINE BIOSYNTHESIS / ENZYME REGULATION VIA POLYMERIZATION / PYRIMIDINE BIOSYNTHESIS / ENZYME REGULATION VIA POLYMERIZATION /  FEEDBACK INHIBITION (酵素阻害剤) / FEEDBACK INHIBITION (酵素阻害剤) /  LIGASE (リガーゼ) / PROTEIN FIBRIL / LIGASE (リガーゼ) / PROTEIN FIBRIL /  nucleotide metabolism (ヌクレオチド) / nucleotide metabolism (ヌクレオチド) /  enzyme (酵素) / enzyme (酵素) /  filament / active / metabolic filament filament / active / metabolic filament |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN文献について
EMN文献について














