+検索条件
-Structure paper
| タイトル | How Pol α-primase is targeted to replisomes to prime eukaryotic DNA replication. |
|---|---|
| ジャーナル・号・ページ | Mol Cell, Vol. 83, Issue 16, Page 2911-2924.e16, Year 2023 |
| 掲載日 | 2023年8月17日 |
 著者 著者 | Morgan L Jones / Valentina Aria / Yasemin Baris / Joseph T P Yeeles /  |
| PubMed 要旨 | During eukaryotic DNA replication, Pol α-primase generates primers at replication origins to start leading-strand synthesis and every few hundred nucleotides during discontinuous lagging-strand ...During eukaryotic DNA replication, Pol α-primase generates primers at replication origins to start leading-strand synthesis and every few hundred nucleotides during discontinuous lagging-strand replication. How Pol α-primase is targeted to replication forks to prime DNA synthesis is not fully understood. Here, by determining cryoelectron microscopy (cryo-EM) structures of budding yeast and human replisomes containing Pol α-primase, we reveal a conserved mechanism for the coordination of priming by the replisome. Pol α-primase binds directly to the leading edge of the CMG (CDC45-MCM-GINS) replicative helicase via a complex interaction network. The non-catalytic PRIM2/Pri2 subunit forms two interfaces with CMG that are critical for in vitro DNA replication and yeast cell growth. These interactions position the primase catalytic subunit PRIM1/Pri1 directly above the exit channel for lagging-strand template single-stranded DNA (ssDNA), revealing why priming occurs efficiently only on the lagging-strand template and elucidating a mechanism for Pol α-primase to overcome competition from RPA to initiate primer synthesis. |
 リンク リンク |  Mol Cell / Mol Cell /  PubMed:37506699 / PubMed:37506699 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.98 - 8.75098 Å |
| 構造データ |  EMDB-15303: S. cerevisiae pol alpha bound to the core replisome engaged with a fork DNA substrate containing a 60 nucleotide lagging strand - unbinned. 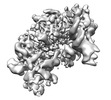 EMDB-15304: Csm3/Tof1 local refinement - pol alpha bound replisome 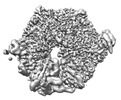 EMDB-15305: C-tier local refinement, pol alpha - replisome complex. Locally filtered  EMDB-15306: Pol12, PolA1 and Pri2 N-terminal domain local refinement. Pol alpha - replisome complex. EMDB-15309: Pol alpha - replisome complex containing Ctf4. Engaged on a DNA fork containing a 60 nucleotide lagging strand.  EMDB-15310: Ctf4 - GINS - Cdc45 local refinement. Pol alpha - replisome complex.  EMDB-15340: MCM C-tier local refinement. Human replisome bound by pol alpha, engaged on fork DNA with 60 nt lagging strand. EMDB-15341: Human replisome bound by pol alpha, engaged on fork DNA containing a 60 nt lagging strand. 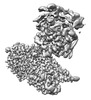 EMDB-15342: AND-1, CDC45, GINS local refinement. Human replisome bound by pol alpha - engaged on a 60 nt lagging strand DNA fork substrate.  EMDB-15349: Human replisome bound by Pol Alpha, locally filtered. Strong PRIM1 density. Engaged on a fork DNA substrate containing a 60 nt lagging strand. 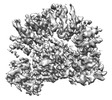 EMDB-15356: TIMELESS/TIPIN local refinement. Human replisome bound by Pol Alpha engaged on fork DNA with a 60 nt lagging strand.  EMDB-15902: S. cerevisiae replisome + Ctf4 + pol alpha. Unbinned, locally filtered. 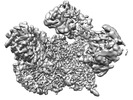 EMDB-15904: Composite map - human replisome + Pol alpha  EMDB-15918: Human replisome + pol alpha. Not engaged on DNA.  EMDB-15922: Human replisome bound to pol alpha engaged on a DNA fork substrate containing a 15 nucleotide lagging strand.  EMDB-15923: Human replisome prepared in the presence of a fork DNA substrate containing a 60 nucleotide lagging strand. Off DNA. EMDB-15924, PDB-8b9c:  EMDB-16247: Local refinement, Pri1:Pri2-CTD. Pol alpha primase associated budding yeast replisome. 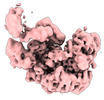 EMDB-16248: Pol alpha primase associated budding yeast replisome. Consensus refinement containing clear density for the Pri2-CTD and Pol1 exo/cat domains. 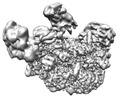 EMDB-16320: Pol-alpha primase associated replisome binned consensus refinement in the absence of Ctf4, 60 nucleotide 5'-flap DNA fork  EMDB-16322: Pol alpha-primase associated replisome unbinned consensus refinement in the absence of Ctf4, 60 nucleotide 5'-flap DNA fork  EMDB-16323: Un-binned consensus refinement of Pol alpha-primase associated with the replisome only via the Pri2:Mcm5ZnF interface, 60 nucleotide 5'-flap DNA fork  EMDB-16885: Pol12:Pol1CTD local refinement |
| 化合物 |  ChemComp-ANP:  ChemComp-MG:  ChemComp-ZN: |
| 由来 |
|
 キーワード キーワード | REPLICATION / helicase / polymerase / pol alpha / priming / replisome / human |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について









 homo sapiens (ヒト)
homo sapiens (ヒト)