-検索条件
-検索結果
検索 (著者・登録者: kay & grünewald)の結果108件中、1から50件目までを表示しています
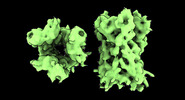
EMDB-17704: 
Subtomogram average of Vaccinia A10 trimer with open center from in vitro cores
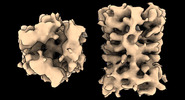
EMDB-17708: 
Subtomogram average of Vaccinia A10 trimer with tight center from in vitro cores

EMDB-17753: 
Subtomogram average of Vaccinia A10 trimer from in situ cores

EMDB-42250: 
Varicella-zoster virus glycoprotein B; H527P prefusion class I.

EMDB-42251: 
Varicella-zoster virus glycoprotein B; H527P prefusion mutant class II.

EMDB-16670: 
Structure of the SNV L protein bound to 5' RNA

PDB-8ci5: 
Structure of the SNV L protein bound to 5' RNA

EMDB-15532: 
Plasmodium falciparum sporozoite subpellicular microtubule with interrupted luminal helix determined in situ

EMDB-15534: 
Plasmodium falciparum gametocyte subpellicular microtubule with 13 protofilaments determined in situ

EMDB-15535: 
Plasmodium falciparum gametocyte subpellicular microtubule with 14 protofilaments determined in situ

EMDB-15536: 
Plasmodium falciparum gametocyte subpellicular microtubule with 15 protofilaments determined in situ

EMDB-15537: 
Plasmodium falciparum gametocyte subpellicular microtubule with 16 protofilaments determined in situ

EMDB-15538: 
Plasmodium falciparum gametocyte subpellicular microtubule with 17 protofilaments determined in situ

EMDB-15539: 
Plasmodium falciparum gametocyte subpellicular microtubule with 18 protofilaments determined in situ

EMDB-15607: 
Structure of the SFTSV L protein bound to 5' cRNA hook [5' HOOK]

EMDB-15608: 
Structure of the SFTSV L protein stalled at early elongation [EARLY-ELONGATION]

EMDB-15610: 
Structure of the SFTSV L protein stalled at early elongation with the endonuclease domain in a raised conformation [EARLY-ELONGATION-ENDO]

EMDB-15614: 
Structure of the SFTSV L protein stalled at late elongation [LATE-ELONGATION]

EMDB-15615: 
Structure of the SFTSV L protein bound in a resting state [RESTING]

PDB-8as6: 
Structure of the SFTSV L protein bound to 5' cRNA hook [5' HOOK]

PDB-8as7: 
Structure of the SFTSV L protein stalled at early elongation [EARLY-ELONGATION]

PDB-8asb: 
Structure of the SFTSV L protein stalled at early elongation with the endonuclease domain in a raised conformation [EARLY-ELONGATION-ENDO]

PDB-8asd: 
Structure of the SFTSV L protein stalled at late elongation [LATE-ELONGATION]

PDB-8asg: 
Structure of the SFTSV L protein bound in a resting state [RESTING]

EMDB-15627: 
Tomogram of the Mimivirus genomic fiber

EMDB-15628: 
Tomogram of the Mimivirus genomic fiber

EMDB-15629: 
Tomogram of the Mimivirus genomic fiber

EMDB-15630: 
Tomogram of the Mimivirus genomic fiber

EMDB-13641: 
Structure of the Mimivirus genomic fibre asymmetric unit

EMDB-14353: 
Structure of the Mimivirus genomic fibre in its compact 6-start helix form

EMDB-14354: 
Structure of the Mimivirus genomic fibre in its compact 5-start helix form

EMDB-14355: 
Structure of the Mimivirus genomic fibre in its relaxed 5-start helix form

PDB-7ptv: 
Structure of the Mimivirus genomic fibre asymmetric unit

PDB-7yx3: 
Structure of the Mimivirus genomic fibre in its compact 6-start helix form

PDB-7yx4: 
Structure of the Mimivirus genomic fibre in its compact 5-start helix form

PDB-7yx5: 
Structure of the Mimivirus genomic fibre in its relaxed 5-start helix form

EMDB-14167: 
Amyloid fibril from the antimicrobial peptide uperin 3.5

EMDB-14168: 
Amyloid fibril from the antimicrobial peptide aurein 3.3

PDB-7qv5: 
Amyloid fibril from the antimicrobial peptide uperin 3.5

PDB-7qv6: 
Amyloid fibril from the antimicrobial peptide aurein 3.3

EMDB-12807: 
Apo-structure of Lassa virus L protein (well-resolved polymerase core) [APO-CORE]

EMDB-12860: 
Apo-structure of Lassa virus L protein (well-resolved endonuclease) [APO-ENDO]

EMDB-12861: 
Apo-structure of Lassa virus L protein (well-resolved alpha ribbon) [APO-RIBBON]

EMDB-12862: 
Lassa virus L protein bound to 3' promoter RNA (well-resolved polymerase core) [3END-CORE]

EMDB-12863: 
Lassa virus L protein bound to 3' promoter RNA (well-resolved endonuclease) [3END-ENDO]

EMDB-12953: 
Lassa virus L protein with endonuclease and C-terminal domains in close proximity [MID-LINK]

EMDB-12954: 
Lassa virus L protein bound to the distal promoter duplex [DISTAL-PROMOTER]

EMDB-12955: 
Lassa virus L protein in a pre-initiation conformation [PREINITIATION]

EMDB-12956: 
Lassa virus L protein in an elongation conformation [ELONGATION]

PDB-7och: 
Apo-structure of Lassa virus L protein (well-resolved polymerase core) [APO-CORE]
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
