-検索条件
-検索結果
検索 (著者・登録者: van & den & berg & b)の結果176件中、1から50件目までを表示しています

EMDB-45597: 
Cryo-EM structure of the human ether-a-go-go related K+ channel (hERG) in 300 mM K+

EMDB-45598: 
Cryo-EM structure of the human ether-a-go-go related K+ channel (hERG) in 3 mM K+

EMDB-45599: 
Cryo-EM structure of the human ether-a-go-go related K+ channel (hERG) in 300 mM K+ without symmetry

EMDB-45600: 
Cryo-EM structure of the human ether-a-go-go related K+ channel (hERG) in 3 mM K+ without symmetry

PDB-9chp: 
Cryo-EM structure of the human ether-a-go-go related K+ channel (hERG) in 300 mM K+

PDB-9chq: 
Cryo-EM structure of the human ether-a-go-go related K+ channel (hERG) in 3 mM K+

PDB-9chr: 
Cryo-EM structure of the human ether-a-go-go related K+ channel (hERG) in 300 mM K+ without symmetry

PDB-9chs: 
Cryo-EM structure of the human ether-a-go-go related K+ channel (hERG) in 3 mM K+ without symmetry
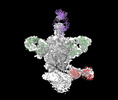
EMDB-44246: 
Cryo-EM structure of HIV-1 JRFL v6 Env in complex with vaccine-elicited, Membrane Proximal External Region (MPER) directed antibody DH1317.4.

EMDB-18592: 
E.coli DNA gyrase in complex with 217 bp substrate DNA and LEI-800

PDB-8qqi: 
E.coli DNA gyrase in complex with 217 bp substrate DNA and LEI-800

EMDB-17377: 
Structure of human SIT1 (focussed map / refinement)

EMDB-17378: 
Structure of human SIT1:ACE2 complex (open PD conformation)

EMDB-17379: 
Structure of human SIT1:ACE2 complex (closed PD conformation)

EMDB-17380: 
Structure of human SIT1 bound to L-pipecolate (focussed map / refinement)

EMDB-17381: 
Structure of human SIT1:ACE2 complex (open PD conformation) bound to L-pipecolate

EMDB-17382: 
Structure of human SIT1:ACE2 complex (closed PD conformation) bound to L-pipecolate

PDB-8p2w: 
Structure of human SIT1 (focussed map / refinement)

PDB-8p2x: 
Structure of human SIT1:ACE2 complex (open PD conformation)

PDB-8p2y: 
Structure of human SIT1:ACE2 complex (closed PD conformation)

PDB-8p2z: 
Structure of human SIT1 bound to L-pipecolate (focussed map / refinement)

PDB-8p30: 
Structure of human SIT1:ACE2 complex (open PD conformation) bound to L-pipecolate

PDB-8p31: 
Structure of human SIT1:ACE2 complex (closed PD conformation) bound to L-pipecolate
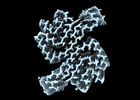
EMDB-19986: 
Structure of recombinant alpha-synuclein fibrils 1B capable of seeding GCIs in vivo

PDB-9euu: 
Structure of recombinant alpha-synuclein fibrils 1B capable of seeding GCIs in vivo

EMDB-16328: 
Outer membrane attachment porin OmpM1 from Veillonella parvula

EMDB-16332: 
Outer membrane attachment porin OmpM1 from Veillonella parvula, native

EMDB-16333: 
Outer membrane attachment porin OmpM1 from Veillonella parvula, C3 symmetry

PDB-8bym: 
Outer membrane attachment porin OmpM1 from Veillonella parvula

PDB-8bys: 
Outer membrane attachment porin OmpM1 from Veillonella parvula, native

PDB-8byt: 
Outer membrane attachment porin OmpM1 from Veillonella parvula, C3 symmetry

EMDB-40334: 
Human OCT1 (Apo) in inward-open conformation

EMDB-40335: 
Human OCT1 bound to diltiazem in inward-open conformation

EMDB-40336: 
Human OCT1 bound to fenoterol in inward-open conformation

EMDB-40337: 
Human OCT1 bound to metformin in inward-open conformation

EMDB-40339: 
Human OCT1 bound to thiamine in inward-open conformation

PDB-8sc1: 
Human OCT1 (Apo) in inward-open conformation

PDB-8sc2: 
Human OCT1 bound to diltiazem in inward-open conformation

PDB-8sc3: 
Human OCT1 bound to fenoterol in inward-open conformation

PDB-8sc4: 
Human OCT1 bound to metformin in inward-open conformation

PDB-8sc6: 
Human OCT1 bound to thiamine in inward-open conformation

EMDB-16114: 
Vitamin B12 transporter BtuB1 with lipoprotein BtuG1 from B. theta

EMDB-17574: 
BtuB3G3 bound to cyanocobalamin with disordered EL8

EMDB-17575: 
BtuB3G3 bound to cyanocobalamin with ordered EL8

PDB-8blw: 
Vitamin B12 transporter BtuB1 with lipoprotein BtuG1 from B. theta

PDB-8p97: 
BtuB3G3 bound to cyanocobalamin with disordered EL8

PDB-8p98: 
BtuB3G3 bound to cyanocobalamin with ordered EL8

EMDB-15288: 
Substrate-free levan utilisation machinery (utilisome)

EMDB-15289: 
Levan utilisation machinery (utilisome) with levan fructo-oligosaccharides DP 8-12

EMDB-15290: 
Core SusCD transporter units from the levan utilisome with levan fructo-oligosaccharides DP 8-12
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
