-検索条件
-検索結果
検索 (著者・登録者: young & mj)の結果88件中、1から50件目までを表示しています
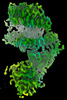
EMDB-29530: 
SARS-CoV-2 XBB.1 spike RBD bound to the human ACE2 ectodomain and the S309 neutralizing antibody Fab fragment
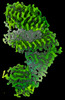
EMDB-29531: 
SARS-CoV-2 BQ.1.1 spike RBD bound to the human ACE2 ectodomain and the S309 neutralizing antibody Fab fragment
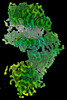
EMDB-40240: 
SARS-CoV-2 BN.1 spike RBD bound to the human ACE2 ectodomain and the S309 neutralizing antibody Fab fragment

EMDB-28036: 
Cryo-EM structure of the full-length human NF1 dimer

EMDB-40253: 
Truncated Braf/Mek/14-3-3 complex

EMDB-27428: 
Cryo-EM structure of a RAS/RAF complex (state 1)
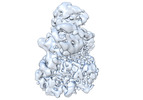
EMDB-27429: 
Cryo-EM structure of a RAS/RAF complex (state 2)

EMDB-27826: 
Cryo-EM structure of the full-length human NF1 dimer

EMDB-27777: 
Streptomyces venezuelae RNAP transcription open promoter complex with WhiA and WhiB transcription factors

EMDB-27778: 
Streptomyces venezuelae RNAP unconstrained open promoter complex with WhiA and WhiB transcription factors
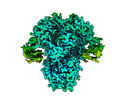
EMDB-26727: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the proximal conformation
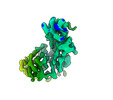
EMDB-26729: 
CCoV-HuPn-2018 S in the proximal conformation (local refinement of domain 0)
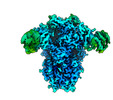
EMDB-26730: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the swung out conformation
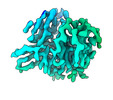
EMDB-26731: 
CCoV-HuPn-2018 S in the swung out conformation (local refinement of domain 0)

EMDB-26507: 
SARS-CoV-2 spike in complex with Multivalent miniprotein inhibitor FUS231-P24 (2RBDs open)

EMDB-26508: 
SARS-CoV-2 spike in complex with multivalent miniprotein inhibitor FUS231-P24 (3RBDs open)

EMDB-26509: 
SARS-CoV-2 spike in complex with multivalent miniprotein inhibitor FUS31-G10 (2RBDs open)

EMDB-26510: 
SARS-CoV-2 spike in complex with multivalent miniprotein inhibitor FUS31-G10 (3RBDs open)

EMDB-26511: 
SARS-CoV-2 spike in complex with AHB2-2GS-SB175 (local refinement of the RBD and AHB2)

EMDB-26512: 
SARS-CoV-2 spike in complex with AHB2-2GS-SB175

EMDB-24346: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-80 IgG

EMDB-24348: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-081 Fab

EMDB-24349: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-091 IgG

EMDB-24350: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-094 VHH-Fc

EMDB-24351: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-096 IgG

EMDB-24358: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-147 IgG

EMDB-24359: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-246 IgG

EMDB-24335: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-259 IgG

EMDB-24336: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-186 IgG

EMDB-24337: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-199 IgG

EMDB-24338: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-249 IgG

EMDB-24339: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-252 IgG

EMDB-24340: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-049 IgG

EMDB-24341: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-073 scFv

EMDB-24342: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-043 IgG

EMDB-24343: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-010 IgG

EMDB-24344: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-148 IgG

EMDB-24345: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-002 Fab

EMDB-24352: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-250 Fab

EMDB-24353: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-063 scFv

EMDB-24354: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-021 Fab

EMDB-24355: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-247 IgG

EMDB-24356: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-020 Fab

EMDB-24357: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-090 IgG

EMDB-24360: 
EM map of SARS-CoV-2 Spike in complex with CoVIC-245 IgG

EMDB-24361: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-074 IgG

EMDB-24383: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-140 IgG

EMDB-24384: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-134 IgG

EMDB-24388: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-038 IgG

EMDB-24293: 
Negative stain EM map of SARS-CoV-2 Spike in complex with human ACE2
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
