-検索条件
-検索結果
検索 (著者・登録者: ye & md)の結果150件中、1から50件目までを表示しています

EMDB-44587: 
Cryo-EM Structure of the Helicobacter pylori dcagT PR

EMDB-44496: 
Cryo-EM co-structure of AcrB with the CU032 efflux pump inhibitor

EMDB-44500: 
Cryo-EM co-structure of AcrB with the EPM35 efflux pump inhibitor

EMDB-44501: 
Cryo-EM co-structure of AcrB with the CU232 efflux pump inhibitor
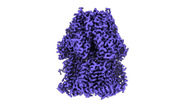
EMDB-44506: 
Cryo-EM co-structure of AcrB with CU244

EMDB-40677: 
Structural insights into cellular control of the human CPEB3 prion, functionally regulated by a labile-amyloid-forming segment

EMDB-42290: 
Cryo-EM Structure of the Helicobacter pylori CagYdAP OMC

EMDB-42393: 
Cryo-EM Structure of the Helicobacter pylori dcagM PR

EMDB-42392: 
Cryo-EM Structure of the Helicobacter pylori cagYdAP PR

EMDB-41171: 
Cryo-EM structure of cardiac amyloid fibril from a variant ATTR I84S amyloidosis patient-3, wild-type morphology

EMDB-41172: 
Cryo-EM structure of cardiac amyloid fibril from a variant ATTR I84S amyloidosis patient-3, variant-type morphology

EMDB-27641: 
The structure of the interleukin 11 signalling complex, truncated gp130

EMDB-27642: 
The structure of the IL-11 signalling complex, with full-length extracellular gp130

EMDB-41109: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1

EMDB-41113: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1

EMDB-41259: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1
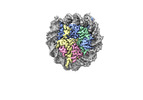
EMDB-41272: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1

EMDB-28825: 
Human CCC complex

EMDB-28827: 
Human CCC complex

EMDB-27323: 
Cardiac amyloid fibrils extracted from a variant ATTR I84S amyloidosis patient (2).

EMDB-26685: 
Cardiac amyloid fibrils extracted from a variant ATTR I84S amyloidosis patient

EMDB-28092: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-093

EMDB-28090: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-040

EMDB-28091: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-045
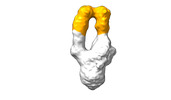
EMDB-28093: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-156

EMDB-34806: 
SARS-CoV-2 Delta Spike in complex with FP-12A

EMDB-34807: 
SARS-CoV-2 Delta Spike in complex with IS-9A

EMDB-34808: 
SARS-CoV-2 Omicron BA.1 Spike in complex with IY-2A

PDB-8hhx: 
SARS-CoV-2 Delta Spike in complex with FP-12A

PDB-8hhy: 
SARS-CoV-2 Delta Spike in complex with IS-9A

PDB-8hhz: 
SARS-CoV-2 Omicron BA.1 Spike in complex with IY-2A
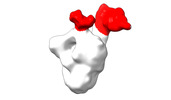
EMDB-28094: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-234

EMDB-28095: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-260

EMDB-28096: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-279

EMDB-28097: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-290
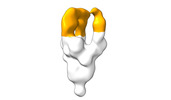
EMDB-28098: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-294

EMDB-28099: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-295

EMDB-28100: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-299

EMDB-28102: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-334
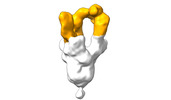
EMDB-28103: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-360

EMDB-28104: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-361

EMDB-28105: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-362

EMDB-28106: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-368

EMDB-28168: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-292

EMDB-28169: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-333
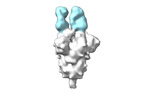
EMDB-28170: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-355

EMDB-28171: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-371

EMDB-15542: 
Human mitochondrial ribosome small subunit in complex with streptomycin

EMDB-27502: 
Cryo-EM structure of SARS-CoV-2 Alpha (B.1.1.7) spike protein

EMDB-27503: 
Cryo-EM structure of SARS-CoV-2 Alpha (B.1.1.7) spike protein in complex with human ACE2
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
