-検索条件
-検索結果
検索 (著者・登録者: ye & k)の結果5,344件中、1から50件目までを表示しています

EMDB-43551: 
CCHFV GP38 bound with ADI-46143 and ADI-46158 Fabs

EMDB-43552: 
CCHFV GP38 bound with ADI-58062 and ADI-63530 Fabs

EMDB-43553: 
CCHFV GP38 bound with ADI-58026 and ADI-63547 Fabs

EMDB-43604: 
CCHFV GP38 bound to ADI-46152 and ADI-58048 Fabs

PDB-8vww: 
CCHFV GP38 bound to ADI-46152 and ADI-58048 Fabs

EMDB-18990: 
CryoEM map of tau PHF sarkosyl-extracted from a human AD patient (associated with in situ tomography)

EMDB-18402: 
cryo-EM structure of apo-TcdB

EMDB-18403: 
cryo-EM map of apo Clostridioides difficile toxin B

EMDB-18409: 
cryo-EM structure of TcdB-FZD7

EMDB-18410: 
cryo-EM structure of TcdB-FZD7

EMDB-18411: 
cryo-EM structure of TcdB-FZD7

EMDB-43011: 
Phosphorylated, ATP-bound, E1371Q human cystic fibrosis transmembrane conductance regulator (E1371Q-CFTR)

EMDB-43014: 
Phosphorylated, ATP-bound, inhibitor 172-bound E1371Q human cystic fibrosis transmembrane conductance regulator

EMDB-41256: 
Cryotomogram of DIV 4 cultured hippocampal neuron
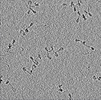
EMDB-41257: 
Purified arrayed human chromatin

EMDB-41258: 
In vitro assembled actin and cofilactin filaments

EMDB-41263: 
Purified alpha-CaMKII Holoenzymes

EMDB-41264: 
DIV 1 hippocampal neuron (weighted back-projection and greyscale segmentation)

EMDB-45655: 
Cryo-EM structure of alpha5beta1 integrin in complex with NeoNectin

PDB-9ckv: 
Cryo-EM structure of alpha5beta1 integrin in complex with NeoNectin

EMDB-50148: 
Tau PHF subtomogram average relating to CS1 extended data Figure 9A

EMDB-50152: 
Tau PHF subtomogram average relating to CS2 Figure 3i-j.

EMDB-50153: 
Tau PHF subtomogram average relating to CS3 extended data Figure 9c

EMDB-50155: 
Tau PHF subtomogram average relating to CS4 extended data Figure 9d

EMDB-50156: 
Tau PHF subtomogram average relating to CS5 extended data Figure 9b

EMDB-50157: 
Tau PHF subtomogram average relating to CS6 extended data Figure 9e

EMDB-50159: 
Tau PHF subtomogram average relating to CS7 extended data Figure 9f

EMDB-50160: 
Tau PHF subtomogram average relating to LOL1_PHF Figure 4g-h

EMDB-50161: 
Tau SF subtomogram average relating to LOL1_SF Figure 4g-h

EMDB-50162: 
Tau SF subtomogram average relating to LOL2_SF Figure 4i-j

EMDB-50580: 
SOLIST cryo-tomogram of native left ventricle mouse heart muscle #1

EMDB-50582: 
SOLIST native mouse heart muscle tomogram #2

EMDB-17691: 
60-meric complex of dihydrolipoamide acetyltransferase (E2) of the human pyruvate dehydrogenase complex (icosahedral symmetry)
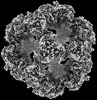
EMDB-17694: 
60-meric complex of dihydrolipoamide acetyltransferase (E2) of the human pyruvate dehydrogenase complex (tetrahedral symmetry)

EMDB-18616: 
E2/E3BP core of the human pyruvate dehydrogenase complex (map 1; 3.4 A)

EMDB-18617: 
E2/E3BP core of the human pyruvate dehydrogenase complex (map 2; 3.7 A)

PDB-8piu: 
60-meric complex of dihydrolipoamide acetyltransferase (E2) of the human pyruvate dehydrogenase complex

EMDB-41433: 
Escherichia coli RNA polymerase unwinding intermediate (I1a) at the lambda PR promoter

EMDB-41437: 
Escherichia coli RNA polymerase unwinding intermediate (I1d) at the lambda PR promoter

EMDB-41439: 
Escherichia coli RNA polymerase unwinding intermediate (I1b) at the lambda PR promoter

EMDB-41448: 
Escherichia coli RNA polymerase unwinding intermediate (I1c) at the lambda PR promoter

EMDB-41456: 
Escherichia coli RNA polymerase closed complex intermediate at the lambda PR promoter

PDB-8to1: 
Escherichia coli RNA polymerase unwinding intermediate (I1a) at the lambda PR promoter

PDB-8to6: 
Escherichia coli RNA polymerase unwinding intermediate (I1d) at the lambda PR promoter

PDB-8to8: 
Escherichia coli RNA polymerase unwinding intermediate (I1b) at the lambda PR promoter

PDB-8toe: 
Escherichia coli RNA polymerase unwinding intermediate (I1c) at the lambda PR promoter

PDB-8tom: 
Escherichia coli RNA polymerase closed complex intermediate at the lambda PR promoter
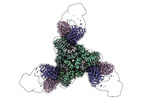
EMDB-42527: 
Pre-fusion Measles virus fusion protein complexed with Fab 77
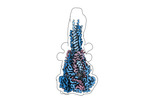
EMDB-42539: 
Structure of the Measles virus Fusion protein in the post-fusion conformation
EMDB-42593: 
Structure of the Measles virus Fusion protein in the pre-fusion conformation
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
