-検索条件
-検索結果
検索 (著者・登録者: yan & jj)の結果166件中、1から50件目までを表示しています

EMDB-18180: 
cryoEM structure of SARS-CoV2 Spike trimer in complex with Fab23

PDB-8q5y: 
cryoEM structure of SARS-CoV2 Spike trimer in complex with Fab23

EMDB-43475: 
Human GABAA receptor alpha1-beta2-gamma2 subtype in complex with GABA plus methaqualone

EMDB-43485: 
Human GABAA receptor alpha1-beta2-gamma2 subtype in complex with GABA plus PPTQ

EMDB-40046: 
CryoEM structure of Influenza A virus A/Melbourner/1/1946 (H1N1) hemagglutinin bound to GS10-X6-BE4 Fab

EMDB-38216: 
Cryo-EM structure of SARS-CoV-2 S-BQ.1 in complex with antibody O5C2

EMDB-35827: 
Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate

EMDB-37652: 
Structure of CbCas9 bound to 6-nucleotide complementary DNA substrate

EMDB-37656: 
Structure of CbCas9-PcrIIC1 complex bound to 28-bp DNA substrate (20-nt complementary)

EMDB-37657: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (symmetric 20-nt complementary)

EMDB-37762: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (non-targeting complex)
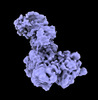
EMDB-40261: 
DDB1/CRBN in complex with ARV-471 and the ER ligand-binding domain

EMDB-42676: 
5-HT2AR bound to Lisuride in complex with a mini-Gq protein and an active-state stabilizing single-chain variable fragment (scFv16) obtained by cryo-electron microscopy (cryoEM)

EMDB-42999: 
5HT2AR-miniGq heterotrimer in complex with a novel agonist obtained from large scale docking

EMDB-36776: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron HYER1 at symmetric apo state

EMDB-36777: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron DR1 at symmetric pre-cleavage state

EMDB-36778: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron HYER1 at symmetric post cleavge state

EMDB-36786: 
The Streptococcus azizii ORF-less Group IIC intron HYER2 at apo state

EMDB-36779: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron HYER1 with 10-nt TRS at symmetric apo state

EMDB-43008: 
Fab fragment of human mAb #58 in complex with computationally optimized broadly reactive H1 influenza hemagglutinin X6

EMDB-37295: 
Cryo-EM structure of the yeast TOM core complex crosslinked by BS3 (from TOM-TIM23 complex)

EMDB-37294: 
Cryo-EM structure of the yeast TOM core complex (from TOM-TIM23 complex)

EMDB-36057: 
Cryo-EM map of DAM (a newly designed immunogen for monkeypox virus) in complex with half of the antigen-binding fragment (Fab) of A27D7 and the single-chain fragment variable (scFv) of 7D11

EMDB-35985: 
Cryo-EM structure of starch degradation complex of BAM1-LSF1-MDH

EMDB-36056: 
Cryo-EM map of half of DAM (the A35 part) in complex with the antigen-binding fragment (Fab) of A27D7

EMDB-37429: 
Cryo-EM structure of the SEP363856-bound mTAAR1-Gs complex

EMDB-37430: 
Cryo-EM structure of the ZH8651-bound mTAAR1-Gs complex

EMDB-37431: 
Cryo-EM structure of the TMA-bound mTAAR1-Gs complex

EMDB-37432: 
Cryo-EM structure of the PEA-bound mTAAR1-Gs complex

EMDB-37433: 
Cryo-EM structure of the ZH8667-bound mTAAR1-Gs complex

EMDB-37434: 
Cryo-EM structure of the ZH8651-bound hTAAR1-Gs complex

EMDB-37435: 
Cryo-EM structure of the ZH8651-bound mTAAR1-Gq complex

EMDB-37436: 
Cryo-EM structure of the PEA-bound hTAAR1-Gs complex

EMDB-37437: 
Cryo-EM structure of the CHA-bound mTAAR1-Gq complex

EMDB-37438: 
Cryo-EM structure of the CHA-bound mTAAR1 complex

EMDB-28833: 
Cryo-EM structure of X6 COBRA (H1N1) hemagglutinin bound to CR6261 Fab

EMDB-35347: 
Structure of R2 with 3'UTR and DNA in binding state

EMDB-35348: 
Structure of R2 with 3'UTR and DNA in unwinding state

EMDB-35349: 
Structure of R2 with 5'ORF

EMDB-35350: 
Structure of R2 with 5'ORF and 3'UTR

EMDB-26855: 
Arabidopsis DDM1 bound to nucleosome (H2A.W, H2B, H3.3, H4, with 147 bp DNA)

EMDB-29930: 
T. cruzi topoisomerase II alpha bound to dsDNA and the covalent inhibitor CT1

PDB-8gcc: 
T. cruzi topoisomerase II alpha bound to dsDNA and the covalent inhibitor CT1
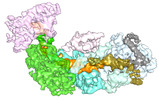
EMDB-27257: 
Apo gRAMP

EMDB-27259: 
gRAMP-match PFS target
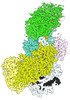
EMDB-27260: 
gRAMP-TPR-CHAT (Craspase)

EMDB-27261: 
gRAMP-TPR-CHAT Non match PFS target RNA(Craspase)
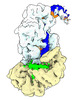
EMDB-27262: 
gRAMP-TPR-CHAT match PFS target RNA(Craspase)
EMDB-27263: 
gRAMP non-matching PFS-with Mg

EMDB-29857: 
Molecular mechanism of nucleotide inhibition of human uncoupling protein 1
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
