+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of the CHA-bound mTAAR1 complex | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | CHA / mTAAR1 / MEMBRANE PROTEIN | |||||||||
| Function / homology |  Function and homology information Function and homology informationG protein-coupled amine receptor activity / Amine ligand-binding receptors / trace-amine receptor activity / G alpha (s) signalling events / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / adenylate cyclase-activating dopamine receptor signaling pathway / phospholipase C-activating G protein-coupled receptor signaling pathway / G protein-coupled receptor signaling pathway / endoplasmic reticulum membrane ...G protein-coupled amine receptor activity / Amine ligand-binding receptors / trace-amine receptor activity / G alpha (s) signalling events / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / adenylate cyclase-activating dopamine receptor signaling pathway / phospholipase C-activating G protein-coupled receptor signaling pathway / G protein-coupled receptor signaling pathway / endoplasmic reticulum membrane / membrane / plasma membrane Similarity search - Function | |||||||||
| Biological species |  | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 3.04 Å | |||||||||
 Authors Authors | Rong NK / Guo LL / Zhang MH / Li Q / Yang F / Sun JP | |||||||||
| Funding support |  China, 1 items China, 1 items
| |||||||||
 Citation Citation |  Journal: Cell / Year: 2023 Journal: Cell / Year: 2023Title: Structural and signaling mechanisms of TAAR1 enabled preferential agonist design. Authors: Pan Shang / Naikang Rong / Jing-Jing Jiang / Jie Cheng / Ming-Hui Zhang / Dongwei Kang / Lei Qi / Lulu Guo / Gong-Ming Yang / Qun Liu / Zhenzhen Zhou / Xiao-Bing Li / Kong-Kai Zhu / Qing- ...Authors: Pan Shang / Naikang Rong / Jing-Jing Jiang / Jie Cheng / Ming-Hui Zhang / Dongwei Kang / Lei Qi / Lulu Guo / Gong-Ming Yang / Qun Liu / Zhenzhen Zhou / Xiao-Bing Li / Kong-Kai Zhu / Qing-Biao Meng / Xiang Han / Wenqi Yan / Yalei Kong / Lejin Yang / Xiaohui Wang / Dapeng Lei / Xin Feng / Xinyong Liu / Xiao Yu / Yue Wang / Qian Li / Zhen-Hua Shao / Fan Yang / Jin-Peng Sun /  Abstract: Trace amine-associated receptor 1 (TAAR1) senses a spectrum of endogenous amine-containing metabolites (EAMs) to mediate diverse psychological functions and is useful for schizophrenia treatment ...Trace amine-associated receptor 1 (TAAR1) senses a spectrum of endogenous amine-containing metabolites (EAMs) to mediate diverse psychological functions and is useful for schizophrenia treatment without the side effects of catalepsy. Here, we systematically profiled the signaling properties of TAAR1 activation and present nine structures of TAAR1-Gs/Gq in complex with EAMs, clinical drugs, and synthetic compounds. These structures not only revealed the primary amine recognition pocket (PARP) harboring the conserved acidic D for conserved amine recognition and "twin" toggle switch for receptor activation but also elucidated that targeting specific residues in the second binding pocket (SBP) allowed modulation of signaling preference. In addition to traditional drug-induced Gs signaling, Gq activation by EAM or synthetic compounds is beneficial to schizophrenia treatment. Our results provided a structural and signaling framework for molecular recognition by TAAR1, which afforded structural templates and signal clues for TAAR1-targeted candidate compounds design. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_37438.map.gz emd_37438.map.gz | 987.6 KB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-37438-v30.xml emd-37438-v30.xml emd-37438.xml emd-37438.xml | 14.5 KB 14.5 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_37438.png emd_37438.png | 89.1 KB | ||
| Filedesc metadata |  emd-37438.cif.gz emd-37438.cif.gz | 5.5 KB | ||
| Others |  emd_37438_half_map_1.map.gz emd_37438_half_map_1.map.gz emd_37438_half_map_2.map.gz emd_37438_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37438 http://ftp.pdbj.org/pub/emdb/structures/EMD-37438 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37438 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37438 | HTTPS FTP |
-Related structure data
| Related structure data |  8wccMC  8wc3C  8wc4C  8wc5C  8wc6C  8wc7C  8wc8C  8wc9C  8wcaC  8wcbC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_37438.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_37438.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.92 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: #1
| File | emd_37438_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #2
| File | emd_37438_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : Cryo-EM structure of the CHA-bound mTAAR1 complex
| Entire | Name: Cryo-EM structure of the CHA-bound mTAAR1 complex |
|---|---|
| Components |
|
-Supramolecule #1: Cryo-EM structure of the CHA-bound mTAAR1 complex
| Supramolecule | Name: Cryo-EM structure of the CHA-bound mTAAR1 complex / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1 |
|---|---|
| Source (natural) | Organism:  |
-Macromolecule #1: Trace amine-associated receptor 1
| Macromolecule | Name: Trace amine-associated receptor 1 / type: protein_or_peptide / ID: 1 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 37.656586 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MHLCHAITNI SHRNSDWSRE VQASLYSLMS LIILATLVGN LIVIISISHF KQLHTPTNWL LHSMAIVDFL LGCLIMPCSM VRTVERCWY FGEILCKVHT STDIMLSSAS IFHLAFISID RYCAVCDPLR YKAKINISTI LVMILVSWSL PAVYAFGMIF L ELNLKGVE ...String: MHLCHAITNI SHRNSDWSRE VQASLYSLMS LIILATLVGN LIVIISISHF KQLHTPTNWL LHSMAIVDFL LGCLIMPCSM VRTVERCWY FGEILCKVHT STDIMLSSAS IFHLAFISID RYCAVCDPLR YKAKINISTI LVMILVSWSL PAVYAFGMIF L ELNLKGVE ELYRSQVSDL GGCSPFFSKV SGVLAFMTSF YIPGSVMLFV YYRIYFIAKG QARSINRTNV QVGLEGKSQA PQ SKETKAA KTLGIMVGVF LVCWCPFFLC TVLDPFLGYV IPPSLNDALY WFGYLNSALN PMVYAFFYPW FRRALKMVLL GKI FQKDSS RSKLFL UniProtKB: Trace amine-associated receptor 1 |
-Macromolecule #2: CYCLOHEXYLAMMONIUM ION
| Macromolecule | Name: CYCLOHEXYLAMMONIUM ION / type: ligand / ID: 2 / Number of copies: 1 / Formula: HAI |
|---|---|
| Molecular weight | Theoretical: 100.182 Da |
| Chemical component information | 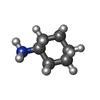 ChemComp-HAI: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.4 |
|---|---|
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: FEI FALCON IV (4k x 4k) / Average electron dose: 1.875 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: DIFFRACTION / Nominal defocus max: 2.0 µm / Nominal defocus min: 1.0 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
- Image processing
Image processing
| Startup model | Type of model: OTHER |
|---|---|
| Final reconstruction | Resolution.type: BY AUTHOR / Resolution: 3.04 Å / Resolution method: FSC 0.143 CUT-OFF / Number images used: 296951 |
| Initial angle assignment | Type: ANGULAR RECONSTITUTION |
| Final angle assignment | Type: ANGULAR RECONSTITUTION |
 Movie
Movie Controller
Controller





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)