-検索条件
-検索結果
検索 (著者・登録者: werner & f)の結果149件中、1から50件目までを表示しています

EMDB-18120: 
The closed state of the ASFV apo-RNA polymerase

EMDB-18129: 
The open state of the ASFV apo-RNA polymerase

EMDB-18163: 
consensus map of the ASFV RNA polymerase in open conformation

EMDB-18164: 
The stalk domain of the ASFV RNA polymerase obtained from Multibody refinement

PDB-8q3b: 
The closed state of the ASFV apo-RNA polymerase

PDB-8q3k: 
The open state of the ASFV apo-RNA polymerase

EMDB-16172: 
Cryo-EM structure of the Arabidopsis thaliana I+III2 supercomplex (Complete conformation 2 composition)

PDB-8bq6: 
Cryo-EM structure of the Arabidopsis thaliana I+III2 supercomplex (Complete conformation 2 composition)

EMDB-16171: 
Cryo-EM structure of the Arabidopsis thaliana I+III2 supercomplex (Complete conformation 1 composition)

PDB-8bq5: 
Cryo-EM structure of the Arabidopsis thaliana I+III2 supercomplex (Complete conformation 1 composition)

EMDB-16156: 
Cryo-EM structure of the Arabidopsis thaliana I+III2 supercomplex (Conformation 1 membrane arm)

EMDB-16168: 
Cryo-EM structure of the Arabidopsis thaliana I+III2 supercomplex (Complete composition)

PDB-8bpx: 
Cryo-EM structure of the Arabidopsis thaliana I+III2 supercomplex (Complete composition)

EMDB-16153: 
Cryo-EM structure of the Arabidopsis thaliana I+III2 supercomplex (Complete consensus)

EMDB-16166: 
Cryo-EM structure of the Arabidopsis thaliana I+III2 supercomplex (Conformation 2 consensus)

EMDB-16167: 
Cryo-EM structure of the Arabidopsis thaliana I+III2 supercomplex (Conformation 1 consensus)

EMDB-15312: 
Complex III2 from Yarrowia lipolytica, combined datasets, consensus refinement

EMDB-15313: 
Complex III2 from Yarrowia lipolytica, atovaquone and antimycin A bound
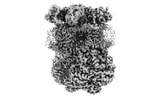
EMDB-15314: 
Complex III2, b-position, with decylubiquinone and ascorbate-reduced

EMDB-15315: 
Complex III2 from Yarrowia lipolytica, ascorbate-reduced, b-position

EMDB-15316: 
Complex III2 from Yarrowia lipolytica, ascorbate-reduced, int-position

EMDB-15317: 
Complex III2 from Yarrowia lipolytica, ascorbate-reduced, c-position
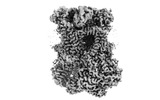
EMDB-15318: 
Complex III2 from Yarrowia lipolytica, oxidised with ferricyanide, b-position
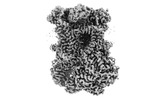
EMDB-15319: 
Complex III2 from Yarrowia lipolytica, oxidised with ferricyanide, int-position
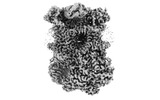
EMDB-15320: 
Complex III2 from Yarrowia lipolytica, oxidised with ferricyanide, c-position
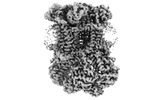
EMDB-15321: 
Complex III2 from Yarrowia lipolytica, antimycin A bound, b-position
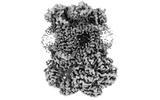
EMDB-15322: 
Complex III2 from Yarrowia lipolytica,antimycin A bound, int-position
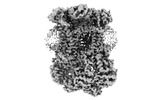
EMDB-15323: 
Complex III2 from Yarrowia lipolytica, antimycin A bound, c-position
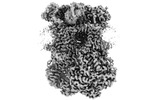
EMDB-15324: 
Complex III2 from Yarrowia lipolytica, decylubiquinol bound, b-position

EMDB-15325: 
Complex III2 from Yarrowia lipolytica, with decylubiquinol and antimycin A, consensus refinement

EMDB-15326: 
Complex III2 from Yarrowia lipolytica, apo, b-position
EMDB-15332: 
Complex III2 from Yarrowia lipolytica, apo, int-position

EMDB-15333: 
Complex III2 from Yarrowia lipolytica, apo, c-position

EMDB-15334: 
Complex III2 from Yarrowia lipolytica, with decylubiquinol, oxidised, b-position

PDB-8ab6: 
Complex III2 from Yarrowia lipolytica, combined datasets, consensus refinement

PDB-8ab7: 
Complex III2 from Yarrowia lipolytica, atovaquone and antimycin A bound

PDB-8ab8: 
Complex III2, b-position, with decylubiquinone and ascorbate-reduced

PDB-8ab9: 
Complex III2 from Yarrowia lipolytica, ascorbate-reduced, b-position

PDB-8aba: 
Complex III2 from Yarrowia lipolytica, ascorbate-reduced, int-position

PDB-8abb: 
Complex III2 from Yarrowia lipolytica, ascorbate-reduced, c-position

PDB-8abe: 
Complex III2 from Yarrowia lipolytica, oxidised with ferricyanide, b-position

PDB-8abf: 
Complex III2 from Yarrowia lipolytica, oxidised with ferricyanide, int-position

PDB-8abg: 
Complex III2 from Yarrowia lipolytica, oxidised with ferricyanide, c-position

PDB-8abh: 
Complex III2 from Yarrowia lipolytica, antimycin A bound, b-position

PDB-8abi: 
Complex III2 from Yarrowia lipolytica,antimycin A bound, int-position

PDB-8abj: 
Complex III2 from Yarrowia lipolytica, antimycin A bound, c-position

PDB-8abk: 
Complex III2 from Yarrowia lipolytica, decylubiquinol bound, b-position

PDB-8abl: 
Complex III2 from Yarrowia lipolytica, with decylubiquinol and antimycin A, consensus refinement

PDB-8abm: 
Complex III2 from Yarrowia lipolytica, apo, b-position

PDB-8ac3: 
Complex III2 from Yarrowia lipolytica, apo, int-position
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
