-検索条件
-検索結果
検索 (著者・登録者: takada & k)の結果92件中、1から50件目までを表示しています

EMDB-19638: 
YlmH bound to PtRNA-50S

EMDB-19641: 
YlmH bound to stalled 50S subunits with RqcH and PtRNA

PDB-8s1p: 
YlmH bound to PtRNA-50S

PDB-8s1u: 
YlmH bound to stalled 50S subunits with RqcH and PtRNA

EMDB-35029: 
SARS-CoV2 spike protein with ACE2, no ACE2 binding.

EMDB-35030: 
SARS-CoV2 spike protein with ACE2. 1 ACE2 bound form.

EMDB-35031: 
SARS-CoV2 spike protein with ACE2. 2 ACE2 bound form.

EMDB-35032: 
SARS-CoV2 spike protein with ACE2. 3 ACE2 bound form.

EMDB-35036: 
SARS-CoV2 spike protein with ACE2 decoy.no ACE2 decoy binding

EMDB-35037: 
SARS-CoV2 spike protein with ACE2 decoy. 1 ACE2 decoy bound form.

EMDB-35038: 
SARS-CoV2 spike protein with ACE2 decoy. 1 ACE2 decoy bound and 2 RBD up form.

EMDB-35039: 
SARS-CoV2 spike protein with ACE2 decoy. 2 ACE2 decoy bound form.

EMDB-35040: 
SARS-CoV2 spike protein with ACE2 decoy. 3 ACE2 decoy bound form.

EMDB-36345: 
RBD of SARS-CoV2 spike protein with ACE2 decoy

PDB-8jje: 
RBD of SARS-CoV2 spike protein with ACE2 decoy
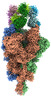
EMDB-33972: 
Cryo-EM structure of the SARS-CoV-2 spike protein (3-up RBD) bound to neutralizing antibody CSW1-1805
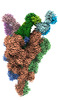
EMDB-33973: 
Cryo-EM structure of the SARS-CoV-2 spike protein (1-up RBD) bound to neutralizing antibody CSW2-1353
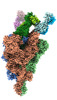
EMDB-33974: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing antibody CSW2-1353
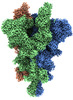
EMDB-33975: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) with C480A mutation

EMDB-27637: 
Structure of EBOV GP lacking the mucin-like domain with 2.1.1D5 scFv and 6D6 scFv bound

EMDB-27638: 
Structure of EBOV GP lacking the mucin-like domain with 9.20.1A2 Fab and 6D6 scFv bound

PDB-8dpl: 
Structure of EBOV GP lacking the mucin-like domain with 2.1.1D5 scFv and 6D6 scFv bound

PDB-8dpm: 
Structure of EBOV GP lacking the mucin-like domain with 9.20.1A2 Fab and 6D6 scFv bound

EMDB-16246: 
ARE-ABCF VmlR2 bound to a 70S ribosome

PDB-8buu: 
ARE-ABCF VmlR2 bound to a 70S ribosome

EMDB-13242: 
PoxtA-EQ2 antibiotic resistance ABCF bound to E. faecalis 70S ribosome, state I

PDB-7p7r: 
PoxtA-EQ2 antibiotic resistance ABCF bound to E. faecalis 70S ribosome, state I

EMDB-13241: 
E. faecalis 70S ribosome bound by PoxtA-EQ2, high-resolution combined volume

EMDB-13243: 
PoxtA-EQ2 antibiotic resistance ABCF bound to E. faecalis 70S ribosome, state II

EMDB-13244: 
PoxtA-EQ2 antibiotic resistance ABCF bound to E. faecalis 70S ribosome, state III

EMDB-13245: 
E. faecalis 70S ribosome with P-tRNA, state IV

PDB-7p7q: 
E. faecalis 70S ribosome bound by PoxtA-EQ2, high-resolution combined volume

PDB-7p7s: 
PoxtA-EQ2 antibiotic resistance ABCF bound to E. faecalis 70S ribosome, state II

PDB-7p7t: 
PoxtA-EQ2 antibiotic resistance ABCF bound to E. faecalis 70S ribosome, state III

PDB-7p7u: 
E. faecalis 70S ribosome with P-tRNA, state IV

EMDB-13017: 
RqcH DR variant bound to 50S-peptidyl-tRNA-RqcP RQC complex (rigid body refinement)

PDB-7ope: 
RqcH DR variant bound to 50S-peptidyl-tRNA-RqcP RQC complex (rigid body refinement)

EMDB-12331: 
LsaA, an antibiotic resistance ABCF, in complex with 70S ribosome from Enterococcus faecalis

EMDB-12332: 
VgaA-LC, an antibiotic resistance ABCF, in complex with 70S ribosome from Staphylococcus aureus

EMDB-12333: 
70S ribosome from Staphylococcus aureus

EMDB-12334: 
VgaL, an antibiotic resistance ABCF, in complex with 70S ribosome from Listeria monocytogenes

PDB-7nhk: 
LsaA, an antibiotic resistance ABCF, in complex with 70S ribosome from Enterococcus faecalis

PDB-7nhl: 
VgaA-LC, an antibiotic resistance ABCF, in complex with 70S ribosome from Staphylococcus aureus

PDB-7nhm: 
70S ribosome from Staphylococcus aureus

PDB-7nhn: 
VgaL, an antibiotic resistance ABCF, in complex with 70S ribosome from Listeria monocytogenes

EMDB-11890: 
Bacillus subtilis ribosome-associated quality control complex state A. Ribosomal 50S subunit with peptidyl tRNA in the A/P position and RqcH.

EMDB-11891: 
Bacillus subtilis ribosome-associated quality control complex state B, multibody refinement focussed on RqcH. Ribosomal 50S subunit with P-tRNA, RqcH, and RqcP/YabO

PDB-7as9: 
Bacillus subtilis ribosome-associated quality control complex state A. Ribosomal 50S subunit with peptidyl tRNA in the A/P position and RqcH.

PDB-7asa: 
Bacillus subtilis ribosome-associated quality control complex state B, multibody refinement focussed on RqcH. Ribosomal 50S subunit with P-tRNA, RqcH, and RqcP/YabO

EMDB-11889: 
Bacillus subtilis ribosome quality control complex state B. Ribosomal 50S subunit with P-tRNA, RqcH, and RqcP/YabO
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
