-検索条件
-検索結果
検索 (著者・登録者: shuai & w)の結果395件中、1から50件目までを表示しています

EMDB-37249: 
Cryo-EM structure of EBV gH/gL-gp42 in complex with fab 2C1

PDB-8khr: 
Cryo-EM structure of EBV gH/gL-gp42 in complex with fab 2C1

EMDB-35827: 
Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate

EMDB-37652: 
Structure of CbCas9 bound to 6-nucleotide complementary DNA substrate

EMDB-37656: 
Structure of CbCas9-PcrIIC1 complex bound to 28-bp DNA substrate (20-nt complementary)

EMDB-37657: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (symmetric 20-nt complementary)

EMDB-37762: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (non-targeting complex)

PDB-8iyq: 
Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate

PDB-8wmh: 
Structure of CbCas9 bound to 6-nucleotide complementary DNA substrate

PDB-8wmm: 
Structure of CbCas9-PcrIIC1 complex bound to 28-bp DNA substrate (20-nt complementary)

PDB-8wmn: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (symmetric 20-nt complementary)

PDB-8wr4: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (non-targeting complex)

EMDB-36776: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron HYER1 at symmetric apo state

EMDB-36777: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron DR1 at symmetric pre-cleavage state

EMDB-36778: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron HYER1 at symmetric post cleavge state

EMDB-36786: 
The Streptococcus azizii ORF-less Group IIC intron HYER2 at apo state

PDB-8k0p: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron HYER1 at symmetric apo state

PDB-8k0q: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron HYER1 at symmetric pre-cleavage state

PDB-8k0r: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron HYER1 at symmetric post cleavge state

PDB-8k15: 
The Streptococcus azizii ORF-less Group IIC intron HYER2 at apo state

EMDB-36779: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron HYER1 with 10-nt TRS at symmetric apo state

PDB-8k0s: 
The Anoxybacillus pushchinoensis ORF-less Group IIC Intron HYER1 with 10-nt TRS at symmetric apo state
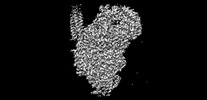
EMDB-37130: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in dimeric form

EMDB-37131: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in monomeric form

PDB-8kdb: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in dimeric form

PDB-8kdc: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in monomeric form

EMDB-36918: 
Cryo-EM structure of Oryza sativa HKT2;1 at 2.5 angstrom

EMDB-36919: 
Cryo-EM structure of Oryza sativa HKT2;2/1 at 2.3 angstrom

PDB-8k66: 
Cryo-EM structure of Oryza sativa HKT2;1 at 2.5 angstrom

PDB-8k69: 
Cryo-EM structure of Oryza sativa HKT2;2/1 at 2.3 angstrom

EMDB-43091: 
hGBP1 conformer on the bacterial outer membrane

EMDB-43153: 
hGBP1 conformer on the bacterial outer membrane

EMDB-35449: 
Eaf3 CHD domain bound to the nucleosome

EMDB-35450: 
Cryo-EM structure of the Rpd3S core complex
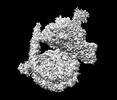
EMDB-35455: 
Rpd3S bound to the nucleosome

PDB-8ihm: 
Eaf3 CHD domain bound to the nucleosome

PDB-8ihn: 
Cryo-EM structure of the Rpd3S core complex

PDB-8iht: 
Rpd3S bound to the nucleosome

EMDB-35456: 
Rpd3S apo state 1

EMDB-35457: 
Rpd3S apo state 2

EMDB-35458: 
Rpd3S apo state 3

EMDB-36368: 
Cryo-EM structure of CCHFV envelope protein Gc trimer in complex with Gc13 Fab

EMDB-36406: 
CCHFV envelope protein Gc in complex with Gc8

EMDB-36407: 
CCHFV envelope protein Gc in complex with Gc13

PDB-8jkd: 
Cryo-EM structure of CCHFV envelope protein Gc trimer in complex with Gc13 Fab

PDB-8jlw: 
CCHFV envelope protein Gc in complex with Gc8

PDB-8jlx: 
CCHFV envelope protein Gc in complex with Gc13

EMDB-35678: 
Apo state of Arabidopsis AZG1 at pH 7.4

EMDB-35679: 
Endogenous substrate adenine bound state of Arabidopsis AZG1 at pH 5.5

EMDB-35680: 
6-BAP bound state of Arabidopsis AZG1
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
