-検索条件
-検索結果
検索 (著者・登録者: shan & yy)の結果全48件を表示しています

EMDB-36800: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state

EMDB-36801: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state, focused refined on KtrA octamer

EMDB-36802: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state, focused refined on KtrB dimer

EMDB-36803: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of MgCl2

EMDB-36804: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA

EMDB-38477: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, vertical C2 symmetry axis

EMDB-38478: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, C1 symmetry

PDB-8k1s: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state

PDB-8k1t: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of MgCl2

PDB-8k1u: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA

PDB-8xmh: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, vertical C2 symmetry axis

PDB-8xmi: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, C1 symmetry

EMDB-34259: 
cryo-EM structure of Omicron BA.5 S protein in complex with XGv282

EMDB-34260: 
Cryo-EM map of Omicron BA.5 S protein in complex with XGv282 focused on RBD_XGv282 sub-complex

EMDB-34261: 
cryo-EM structure of Omicron BA.5 S protein in complex with XGv289

EMDB-34262: 
Cryo-EM map of Omicron BA.5 S protein in complex with XGv289 focused on RBD_XGv289 sub-complex

EMDB-34263: 
cryo-EM structure of Omicron BA.5 S protein in complex with S2L20

EMDB-34264: 
Cryo-EM map of Omicron BA.5 S protein in complex with S2L20 focused on NTD_S2L20 sub-complex

EMDB-33522: 
The pre-fusion structure of Thogotovirus dhori envelope glycoprotein

EMDB-33451: 
Cryo-EM structure of SLC19A1

EMDB-32869: 
S protein of Delta variant in complex with ZWD12

EMDB-32870: 
S protein of Delta variant in complex with ZWD12 focused on RBD_ZWD12 sub-complex

EMDB-32871: 
S protein of Delta variant in complex with ZWC6

EMDB-32872: 
S protein of Delta variant in complex with ZWC6 focused on RBD_ZWC6 sub-complex

EMDB-32920: 
S protein of SARS-CoV-2 in complex with 2G1

EMDB-32921: 
S protein of SARS-CoV-2 in complex with 2G1 focused on RBD_2G1 sub-complex

EMDB-31146: 
Coupling of N7-methyltransferase and 3'-5' exoribonuclease with SARS-CoV-2 polymerase reveals mechanisms for capping and proofreading

EMDB-31138: 
Co-transcriptional capping machineries in SARS-CoV-2 RTC: Coupling of N7-methyltransferase and 3'-5' exoribonuclease with polymerase reveals mechanisms for capping and proofreading

EMDB-11942: 
"Tubulin glycylation controls axonemal dynein activity, flagellar beat and male fertility": Pre-pre-power stroke conformation of the mouse sperm outer dynein arms (ODAs). Both beta- and gamma-heavy chains are in pre-power stroke conformation. Cryo-ET, classification and sub-tomogram averaging.

EMDB-11943: 
"Tubulin glycylation controls axonemal dynein activity, flagellar beat and male fertility": Pre-post-power stroke conformation of the mouse sperm outer dynein arms (ODAs), with gamma-heavy chain in pre-power stroke conformation and beta-heavy chain in post-power stroke conformation. Cryo-ET, classification and sub-tomogram averaging.

EMDB-11944: 
Post-pre-power stroke conformation of the mouse sperm outer dynein arms (ODAs), with gamma-heavy chain in post-power stroke conformation and beta-heavy chain in pre-power stroke conformation. Reconstruction obtained by sub-tomogram classification and averaging from cryo-electron tomograms of active flagella of mouse sperm cells.

EMDB-11945: 
Post-post-power stroke conformation of the mouse sperm outer dynein arms (ODAs), with both gamma-heavy chain and beta-heavy chain in post-power stroke conformation. Reconstruction obtained with sub-tomogram classification and averaging from cryo-electron tomograms of active flagella of mouse sperm cells.

EMDB-11946: 
Axonemal 96nm-repeat from active sperm flagella of wild type mouse. Reconstruction obtained with cryo-ET and sub-tomogram averaging.

EMDB-11947: 
Axonemal 96nm-repeat from active sperm flagella of glycylation deficient mouse (Ttll3-/-Ttll8-/- mutation). Reconstruction obtained with cryo-ET and sub-tomogram averaging.

EMDB-11948: 
Axonemal 96nm-repeat from active flagella of wild type mouse, showing inner dynein arms (IDAs) in pre-power stroke conformations. Reconstruction obtained with classification and averaging of sub-tomograms from cryo-electron tomograms of wild type active flagella.

EMDB-11949: 
Axonemal 96nm-repeat from active flagella of wild type mouse, showing inner dynein arms (IDAs) in post-power stroke conformations. Reconstruction obtained with classification and averaging of sub-tomograms from cryo-electron tomograms of wild type active flagella.

EMDB-22907: 
SARS-CoV-2 Spike bound to Nb6 in closed conformation

EMDB-22908: 
SARS-CoV-2 Spike bound to Nb6 in open conformation

EMDB-22909: 
SARS-CoV-2 Spike bound to Nb11 in closed conformation

EMDB-22910: 
SARS-CoV-2 Spike bound to mNb6 in closed conformation

EMDB-22911: 
SARS-CoV-2 Spike bound to Nb11 in open conformation

PDB-7kkk: 
SARS-CoV-2 Spike in complex with neutralizing nanobody Nb6

PDB-7kkl: 
SARS-CoV-2 Spike in complex with neutralizing nanobody mNb6

EMDB-30276: 
cryo EM map of the S protein of SARS-CoV-2 in complex bound with 4A8

EMDB-30277: 
Cryo EM map of the interface between NTD of SARS-CoV-2 and 4A8
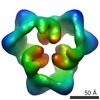
EMDB-1897: 
Reconstruction of the 3D model of AMPK trimer in basal state
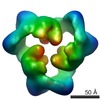
EMDB-1898: 
Reconstruction of the 3D model of AMPK trimer in ATP binding state.
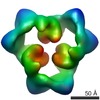
EMDB-1899: 
Reconstruction of the 3D model of AMPK trimer in AMP binding state.
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
