-検索条件
-検索結果
検索 (著者・登録者: li & wq)の結果59件中、1から50件目までを表示しています

EMDB-36484: 
Cryo-EM structure of succinate receptor bound to cis-epoxysuccinic acid coupling to Gi

EMDB-36486: 
Cryo-EM structure of succinate receptor bound to succinate acid coupling MiniGsq

EMDB-36951: 
Cannabinoid Receptor 1 bound to Fenofibrate coupling MiniGsq and Nb35 Complex

EMDB-36952: 
Cannabinoid Receptor 1 bound to Fenofibrate coupling MiniGsq and Nb35 Complex-Global Refine

EMDB-36953: 
Cannabinoid Receptor 1 bound to Fenofibrate coupling MiniGsq and Nb35 Complex-Local Refine

EMDB-34908: 
Cryo-EM structure of ligand histamine-bound Histamine H4 receptor Gi complex

EMDB-34925: 
Cryo-EM structure of ligand histamine-bound Histamine H4 receptor Gi complex

EMDB-27581: 
Cryo-EM structure of the human Sec61 complex in a partially-open apo state (Class 1)

EMDB-27582: 
Cryo-EM structure of the human Sec61 complex in a partially-open apo state (Class 2)

EMDB-27583: 
Cryo-EM structure of the human Sec61 complex inhibited by cotransin

EMDB-27584: 
Cryo-EM structure of the human Sec61 complex inhibited by decatransin

EMDB-27585: 
Cryo-EM structure of the human Sec61 complex inhibited by apratoxin F

EMDB-27586: 
Cryo-EM structure of the human Sec61 complex inhibited by mycolactone

EMDB-27587: 
Cryo-EM structure of the human Sec61 complex inhibited by ipomoeassin F

EMDB-27588: 
Cryo-EM structure of the human Sec61 complex inhibited by cyclotriazadisulfonamide (CADA)

EMDB-27589: 
Cryo-EM structure of the human Sec61 complex inhibited by eeyarestatin I

EMDB-29608: 
Cryo-EM map of chimeric Sec complex (human Sec61 and yeast Sec63-71-72) inhibited by eeyarestatin I

EMDB-29609: 
Cryo-EM map of chimeric Sec complex (human Sec61 and yeast Sec63-71-72) inhibited by cotransin

EMDB-29610: 
Cryo-EM map of chimeric Sec complex (human Sec61 and yeast Sec63-71-72) inhibited by apratoxin F

EMDB-29611: 
Cryo-EM map of chimeric Sec complex (human Sec61 and yeast Sec63-71-72) in a partially-open apo state (Class 1)
EMDB-29612: 
Cryo-EM map of chimeric Sec complex (human Sec61 and yeast Sec63-71-72) in a partially-open apo state (Class 2)
EMDB-29613: 
Cryo-EM map of chimeric Sec complex (human Sec61 and yeast Sec63-71-72) inhibited by decatransin
EMDB-29614: 
Cryo-EM map of chimeric Sec complex (human Sec61 and yeast Sec63-71-72) inhibited by ipomoeassin F (Class 1)
EMDB-29616: 
Cryo-EM map of chimeric Sec complex (human Sec61 and yeast Sec63-71-72) inhibited by cyclotriazadisulfonamide (CADA)
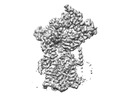
EMDB-29617: 
Cryo-EM map of chimeric Sec complex (human Sec61 and yeast Sec63-71-72) inhibited by mycolactone
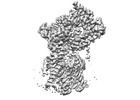
EMDB-29635: 
Cryo-EM map of chimeric Sec complex (human Sec61 and yeast Sec63-71-72) inhibited by ipomoeassin F (Class 2)
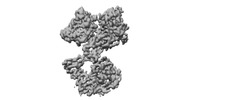
EMDB-32654: 
The local refined map of Omicron spike with bispecific antibody FD01

EMDB-32655: 
The state 1 of Omicron Spike with bispecific antibody FD01

EMDB-32656: 
The state 2 of Omicron Spike with bispecific antibody FD01

EMDB-32657: 
The state 3 of Omicron Spike with bispecific antibody FD01

EMDB-32659: 
The state 4 of Omicron Spike with bispecific antibody FD01

EMDB-32660: 
The state 5 of Omicron Spike with bispecific antibody FD01

EMDB-32661: 
The state 6 of Omicron Spike with bispecific antibody FD01

EMDB-32901: 
SARS-CoV-2 Omicron Spike trimer

EMDB-32638: 
SARS-CoV-2 Spike in complex with IgG 553-15 (S-553-15 dimer trimer )

EMDB-32639: 
SARS-CoV-2 Spike in complex with IgG 553-15 (S-553-15 trimer)

EMDB-32641: 
Locally refined region of SARS-CoV-2 Spike in complex with IgG 553-15

EMDB-32646: 
SARS-CoV-2 Spike in complex with IgG 553-60 (1-up trimer)

EMDB-32647: 
SARS-CoV-2 Spike in complex with IgG 553-60 (2-up trimer)

EMDB-32648: 
Locally refined region of SARS-CoV-2 Spike in complex with IgG 553-60
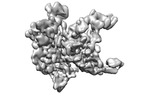
EMDB-32651: 
SARS-CoV-2 Omicron S monomer complexed with 553-49

EMDB-32902: 
SARS-CoV-2 (D614G) Spike trimer

EMDB-32552: 
Omicron spike trimer with 6m6 antibody

EMDB-32553: 
Omicron Spike bitrimer with 6m6 antibody

EMDB-32554: 
Local refine of Omicron spike bitrimer with 6m6 antibody

EMDB-32500: 
The state 2 complex structure of Omicron spike with Bn03 (2-up RBD, 4 nanobodies)

EMDB-32501: 
The state 1 complex structure of Omicron spike with Bn03 (1-up RBD, 3 nanobodies)

EMDB-32502: 
NTD-RBD-Bn03 local refinement

EMDB-32503: 
The state 3 complex structure of Omicron spike with Bn03 (2-up RBD, 5 nanobodies)

EMDB-30861: 
Cryo-electron microscopy density map of the the RBD V367F in complex with MA1ScFv, MA2Fab, and MA5Fab
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
