-検索条件
-検索結果
検索 (著者・登録者: lai & l)の結果1,975件中、1から50件目までを表示しています

EMDB-50321: 
sub-tomogram averaging map of CHIKV replication organelle connection to the plasma membrane
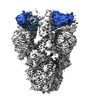
EMDB-39546: 
SARS-CoV-2 Delta Spike in complex with JL-8C

EMDB-39547: 
SARS-CoV-2 Delta Spike in complex with JM-1A

EMDB-39685: 
SARS-CoV-2 Delta Spike in complex with Fab of JE-5C

EMDB-39686: 
SARS-CoV-2 Spike (BA.1) in complex with Fab of JH-8B

PDB-8yro: 
SARS-CoV-2 Delta Spike in complex with JL-8C

PDB-8yrp: 
SARS-CoV-2 Delta Spike in complex with JM-1A

PDB-8yz5: 
SARS-CoV-2 Delta Spike in complex with Fab of JE-5C

PDB-8yz6: 
SARS-CoV-2 Spike (BA.1) in complex with Fab of JH-8B

EMDB-35827: 
Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate

EMDB-37652: 
Structure of CbCas9 bound to 6-nucleotide complementary DNA substrate

EMDB-37656: 
Structure of CbCas9-PcrIIC1 complex bound to 28-bp DNA substrate (20-nt complementary)

EMDB-37657: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (symmetric 20-nt complementary)

EMDB-37762: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (non-targeting complex)

PDB-8iyq: 
Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate

PDB-8wmh: 
Structure of CbCas9 bound to 6-nucleotide complementary DNA substrate

PDB-8wmm: 
Structure of CbCas9-PcrIIC1 complex bound to 28-bp DNA substrate (20-nt complementary)

PDB-8wmn: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (symmetric 20-nt complementary)

PDB-8wr4: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (non-targeting complex)

PDB-9fhp: 
CryoEM structure of wild-type Turnip Yellows Virus

EMDB-40825: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

PDB-8sx3: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

EMDB-35909: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase in complex with bedaquiline(BDQ)

EMDB-35911: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase in the apo-form

PDB-8j0s: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase in complex with bedaquiline(BDQ)

PDB-8j0t: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase in the apo-form

EMDB-50071: 
Cryo-EM structure of the icosahedral lumazine synthase from Vicia faba.

PDB-9ez8: 
Cryo-EM structure of the icosahedral lumazine synthase from Vicia faba.

EMDB-17296: 
Stabilised BA.1 SARS-CoV-2 spike with H6 nanobodies in '2 up 1 down' RBD conformation

PDB-8oyu: 
Stabilised BA.1 SARS-CoV-2 spike with H6 nanobodies in '2 up 1 down' RBD conformation

EMDB-19450: 
Human UPF1 RNA helicase with AMPPNP

EMDB-19451: 
Human UPF1 RNA helicase with AMPPNP and RNA

EMDB-35982: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase Fo in complex with bedaquiline(BDQ)

EMDB-35983: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase Fo in the apo-form

EMDB-36015: 
Mycobacterium tuberculosis ATP synthase Peripheral Stalk in the apo-form

EMDB-36017: 
Mycobacterium tuberculosis ATP synthase Peripheral Stalk in complex with bedaquiline(BDQ)

EMDB-36028: 
Mycobacterium tuberculosis ATP synthase F1 in the apo-form

EMDB-36031: 
Mycobacterium tuberculosis ATP synthase F1 in complex with bedaquiline(BDQ)

EMDB-36589: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase in complex with TBAJ-587

EMDB-36590: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase Fo in complex with TBAJ-587

EMDB-36631: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase F1 in complex with TBAJ-587

EMDB-36632: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase Peripheral Stalk in complex with TBAJ-587

EMDB-37243: 
Structure of the human ATP synthase bound to bedaquiline (membrane domain)

EMDB-37244: 
Structure of the human ATP synthase bound to bedaquiline

EMDB-37245: 
Structure of the human ATP synthase bound to bedaquiline (peripheral stalk domain)

EMDB-37251: 
Structure of the human ATP synthase bound to bedaquiline (composite)
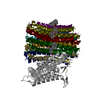
PDB-8j57: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase Fo in complex with bedaquiline(BDQ)

PDB-8j58: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase Fo in the apo-form

PDB-8jr0: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase in complex with TBAJ-587

PDB-8jr1: 
Cryo-EM structure of Mycobacterium tuberculosis ATP synthase Fo in complex with TBAJ-587
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
