-検索条件
-検索結果
検索 (著者・登録者: kim & jm)の結果95件中、1から50件目までを表示しています

EMDB-40711: 
CryoEM structure of Western equine encephalitis virus VLP in complex with the chimeric Du-D1-Mo-D2 MXRA8 receptor

EMDB-27272: 
CryoEM structure of Western equine encephalitis virus VLP
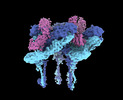
EMDB-27271: 
CryoEM structure of Western equine encephalitis virus VLP in complex with the avian MXRA8 receptor

EMDB-28644: 
CryoEM structure of Western equine encephalitis virus VLP in complex with the avian MXRA8 receptor

EMDB-41144: 
Cryo-EM Structure of GPR61-G protein complex stabilized by scFv16

EMDB-41145: 
Cryo-EM Structure of GPR61-

EMDB-34530: 
Membrane protein A

EMDB-34531: 
Membrane protein B

EMDB-35713: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR1 H225F mutant in lipid nanodisc

EMDB-40181: 
Human TRPV3 tetramer structure, closed conformation

EMDB-40183: 
Human TRPV3 pentamer structure

EMDB-17539: 
Cryo-EM structure of dimeric UBR5

EMDB-17540: 
Cryo-EM structure of full-length human UBR5 (homotetramer)

EMDB-17542: 
Negative stain map of UBR5 (dimer) in complex with RARA/RXRA

EMDB-28523: 
Structure of interleukin receptor common gamma chain (IL2Rgamma) in complex with two antibodies

EMDB-27208: 
anti-HIV-1 gp120-sCD4 complex antibody CG10 Fab in complex with B41-sCD4

EMDB-27209: 
Trimeric HIV-1 Env BG505 SOSIP.664 in complex with sCD4 and antibody 1393A
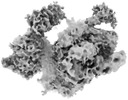
EMDB-27210: 
HIV-1 Env trimer BG505 SOSIP.664 trimer in complex with sCD4 and 1361 Fab

EMDB-27211: 
HIV-1 Env trimer BG505 SOSIP in complex with sCD4 and 697D Fab

EMDB-27212: 
HIV-1 Env trimer BG505 SOSIP in complex with sCD4 and antibody 830A

EMDB-24378: 
5-HT2AR bound to a novel agonist in complex with a mini-Gq protein and an active-state stabilizing single-chain variable fragment (scFv16) obtained by cryo-electron microscopy (cryoEM)

EMDB-32377: 
2.02 angstrom cryo-EM structure of the pump-like channelrhodopsin ChRmine

EMDB-32378: 
2.12 angstrom cryo-EM map of the pump-like channelrhodopsin ChRmine with Fab antibody fragment

EMDB-12465: 
SARS-CoV-2 Spike RBD (dimer) in complex with two Fu2 nanobodies

EMDB-12561: 
SARS-CoV-2 Spike (dimers) in complex with six Fu2 nanobodies

EMDB-23544: 
Human phosphofructokinase-1 liver type bound to activator NA-11

EMDB-13095: 
CryoEM structure of the ABC transporter BmrA E504A mutant in complex with ATP-Mg

EMDB-12170: 
Multidrug resistance transporter BmrA mutant E504A bound with ATP, Mg, and Rhodamine 6G solved by Cryo-EM

EMDB-25448: 
Negative-stain EM reconstruction of SpFN_1B-06-PL, a SARS-CoV-2 spike fused to H.pylori ferritin nanoparticle vaccine candidate
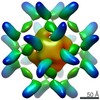
EMDB-25449: 
RFN_131, a Ferritin-based Nanoparticle Vaccine Candidate Displaying the SARS-CoV-2 Receptor-Binding Domain

EMDB-25450: 
pCoV146, a Ferritin-based Nanoparticle Vaccine Candidate Displaying the SARS-CoV-2 Spike Receptor-Binding and N-Terminal Domains

EMDB-25451: 
pCoV111, a Ferritin-based Nanoparticle Vaccine Candidate Displaying the SARS-CoV-2 Spike S1 Subunit

EMDB-24346: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-80 IgG

EMDB-24348: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-081 Fab

EMDB-24349: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-091 IgG

EMDB-24350: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-094 VHH-Fc

EMDB-24351: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-096 IgG

EMDB-24358: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-147 IgG

EMDB-24359: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-246 IgG

EMDB-24335: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-259 IgG

EMDB-24336: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-186 IgG

EMDB-24337: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-199 IgG

EMDB-24338: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-249 IgG

EMDB-24339: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-252 IgG

EMDB-24340: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-049 IgG

EMDB-24341: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-073 scFv

EMDB-24342: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-043 IgG

EMDB-24343: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-010 IgG

EMDB-24344: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-148 IgG

EMDB-24345: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-002 Fab
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
