-検索条件
-検索結果
検索 (著者・登録者: chen & yk)の結果140件中、1から50件目までを表示しています

EMDB-42950: 
Structure of CCP5 class1

EMDB-42951: 
Structure of CCP5 class2

EMDB-42952: 
Structure of CCP5 class3

EMDB-42971: 
CCP5 in complex with microtubules class1

EMDB-42972: 
CCP5 in complex with microtubules class2

EMDB-42973: 
CCP5 in complex with microtubules class3

PDB-8v3q: 
Structure of CCP5 class1

PDB-8v3r: 
Structure of CCP5 class2

PDB-8v3s: 
Structure of CCP5 class3

PDB-8v4k: 
CCP5 in complex with microtubules class1

PDB-8v4l: 
CCP5 in complex with microtubules class2

PDB-8v4m: 
CCP5 in complex with microtubules class3

EMDB-50580: 
SOLIST cryo-tomogram of native left ventricle mouse heart muscle #1

EMDB-50582: 
SOLIST native mouse heart muscle tomogram #2

EMDB-39126: 
Structure of the FADD/Caspase-8/cFLIP death effector domain assembly

EMDB-39127: 
Structure of the FADD/Caspase-8/cFLIP death effector domain assembly

PDB-8ybx: 
Structure of the FADD/Caspase-8/cFLIP death effector domain assembly

EMDB-36800: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state

EMDB-36801: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state, focused refined on KtrA octamer

EMDB-36802: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state, focused refined on KtrB dimer

EMDB-36803: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of MgCl2

EMDB-36804: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA

EMDB-38477: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, vertical C2 symmetry axis

EMDB-38478: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, C1 symmetry

EMDB-36062: 
Asfv topoisomerase 2 - apo conformer Ia

EMDB-36063: 
Asfv topoisomerase 2 - apo conformer Ib

EMDB-36064: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIa

EMDB-36065: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIb

EMDB-36066: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIIa

EMDB-36067: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIIb

EMDB-36116: 
Cryo-EM structure of the African swine fever virus topoisomerase 2 complexed with Cut02aDNA and etoposide (EDI-1)

EMDB-36117: 
Cryo-EM structure of the African swine fever virus topoisomerase 2 complexed with Cut02bDNA and etoposide (EDI-2)

EMDB-36118: 
Cryo-EM structure of the African swine fever virus topoisomerase 2 complexed with Cut02aDNA and m-AMSA (EDI-3)

EMDB-36473: 
Cryo-EM structure of the full-length African swine fever virus topoisomerase 2 complexed with Cut02aDNA and etoposide

EMDB-41810: 
Cryo-EM structure of vaccine-elicited CD4 binding site antibody DH1285 bound to HIV-1 CH505TFchim.6R.SOSIP.664v4.1 Env Local Refinement

EMDB-41820: 
Cryo-EM structure of vaccine-elicited CD4 binding site antibody DH1285 bound to HIV-1 CH505TFchim.6R.SOSIP.664v4.1 Env

EMDB-41823: 
Cryo-EM structure of vaccine-elicited CD4 binding site antibody DH1285 bound to HIV-1 CH505TFchim.6R.SOSIP.664v4.1 Env

EMDB-41838: 
Cryo-EM structure of vaccine-elicited CD4 binding site antibody DH1285 bound to partially open HIV-1 CH505TFchim.6R.SOSIP.664v4.1 Env

EMDB-36137: 
Cryo-EM structure of Holo form of ScBfr with O symmetry

EMDB-36139: 
Cryo-EM structure of Holo form of ScBfr in C1 symmetry

EMDB-36140: 
Cryo-EM structure of Fe-biomineral from bacterioferritin

EMDB-14379: 
Cryo-EM structure of an intact carboxysome - core layer

EMDB-33639: 
Cryo-EM structure of Apo form of ScBfr

EMDB-33640: 
Cryo-EM structure of bacterioferritin holoform 1a

EMDB-33645: 
Cryo-EM structure if bacterioferritin holoform

EMDB-34551: 
Cryo-EM structure of HACE1 monomer

EMDB-34586: 
Cryo-EM structure of HACE1 dimer

EMDB-14376: 
Cryo-EM structure of the Cyanobium sp. PCC 7001 RuBisCO enzyme at 3.8 A resolution with C1 symmetry
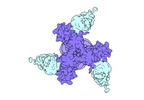
EMDB-27706: 
Vaccine elicited Antibody MU89 bound to CH848.D949.10.17_N133D_N138T.DS.SOSIP.664 HIV-1 Env trimer
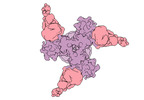
EMDB-27776: 
Vaccine elicited Antibody MU89+S27Y bound to CH848.D949.10.17_N133D_N138T.DS.SOSIP.664 HIV-1 Env trimer
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
