-検索条件
-検索結果
検索 (著者・登録者: chen & cz)の結果265件中、1から50件目までを表示しています

EMDB-42837: 
Cryo-EM structure of GeoCas9 in complex with sgRNA and target DNA

EMDB-42838: 
Cryo-EM structure of iGeoCas9 in complex with sgRNA and target DNA

PDB-8uza: 
Cryo-EM structure of GeoCas9 in complex with sgRNA and target DNA

PDB-8uzb: 
Cryo-EM structure of iGeoCas9 in complex with sgRNA and target DNA

EMDB-37727: 
Cryo-ET structure of RuBisCO from 3.9 angstroms Synechococcus elongatus PCC 7942

EMDB-37728: 
Cryo-ET map of RuBisCO at 4.4 angstroms from Synechococcus elongatus PCC 7942 beta-carboxysome
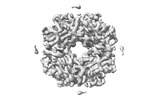
EMDB-37729: 
Cryo-ET map of RuBisCO-SSUL at 5.9 angstroms from Synechococcus elongatus PCC 7942 beta-carboxysome

EMDB-37730: 
Cryo-ET map of RuBisCO at the outermost layer that is loosely attached to the shell of Synechococcus elongatus PCC 7942 beta-carboxysome

EMDB-37731: 
Cryo-ET map of RuBisCO at the outermost layer that is tightly attached to the shell of Synechococcus elongatus PCC 7942 beta-carboxysome

EMDB-39126: 
Structure of the FADD/Caspase-8/cFLIP death effector domain assembly

EMDB-39127: 
Structure of the FADD/Caspase-8/cFLIP death effector domain assembly

PDB-8ybx: 
Structure of the FADD/Caspase-8/cFLIP death effector domain assembly

PDB-8yy8: 
Fzd7 -Gs complex

EMDB-41592: 
S. thermodepolymerans KpsMT(E151Q)-KpsE in complex with ATP

EMDB-41593: 
S. thermodepolymerans KpsMT-KpsE in complex with ADP:AlF4-

EMDB-41595: 
S. thermodepolymerans KpsM-KpsE in Apo 2 state with rigid body fitted KpsT

EMDB-41601: 
S. thermodepolymerans KpsMT-KpsE Apo 1

EMDB-41602: 
S. thermodepolymerans KpsM-KpsE in Glycolipid 2 state with rigid body fitted KpsT

EMDB-41626: 
S. thermodepolymerans KpsM-KpsE in Glycolipid 1 state with rigid body fitted KpsT

EMDB-41677: 
S. thermodepolymerans KpsMT-KpsE Apo 1 - consensus map
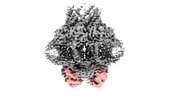
EMDB-41678: 
S. thermodepolymerans KpsMT-KpsE Apo 1 - KpsT-focused map

EMDB-41681: 
S. thermodepolymerans KpsMT(E151Q)-KpsE in complex with ATP - consensus map
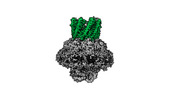
EMDB-41682: 
S. thermodepolymerans KpsMT(E151Q)-KpsE in complex with ATP - crown focused map

EMDB-41697: 
S. thermodepolymerans KpsMT-KpsE in Apo 2 state - consensus map

EMDB-41698: 
S. thermodepolymerans KpsMT-KpsE in Apo 2 state - KpsT focused map

EMDB-41718: 
S. thermodepolymerans KpsMT-KpsE with bound glycolipid - state 1 - consensus map

EMDB-41720: 
S. thermodepolymerans KpsMT-KpsE with bound glycolipid - state 1 - KpsT focused map

EMDB-41721: 
S. thermodepolymerans KpsMT-KpsE with bound glycolipid - state 1 - KpsM focused map

EMDB-41771: 
S. thermodepolymerans KpsMT-KpsE with bound glycolipid - state 2 - consensus map

EMDB-41772: 
S. thermodepolymerans KpsMT-KpsE with bound glycolipid - state 2 - KpsT focused map
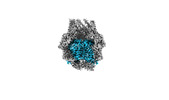
EMDB-41773: 
S. thermodepolymerans KpsMT-KpsE with bound glycolipid - state 2 - KpsM focused map

PDB-8tsh: 
S. thermodepolymerans KpsMT(E151Q)-KpsE in complex with ATP

PDB-8tsi: 
S. thermodepolymerans KpsMT-KpsE in complex with ADP:AlF4-

PDB-8tsl: 
S. thermodepolymerans KpsM-KpsE in Apo 2 state with rigid body fitted KpsT

PDB-8tsw: 
S. thermodepolymerans KpsMT-KpsE Apo 1

PDB-8tt3: 
S. thermodepolymerans KpsM-KpsE in Glycolipid 2 state with rigid body fitted KpsT

PDB-8tun: 
S. thermodepolymerans KpsM-KpsE in Glycolipid 1 state with rigid body fitted KpsT

EMDB-37154: 
Cyanophage A-1(L) neck/gp7-terminator

EMDB-37155: 
Cyanophage A-1(L) neck/gp5-neck fiber

EMDB-37150: 
The CBD domain of cyanophage A-1(L) short tail fiber

EMDB-37151: 
Cyanophage A-1(L) baseplate-initiators

EMDB-37152: 
Cyanophage A-1(L) tail fiber

EMDB-37153: 
Cyanophage A-1(L) sheath-tube

EMDB-41590: 
Cyanophage A-1(L) portal

EMDB-37375: 
Allosteric regulation of nitrate transporter NRT via the signalling protein PII

EMDB-37644: 
Cryo-EM structure of cyanobacterial nitrate/nitrite transporter NrtBCD in complex with signalling protein PII

EMDB-37645: 
Cryo-EM structure of cyanobacterial nitrate/nitrite transporter NrtBCD in complex with nitrate

EMDB-42101: 
Complex of the phosphorylated human cystic fibrosis transmembrane conductance regulator (CFTR) with CFTRinh-172 and ATP/Mg

PDB-8ubr: 
Complex of the phosphorylated human cystic fibrosis transmembrane conductance regulator (CFTR) with CFTRinh-172 and ATP/Mg

EMDB-38543: 
Cryo-EM map of the internal RuBisCOs in the alpha-carboxysome from Prochlorococcus MED4
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
