-検索条件
-検索結果
検索 (著者・登録者: yin & w)の結果3,260件中、1から50件目までを表示しています

EMDB-39126: 
Structure of the FADD/Caspase-8/cFLIP death effector domain assembly

EMDB-39127: 
Structure of the FADD/Caspase-8/cFLIP death effector domain assembly

PDB-8ybx: 
Structure of the FADD/Caspase-8/cFLIP death effector domain assembly

EMDB-38215: 
Human GPR34 -Gi complex bound to S3E-LysoPS

EMDB-38217: 
Human GPR34 -Gi complex bound to S3E-LysoPS, receptor focused

PDB-8xbe: 
Human GPR34 -Gi complex bound to S3E-LysoPS

PDB-8xbg: 
Human GPR34 -Gi complex bound to S3E-LysoPS, receptor focused

EMDB-39724: 
A homotrimeric GPCR architecture of the human cytomegalovirus (UL78) revealed by cryo-EM

PDB-8z1e: 
A homotrimeric GPCR architecture of the human cytomegalovirus (UL78) revealed by cryo-EM

EMDB-41639: 
Langya henipavirus fusion protein in postfusion state

EMDB-41640: 
Langya henipavirus fusion protein in prefusion state

EMDB-41641: 
Langya henipavirus postfusion F protein in complex with the 4G5 Fab, local refinement of the viral membrane distal region

EMDB-41642: 
Langya henipavirus postfusion F protein in complex with 4G5 Fab, local refinement of the viral membrane proximal region

EMDB-41644: 
Langya henipavirus postfusion fusion protein in complex with 4G5 Fab (global refinement)

PDB-8tve: 
Langya henipavirus fusion protein in postfusion state

PDB-8tvf: 
Langya henipavirus fusion protein in prefusion state

PDB-8tvg: 
Langya henipavirus postfusion F protein in complex with the 4G5 Fab, local refinement of the viral membrane distal region

PDB-8tvh: 
Langya henipavirus postfusion F protein in complex with 4G5 Fab, local refinement of the viral membrane proximal region
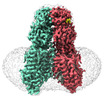
EMDB-36008: 
SIDT1 protein
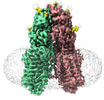
EMDB-36009: 
transport T2

PDB-8j6m: 
SIDT1 protein

PDB-8j6o: 
transport T2

EMDB-37342: 
Structural mechanism of inhibition of the Rho transcription termination factor by Rof

PDB-8w8d: 
Structural mechanism of inhibition of the Rho transcription termination factor by Rof

EMDB-41636: 
Ghanaian virus fusion glycoprotein (GhV F)

EMDB-41643: 
Langya Virus G glycoprotein (LayV G) with stabilizing mutations

EMDB-43593: 
Langya Virus attachment (G) glycoprotein with K85L/L86K mutation

PDB-8tvb: 
Ghanaian virus fusion glycoprotein (GhV F)

PDB-8tvi: 
Langya Virus G glycoprotein (LayV G) with stabilizing mutations

PDB-8vwp: 
Langya Virus attachment (G) glycoprotein with K85L/L86K mutation

EMDB-36849: 
Nipah virus Attachment glycoprotein with 41-6 antibody fragment

PDB-8k3c: 
Nipah virus Attachment glycoprotein with 41-6 antibody fragment
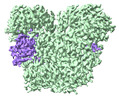
EMDB-36980: 
Cryo-EM structure of DSR2-TTP

EMDB-36982: 
Cryo-EM structure of DSR2-DSAD1 state 2

EMDB-37272: 
Cryo-EM structure of DSR2-DSAD1 state 1
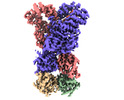
EMDB-37603: 
Cryo-EM structure of DSR2-DSAD1

EMDB-38421: 
Cryo-EM structure of tail tube protein

PDB-8k98: 
Cryo-EM structure of DSR2-TTP

PDB-8k9a: 
Cryo-EM structure of DSR2-DSAD1 state 2

PDB-8w56: 
Cryo-EM structure of DSR2-DSAD1 state 1

PDB-8wkn: 
Cryo-EM structure of DSR2-DSAD1

PDB-8xkn: 
Cryo-EM structure of tail tube protein

EMDB-36594: 
Cryo-EM structure of a designed AAV8-based vector

PDB-8jre: 
Cryo-EM structure of a designed AAV8-based vector
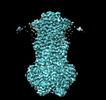
EMDB-40180: 
MsbA bound to cerastecin C

PDB-8gk7: 
MsbA bound to cerastecin C
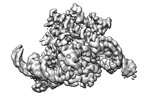
EMDB-35323: 
Cryo-EM structure of the ISFba1 TnpB-reRNA-dsDNA complex

PDB-8iaz: 
Cryo-EM structure of the ISFba1 TnpB-reRNA-dsDNA complex

EMDB-38200: 
Cryo-EM structure of OSCA1.2-liposome-inside-in open state

EMDB-38503: 
Cryo-EM structure of OSCA1.2-liposome-inside-out closed state
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
