-検索条件
-検索結果
検索 (著者・登録者: thomas & ja)の結果1,152件中、1から50件目までを表示しています

EMDB-19250: 
Pseudoatomic model of a second-order Sierpinski triangle formed by the citrate synthase from Synechococcus elongatus

EMDB-19251: 
Structure of a first order Sierpinski triangle formed by the H369R mutant of the citrate synthase from Synechococcus elongatus

PDB-8rjk: 
Pseudoatomic model of a second-order Sierpinski triangle formed by the citrate synthase from Synechococcus elongatus

PDB-8rjl: 
Structure of a first order Sierpinski triangle formed by the H369R mutant of the citrate synthase from Synechococcus elongatus

EMDB-16903: 
60S ribosomal subunit bound to the E3-UFM1 complex (native, UFM1 pulldown)

EMDB-16880: 
60S ribosomal subunit bound to the E3-UFM1 complex - state 3 (native)

EMDB-16902: 
60S ribosomal subunit bound to the E3-UFM1 complex - state 2 (native)
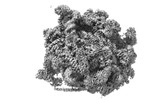
EMDB-16905: 
60S ribosomal subunit bound to the E3-UFM1 complex - state 3 (in-vitro reconstitution)

EMDB-16908: 
60S ribosomal subunit bound to the E3-UFM1 complex - state 1 (native)

EMDB-15525: 
Cryo-EM structure of the RecA postsynaptic filament from S. pneumoniae

PDB-8amf: 
Cryo-EM structure of the RecA postsynaptic filament from S. pneumoniae

EMDB-43222: 
Cryo-EM structure of Tulane virus 9-6-17 variant capsid protein VP1 5-12-18

EMDB-43292: 
Cryo-EM structure of Tulane virus 9-6-17 variant capsid protein VP1 9-14-18, DTT-treated

EMDB-43293: 
Cryo-EM structure of Tulane virus 9-6-17 variant capsid protein VP1 9-14-18 without DTT treatment

PDB-8vgr: 
Cryo-EM structure of Tulane virus 9-6-17 variant capsid protein VP1 5-12-18

PDB-8vjr: 
Cryo-EM structure of Tulane virus 9-6-17 variant capsid protein VP1 9-14-18, DTT-treated

PDB-8vjs: 
Cryo-EM structure of Tulane virus 9-6-17 variant capsid protein VP1 9-14-18 without DTT treatment

EMDB-43542: 
ELIC5 with cysteamine in 2:1:1 POPC:POPE:POPG nanodisc in open conformation

PDB-8vuw: 
ELIC5 with cysteamine in 2:1:1 POPC:POPE:POPG nanodisc in open conformation

EMDB-41374: 
Antibody N3-1 bound to RBDs in the up and down conformations
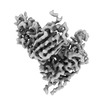
EMDB-41382: 
Antibody N3-1 bound to RBD in the up conformation
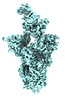
EMDB-41399: 
Antibody N3-1 bound to SARS-CoV-2 spike

PDB-8tm1: 
Antibody N3-1 bound to RBDs in the up and down conformations

PDB-8tma: 
Antibody N3-1 bound to RBD in the up conformation
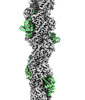
EMDB-16424: 
F-actin decorated by SipA497-669
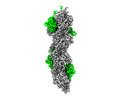
EMDB-16425: 
F-actin decorated by SipA426-685

PDB-8c4c: 
F-actin decorated by SipA497-669

PDB-8c4e: 
F-actin decorated by SipA426-685

EMDB-40856: 
Single particle reconstruction of the human LINE-1 ORF2p without substrate (apo)
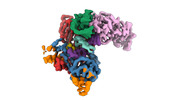
EMDB-40858: 
Structure of LINE-1 ORF2p with template:primer hybrid
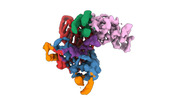
EMDB-40859: 
Structure of LINE-1 ORF2p with an oligo(A) template

PDB-8sxt: 
Structure of LINE-1 ORF2p with template:primer hybrid

PDB-8sxu: 
Structure of LINE-1 ORF2p with an oligo(A) template

EMDB-16805: 
Cryo-EM structure of PcrV/Fab(30-B8)

EMDB-16807: 
Cryo-EM structure of PcrV/Fab(11-E5)

PDB-8cr9: 
Cryo-EM structure of PcrV/Fab(30-B8)

PDB-8crb: 
Cryo-EM structure of PcrV/Fab(11-E5)

EMDB-42124: 
Cryo-EM structure of human STEAP1 in complex with AMG 509 Fab

PDB-8ucd: 
Cryo-EM structure of human STEAP1 in complex with AMG 509 Fab
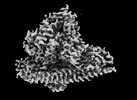
EMDB-41617: 
CryoEM structure of PI3Kalpha

PDB-8tu6: 
CryoEM structure of PI3Kalpha

EMDB-41667: 
Cryo-EM structure of the PP2A:B55-FAM122A complex, B55 body

EMDB-41668: 
Cryo-EM structure of the PP2A:B55-FAM122A complex, PP2Ac body

PDB-8twe: 
Cryo-EM structure of the PP2A:B55-FAM122A complex, B55 body

PDB-8twi: 
Cryo-EM structure of the PP2A:B55-FAM122A complex, PP2Ac body

EMDB-29281: 
Cryo-EM structure of STING oligomer bound to cGAMP and NVS-STG2

EMDB-29282: 
Cryo-EM structure of STING oligomer bound to cGAMP, NVS-STG2 and C53

PDB-8flk: 
Cryo-EM structure of STING oligomer bound to cGAMP and NVS-STG2

PDB-8flm: 
Cryo-EM structure of STING oligomer bound to cGAMP, NVS-STG2 and C53

EMDB-40644: 
Cryo-EM structure of the PP2A:B55-FAM122A complex
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
