-検索条件
-検索結果
検索 (著者・登録者: prazak & v)の結果全44件を表示しています

EMDB-18482: 
Herpes simplex virus 1 capsid (WT) vertices in perinuclear NEC-coated vesicles determined in situ

EMDB-18484: 
Herpes simplex virus 1 nuclear egress complex (WT) determined in situ from perinuclear vesicles

EMDB-17974: 
Pseudorabies virus cytosolic C-capsid (US3 KO) vertices determined in situ

EMDB-17975: 
Pseudorabies virus primary enveloped (perinuclear) C-capsid (US3 KO) vertices determined in situ

EMDB-17976: 
Pseudorabies nuclear C-capsids (US3 KO) vertices determined in situ

EMDB-18473: 
Subtomogram average of pseudorabies virus nuclear egress complex helical form (UL31/34) determined in situ

EMDB-18474: 
Subtomogram average of pseudorabies virus nuclear egress complex (UL31/34) determined in situ

EMDB-18479: 
Pseudorabies virus cytosolic C-capsid (WT) vertices determined in situ

EMDB-18480: 
Pseudorabies virus nuclear C-capsid (WT) vertices determined in situ

EMDB-18481: 
Herpes simplex virus 1 cytosolic C-capsid (WT) vertices determined in situ

EMDB-18483: 
Herpes simplex virus 1 nuclear C-capsid (WT) vertices determined in situ
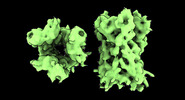
EMDB-17704: 
Subtomogram average of Vaccinia A10 trimer with open center from in vitro cores
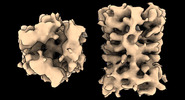
EMDB-17708: 
Subtomogram average of Vaccinia A10 trimer with tight center from in vitro cores

EMDB-17753: 
Subtomogram average of Vaccinia A10 trimer from in situ cores

EMDB-15532: 
Plasmodium falciparum sporozoite subpellicular microtubule with interrupted luminal helix determined in situ

EMDB-15534: 
Plasmodium falciparum gametocyte subpellicular microtubule with 13 protofilaments determined in situ

EMDB-15535: 
Plasmodium falciparum gametocyte subpellicular microtubule with 14 protofilaments determined in situ

EMDB-15536: 
Plasmodium falciparum gametocyte subpellicular microtubule with 15 protofilaments determined in situ

EMDB-15537: 
Plasmodium falciparum gametocyte subpellicular microtubule with 16 protofilaments determined in situ

EMDB-15538: 
Plasmodium falciparum gametocyte subpellicular microtubule with 17 protofilaments determined in situ

EMDB-15539: 
Plasmodium falciparum gametocyte subpellicular microtubule with 18 protofilaments determined in situ

EMDB-15627: 
Tomogram of the Mimivirus genomic fiber

EMDB-15628: 
Tomogram of the Mimivirus genomic fiber

EMDB-15629: 
Tomogram of the Mimivirus genomic fiber

EMDB-15630: 
Tomogram of the Mimivirus genomic fiber

EMDB-13641: 
Structure of the Mimivirus genomic fibre asymmetric unit

EMDB-14353: 
Structure of the Mimivirus genomic fibre in its compact 6-start helix form

EMDB-14354: 
Structure of the Mimivirus genomic fibre in its compact 5-start helix form

EMDB-14355: 
Structure of the Mimivirus genomic fibre in its relaxed 5-start helix form

PDB-7ptv: 
Structure of the Mimivirus genomic fibre asymmetric unit

PDB-7yx3: 
Structure of the Mimivirus genomic fibre in its compact 6-start helix form

PDB-7yx4: 
Structure of the Mimivirus genomic fibre in its compact 5-start helix form

PDB-7yx5: 
Structure of the Mimivirus genomic fibre in its relaxed 5-start helix form

EMDB-12188: 
Structure of DNA origami signpost from cryo-ET and subvolume averaging

PDB-7bho: 
DNA origami signpost designed model

EMDB-11123: 
Pre-fusion conformation of glycoprotein B of Herpes simplex virus 1

PDB-6z9m: 
Pseudoatomic model of the pre-fusion conformation of glycoprotein B of Herpes simplex virus 1

EMDB-10134: 
CryoEM structure of murine perforin-2 ectodomain in a pre-pore form

EMDB-10135: 
CryoEM structure of murine perforin-2 ectodomain in a pore form

EMDB-4995: 
Subtomogram average of a part of the rabies lyssavirus ribonucleoprotein

EMDB-4471: 
Cryo-SOFI enabling low-dose super-resolution correlative light and electron cryo-microscopy

EMDB-4472: 
Cryo-SOFI enabling low-dose super-resolution correlative light and electron cryo-microscopy

EMDB-4473: 
Cryo-SOFI enabling low-dose super-resolution correlative light and electron cryo-microscopy

EMDB-3035: 
The structure of YnaI implies structural and mechanistic conservation in the MscS-family of mechanosensitive channels
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
