PDBの生物学的構造単位について
PDBの生物学的構造単位について
原文:(RCSB PDB-101) Guide to Understanding PDB Data: Introduction to Biological Assemblies and the PDB Archive の内容を基に翻訳(一部編集)しています。
非対称単位
非対称単位(asymmetric unit)は結晶構造の最も小さな部分で、対称操作を行うことにより完全な単位格子(結晶繰り返し単位)を作り出すことができます。 生物学的高分子の結晶で最も一般的な対称操作には回転(rotation)、平行移動(translation)、らせん軸(回転と平行移動の組み合わせ)があります。
非対称単位に対し結晶学的対称操作を行うと1つの単位格子が生成されます。この単位格子を三次元方向に平行移動すると完全な結晶全体の構造が得られます。
以下に簡単な例を示します。2回対称の結晶学的対称軸(黒い卵形)をおおよその中心として、非対称単位(緑の上向き矢印)を180°回転すると2つ目の複製構造(紫色の下向き矢印)ができます。 単位格子はこの2つの矢印で構成されます。次の単位格子を3方向に繰り返し並行移動すると三次元的結晶ができます。

非対称単位には結晶構造中で変換から生成できない最低限必要な位置情報のみが含まれています。 結晶学者は実験データから冗長な部分を取り除いて必要最低限の座標情報のみを構造に登録するのに用います。 必ずしも生物学的に機能する集合体全体の座標情報を記述する必要はないのです。
結晶非対称単位に含まれるものには、以下のパターンがあります。
- 1つの生物学的集合体
- 生物学的集合体の一部
- 複数の生物学的集合体
非対称単位の内容は、結晶化された分子の位置と単位格子内での配置に依存します。結晶化条件と局所パッキングにより、下記2つのパターンがありえます。
- 結晶単位格子に含まれる高分子や複合体の各コピーは、全く同じ配置を持ち互いに対称関係のある位置に配置されている場合、 1つの高分子や複合体で1つの生物学的集合体を構成しているかまたは、対称関係にある2つ以上の高分子や複合体が一緒になってより大きな集合体を形成している状態にあります。
- 個々の高分子や複合体は互いに少しずつ異なる配置を持ち、結晶非対称単位においてそれぞれ固有の位置を占めている場合、似た複数の生物学的集合体が集まって1つの非対称単位を構成する状態にあります。
4つの蛋白質鎖(2つのα-β二量体)を持つヘモグロビンには、この各々に対応するPDBエントリーの良い例があります。
| 非対称単位が1個の生物学的集合体を含む場合 | 非対称単位が生物学的集合体の一部を含む場合 | 非対称単位が複数の生物学的集合体を含む場合 |
|---|---|---|
 |
 |
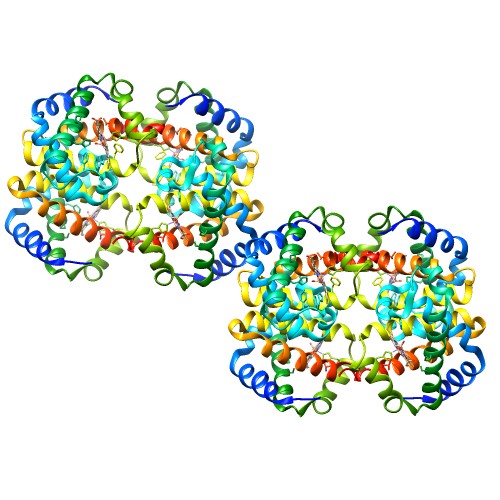 |
| PDBエントリー 2hhb は非対称単位の中に1つのヘモグロビン分子(4つの鎖)を含みます。 | PDBエントリー 1out は非対称単位の中にヘモグロビン分子の半分(2つの鎖)を含みます。結晶学的二回対称軸に沿って単位を複製することによりヘモグロビン分子の残り2鎖を生成することができます。 | PDBエントリー 1hv4 は非対称単位の中に2つのヘモグロビン(8つの鎖)を含みます。 |
生物学的集合体
生物学的集合体(biological assembly)は生物学的単位(biological unit)とも呼ばれ、分子が実際に機能すると示されたか、あるいはそうであると考えられている高分子集合体のことを指します。 例えば、ヘモグロビンが実際機能する状態のものは4つの鎖で構成されています。
操作内容は個々の結晶構造に依存しますが、完全な生物学的集合体の構造を得るには、回転、平行移動、あるいはその組み合わせで構成される対称操作を実行する必要があります。 また、登録された座標の一部だけを選択して生物学的集合体が得られる場合もあります。つまり生物学的集合体を作るには次のような場合があります。
- 非対称単位1つで1つの生物学的集合体となる場合
- 複数の非対称単位で1つの生物学的集合体となる場合
- 非対称単位の一部が生物学的集合体となる場合
上記3つの場合について、ヘモグロビンを例にして示すと次のようになります。
| 生物学的集合体が1個の非対称単位からできている場合 | 生物学的集合体が複数個の非対称単位からできている場合 | 1個の非対称単位に複数の生物学的集合体が含まれる場合 |
|---|---|---|
 |
 |
 |
 |
||
| PDBエントリー 2hhb の場合、生物学的集合体と非対称単位は同一です。 | PDBエントリー 1out の場合、1つの生物学的集合体には2つの非対称単位が含まれています。 | PDBエントリー 1hv4 の場合、生物学的集合体は非対称単位の半分です。 |
| 操作は必要ありません。 | 生物学的集合体を作るには、結晶学的対称操作(結晶学的2回対称軸を中心として180°回転)が必要です。 | このエントリーには構造的に似ているが完全に同一ではない生物学的集合体が2つ含まれています。 |
生物学的集合体は複数の鎖で構成されているとは限りません。
 |
PDBエントリー 7dfr は単量体で、生物学的集合体も1本の鎖しか含まれていません。 |
分子は「クリスタルパッキング」(crystal packing)という現象により、本来複合体を形成しない単位同士が結晶内では複合体を形成しているように見えることがあります。 これは、溶液中でも複合体を形成しているという証拠がないか、あるいは生物学的に関連がないもの同士が複合体を形成していると考えられるものです。 各エントリーをPDBに登録する際、埋もれた部分の表面積と相互作用エネルギーから考え得る集合体全てが計算されます。 これら予想された集合体は、著者が生物学的に関連のある集合体であると考えた集合体と一致する場合もそうでない場合もあります。生物学的集合体に関するコメントには、「著者が提示した」生物学的集合体であるか、「ソフトウェアによって決定された」ものか、またはその両方か、が記載されています。
例えば、PDBエントリー 3fad のT4リゾチーム(T4 lysozyme)の構造には、非対称単位の中に1本の鎖が含まれています。 通常、リゾチームは単量体として機能します。このエントリーでは「著者が提示した」生物学的集合体も「ソフトウェアによって決定された」生物学的集合体も単量体です。 クリスタルパッキングによる、埋もれた表面の表面積と相互作用エネルギーに基づき、ソフトウェア(PISA)は T4リゾチームの特定の変異体/結晶型は二量体を形成するだろうと予測しました。 PDBエントリー 3fad で定義されている集合体は以下の通りです。
| 非対称単位(単量体) | 著者およびソフトウェアによる生物学的集合体(単量体) | ソフトウェアによる生物学的集合体(二量体) |
|---|---|---|
 |
 |
 |
| 非対称単位は単量体です。この座標が登録されています。 | 「著者が提示した」生物学的集合体および「ソフトウェアによって決定された」生物学的集合体はどちらも単量体です。 | PISAというソフトウェアは、この分子が二量体を形成する可能性もあると予測しました。そのため、2つ目の生物学的集合体は「ソフトウェアによって決定された」にすぎません。 |
ウイルスカプシド(viral capsid)の結晶構造には、結晶の非対称単位の一部しか含まれていないことがよくあります。 このようなエントリーの場合、結晶学的非対称単位を生成するには非結晶学的対称操作を登録された座標に対し適用する必要があります。
二十面体のウイルスカプシドには複雑な対称性があり、1つの中心点で交差する5回、3回、2回対称回転によって得られた60個の等価な位置を持っています。 二十面体ウイルスカプシドの登録された座標のほとんどは、二十面体の非対称単位を構成する独特の鎖と非対称単位を生成するための非結晶学的対称操作群で構成されています。 生物学的集合体や結晶学的単位格子を生成するには追加の結晶学的対称操作が必要になるでしょう。 以下に示すのは、PDBエントリー 1qqp に 登録されている、二十面体ウイルスの結晶構造のさまざまな集合体です。
| 二十面体非対称単位 | 結晶非対称単位 | 生物学的集合体 | 結晶学的単位格子 |
|---|---|---|---|
 |
 |
 |
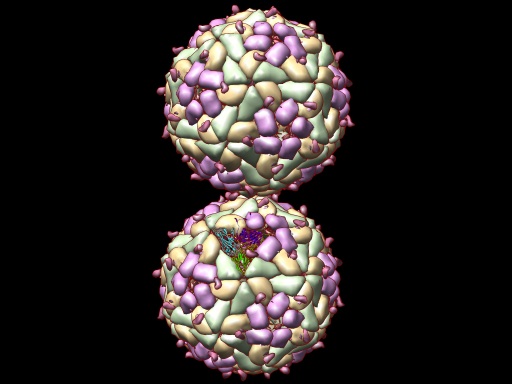 |
| 登録されている座標は1個の正二十面体非対称単位です。各画像において、正二十面体非対称単位がリボンモデルで表示されています。 | 結晶非対称単位は五量体です。 | 生物学的集合体は正二十面体の形状をしています。 | 完全な結晶単位格子には2つの正二十面体ウイルス粒子が含まれています。 |
PDBに登録されているウイルスカプシドは、結晶構造だけではなく、電子顕微鏡、線維回折、固体NMRで決定された構造も含まれています。 正規点(regular point)またはらせん対称による集合体のPDBエントリーは全て、繰り返し単位の座標が含まれていて、生物学的集合体を得るには然るべき結晶学的または非結晶学的対称操作が必要となります。
| 非対称単位 | 生物学的集合体 | |
|---|---|---|
| 例えば、PDBエントリー 1ql2 に登録されている、 糸状バクテリオファージPF1の線維回折構造では、非対称単位には3つのらせんが含まれ、一方生物学的集合体はらせん状のウイルス全体で、 らせん状の回転と移動を示す変換を行うことにより生成されます。 |  |
 |
PDBファイル、mmCIF(PDBML)における生物学的集合体の記述
- PDBフォーマットファイルから生物学的集合体を作る方法
- mmCIF/PDBMLファイルから生物学的集合体を作る方法
- 複数のPDBIDに分けて登録されている場合
PDBフォーマットファイルから生物学的集合体を作る方法
PDBフォーマットファイルでは、生物学的集合体の情報は、REMARK 300とREMARK 350レコードに記載されています。 REMARK 300は、生物学的集合体に関する自由なテキスト形式となっており、登録者による具体的なコメントが記載される場合があります。 一方、REMARK 350には、結晶学的な変換操作だけでなく非結晶学的な操作も含め、生物学的集合体を生成するために必要な全ての変換(回転及び並進)操作が記載されています。 登録者によって提供される変換情報だけでなく、計算によって生物学的集合体(らしき構造)が決定できる場合には、その情報も記載されます。 どちらの場合であるかに応じて、Author-provided(登録者の提示した)生物学的集合体または、software-determined(ソフトウェアによって決定された)生物学的集合体と、示されます。
- 単純な例(3c70)
-
PDBエントリー3c70の場合、REMARK 300の注釈に続いて、REMARK 350には登録座標から生物学的二量体を生成するために必要な変換操作が記載されています。
REMARK 300, 350 レコードREMARK 300 REMARK 300 BIOMOLECULE: 1 REMARK 300 SEE REMARK 350 FOR THE AUTHOR PROVIDED AND/OR PROGRAM REMARK 300 GENERATED ASSEMBLY INFORMATION FOR THE STRUCTURE IN REMARK 300 THIS ENTRY. THE REMARK MAY ALSO PROVIDE INFORMATION ON REMARK 300 BURIED SURFACE AREA. REMARK 350 REMARK 350 COORDINATES FOR A COMPLETE MULTIMER REPRESENTING THE KNOWN REMARK 350 BIOLOGICALLY SIGNIFICANT OLIGOMERIZATION STATE OF THE REMARK 350 MOLECULE CAN BE GENERATED BY APPLYING BIOMT TRANSFORMATIONS REMARK 350 GIVEN BELOW. BOTH NON-CRYSTALLOGRAPHIC AND REMARK 350 CRYSTALLOGRAPHIC OPERATIONS ARE GIVEN. REMARK 350 REMARK 350 BIOMOLECULE: 1 REMARK 350 AUTHOR DETERMINED BIOLOGICAL UNIT: DIMERIC REMARK 350 SOFTWARE DETERMINED QUATERNARY STRUCTURE: DIMERIC REMARK 350 SOFTWARE USED: PISA REMARK 350 TOTAL BURIED SURFACE AREA: 3840 ANGSTROM**2 REMARK 350 SURFACE AREA FOR THE COMPLEX: 19310 ANGSTROM**2 REMARK 350 CHANGE IN SOLVENT FREE ENERGY: -132 KCAL/MOL REMARK 350 APPLY THE FOLLOWING TO CHAINS: A REMARK 350 BIOMT1 1 1.000000 0.000000 0.000000 0.00000 REMARK 350 BIOMT2 1 0.000000 1.000000 0.000000 0.00000 REMARK 350 BIOMT3 1 0.000000 0.000000 1.000000 0.00000 REMARK 350 BIOMT1 2 1.000000 0.000000 0.000000 0.00000 REMARK 350 BIOMT2 2 0.000000 -1.000000 0.000000 106.34400 REMARK 350 BIOMT3 2 0.000000 0.000000 -1.000000 0.00000
この例では、非対称単位は一本の鎖(鎖名A)で構成されています。生物学的二量体は非対称単位のコピー2つから成ります。 1つ目のコピーは、登録構造である非対称単位です(恒等変換は緑色で示されています)。 2つ目のコピーは、回転行列(赤色)と並進ベクトル(青色)から成る結晶学的な対称操作を登録構造に適用することによって、得られます。 ここでの生物学的集合体は登録者の決定とソフトウェア(PISA)による予想が一致していることに注目してください。
- ウイルスカプシドの例(2bfu)
-
この例では、登録座標に含まれる2本鎖(LとS)が正二十面体の非対称単位(完全のウイルスカプシドの1/60)を構成しています。 REMARK 300は自由なテキスト形式の注釈で、REMARK 350には、正二十面体のウイルスを生成するために必要な変換操作が記載されています。 注:5番目から58番目の行列は省略されています。
REMARK 300, 350 レコードREMARK 300 REMARK 300 BIOMOLECULE: 1 REMARK 300 THIS ENTRY CONTAINS THE UNIQUE NON-CRYSTALLOGRAPHIC REPEAT REMARK 300 UNIT, WHICH CONSISTS OF 2 CHAIN(S). SEE REMARK 350 FOR REMARK 300 INFORMATION ON GENERATING THE BIOLOGICAL MOLECULE(S). REMARK 300 THE ASSEMBLY REPRESENTED IN THIS ENTRY HAS REGULAR REMARK 300 ICOSAHEDRAL POINT SYMMETRY (SCHOENFLIES SYMBOL = I). REMARK 350 REMARK 350 GENERATING THE BIOMOLECULE REMARK 350 COORDINATES FOR A COMPLETE MULTIMER REPRESENTING THE KNOWN REMARK 350 BIOLOGICALLY SIGNIFICANT OLIGOMERIZATION STATE OF THE REMARK 350 MOLECULE CAN BE GENERATED BY APPLYING BIOMT TRANSFORMATIONS REMARK 350 GIVEN BELOW. BOTH NON-CRYSTALLOGRAPHIC AND REMARK 350 CRYSTALLOGRAPHIC OPERATIONS ARE GIVEN. REMARK 350 REMARK 350 BIOMOLECULE: 1 REMARK 350 APPLY THE FOLLOWING TO CHAINS: L, S REMARK 350 BIOMT1 1 1.000000 0.000000 0.000000 0.00000 REMARK 350 BIOMT2 1 0.000000 1.000000 0.000000 0.00000 REMARK 350 BIOMT3 1 0.000000 0.000000 1.000000 0.00000 REMARK 350 BIOMT1 2 0.309017 -0.809017 0.500000 0.00000 REMARK 350 BIOMT2 2 0.809017 0.500000 0.309017 0.00000 REMARK 350 BIOMT3 2 -0.500000 0.309017 0.809017 0.00000 REMARK 350 BIOMT1 3 -0.809017 -0.500000 0.309017 0.00000 REMARK 350 BIOMT2 3 0.500000 -0.309017 0.809017 0.00000 REMARK 350 BIOMT3 3 -0.309017 0.809017 0.500000 0.00000 REMARK 350 BIOMT1 4 -0.809017 0.500000 -0.309017 0.00000 REMARK 350 BIOMT2 4 -0.500000 -0.309017 0.809017 0.00000 REMARK 350 BIOMT3 4 0.309017 0.809017 0.500000 0.00000 REMARK 350 BIOMT1 59 -0.309017 -0.809017 -0.500000 0.00000 REMARK 350 BIOMT2 59 -0.809017 0.500000 -0.309017 0.00000 REMARK 350 BIOMT3 59 0.500000 0.309017 -0.809017 0.00000 REMARK 350 BIOMT1 60 -0.500000 -0.309017 -0.809017 0.00000 REMARK 350 BIOMT2 60 0.309017 0.809017 -0.500000 0.00000 REMARK 350 BIOMT3 60 0.809017 -0.500000 -0.309017 0.00000PDBエントリー2bfuの結晶学的非対称単位は、10本の鎖で構成されています(鎖LとS、及び以下の行列を各々の鎖に適用して生成される各4つのコピー)。
MTRIX レコードMTRIX1 1 1.000000 0.000000 0.000000 0.00000 1 MTRIX2 1 0.000000 1.000000 0.000000 0.00000 1 MTRIX3 1 0.000000 0.000000 1.000000 0.00000 1 MTRIX1 2 0.309017 -0.809017 0.500000 0.00000 MTRIX2 2 0.809017 0.500000 0.309017 0.00000 MTRIX3 2 -0.500000 0.309017 0.809017 0.00000 MTRIX1 3 -0.809017 -0.500000 0.309017 0.00000 MTRIX2 3 0.500000 -0.309017 0.809017 0.00000 MTRIX3 3 -0.309017 0.809017 0.500000 0.00000 MTRIX1 4 -0.809017 0.500000 -0.309017 0.00000 MTRIX2 4 -0.500000 -0.309017 0.809017 0.00000 MTRIX3 4 0.309017 0.809017 0.500000 0.00000 MTRIX1 5 0.309017 0.809017 -0.500000 0.00000 MTRIX2 5 -0.809017 0.500000 0.309017 0.00000 MTRIX3 5 0.500000 0.309017 0.809017 0.00000
1つ目の行列は単位行列であり、登録された座標に対応しています。 すでにPDBフォーマットファイルに存在しているため、行列の右側には"1"の印が記載されています。 他の4つの行列を登録座標に適用することによって、5回対称性をもったウイルスの部分集合体が構成されます。
mmCIF/PDBMLファイルから生物学的集合体を作る方法
mmCIFフォーマットファイルでは、「pdbx_struct_assembly」カテゴリと「pdbx_struct_oper_list」カテゴリに各生物学的集合体を作るための具体的情報が記載されています。「pdbx_struct_assembly」には各構造の生物学的集合体の生成に関することが、「pdbx_struct_oper_list」には生物学的集合体の生成に必要な変換規則が記されています。著者が記した特定の生物学的集合体に関する注釈は「struct_biol」カテゴリに記載されています。
- 単純な例(3c70)
-
mmCIFフォーマットにおいて、各カテゴリのデータは下記のように記載されています。
_pdbx_struct_assembly_pdbx_struct_assembly.id 1 _pdbx_struct_assembly.details author_and_software_defined_assembly _pdbx_struct_assembly.method_details PISA
_pdbx_struct_assembly_gen_pdbx_struct_assembly_gen.assembly_id 1 _pdbx_struct_assembly_gen.oper_expression 1,2 _pdbx_struct_assembly_gen.asym_id_list A,B,C,D,E,F,G,H
_pdbx_struct_oper_listloop_ _pdbx_struct_oper_list.id _pdbx_struct_oper_list.type _pdbx_struct_oper_list.name _pdbx_struct_oper_list.matrix[1][1] _pdbx_struct_oper_list.matrix[1][2] _pdbx_struct_oper_list.matrix[1][3] _pdbx_struct_oper_list.vector[1] _pdbx_struct_oper_list.matrix[2][1] _pdbx_struct_oper_list.matrix[2][2] _pdbx_struct_oper_list.matrix[2][3] _pdbx_struct_oper_list.vector[2] _pdbx_struct_oper_list.matrix[3][1] _pdbx_struct_oper_list.matrix[3][2] _pdbx_struct_oper_list.matrix[3][3] _pdbx_struct_oper_list.vector[3] 1 'identity operation' 1_555 1.0000000000 0.0000000000 0.0000000000 0.0000000000 0.0000000000 1.0000000000 0.0000000000 0.0000000000 0.0000000000 0.0000000000 1.0000000000 0.0000000000 2 'crystal symmetry operation' 4_565 1.0000000000 0.0000000000 0.0000000000 0.0000000000 0.0000000000 -1.0000000000 0.0000000000 106.3440000000 0.0000000000 0.0000000000 -1.0000000000 0.0000000000各々に対応するデータは、PDBMLフォーマットでは下記のように記載されています。
pdbx_struct_assemblyCategory<PDBx:pdbx_struct_assemblyCategory> <PDBx:pdbx_struct_assembly id="1"> <PDBx:details>author_and_software_defined_assembly</PDBx:details> <PDBx:method_details>PISA</PDBx:method_details> <PDBx:oligomeric_count>2</PDBx:oligomeric_count> <PDBx:oligomeric_details>dimeric</PDBx:oligomeric_details> </PDBx:pdbx_struct_assembly> </PDBx:pdbx_struct_assemblyCategory>
pdbx_struct_assembly_genCategory<PDBx:pdbx_struct_assembly_genCategory> <PDBx:pdbx_struct_assembly_gen assembly_id="1" asym_id_list="A,B,C,D,E,F,G,H" oper_expression="1,2"/> </PDBx:pdbx_struct_assembly_genCategory>
pdbx_struct_oper_listCategory<PDBx:pdbx_struct_oper_listCategory> <PDBx:pdbx_struct_oper_list id="1"> <-- 1個目は恒等操作(動かさない)--> <PDBx:matrix11>1.0000000000</PDBx:matrix11> <PDBx:matrix12>0.0000000000</PDBx:matrix12> <PDBx:matrix13>0.0000000000</PDBx:matrix13> <PDBx:matrix21>0.0000000000</PDBx:matrix21> <PDBx:matrix22>1.0000000000</PDBx:matrix22> <PDBx:matrix23>0.0000000000</PDBx:matrix23> <PDBx:matrix31>0.0000000000</PDBx:matrix31> <PDBx:matrix32>0.0000000000</PDBx:matrix32> <PDBx:matrix33>1.0000000000</PDBx:matrix33> <PDBx:name>1_555</PDBx:name> <PDBx:symmetry_operation>x,y,z</PDBx:symmetry_operation> <PDBx:type>identity operation</PDBx:type> <PDBx:vector1>0.0000000000</PDBx:vector1> <PDBx:vector2>0.0000000000</PDBx:vector2> <PDBx:vector3>0.0000000000</PDBx:vector3> </PDBx:pdbx_struct_oper_list> <PDBx:pdbx_struct_oper_list id="2"> <-- 2個目はy, zの符合反転後、1単位(106.344)y方向へ平行移動--> <PDBx:matrix11>1.0000000000</PDBx:matrix11> <PDBx:matrix12>0.0000000000</PDBx:matrix12> <PDBx:matrix13>0.0000000000</PDBx:matrix13> <PDBx:matrix21>0.0000000000</PDBx:matrix21> <PDBx:matrix22>-1.0000000000</PDBx:matrix22> <PDBx:matrix23>0.0000000000</PDBx:matrix23> <PDBx:matrix31>0.0000000000</PDBx:matrix31> <PDBx:matrix32>0.0000000000</PDBx:matrix32> <PDBx:matrix33>-1.0000000000</PDBx:matrix33> <PDBx:name>4_565</PDBx:name> <PDBx:symmetry_operation>x,-y+1,-z</PDBx:symmetry_operation> <PDBx:type>crystal symmetry operation</PDBx:type> <PDBx:vector1>0.0000000000</PDBx:vector1> <PDBx:vector2>106.3440000000</PDBx:vector2> <PDBx:vector3>0.0000000000</PDBx:vector3> </PDBx:pdbx_struct_oper_list> </PDBx:pdbx_struct_oper_listCategory>
pdbx_struct_oper_list カテゴリの type にある「1_555」などの値は結晶学での略記法で、アンダースコアの前の数値は対称操作の種類(例:1番→恒等操作、4番→結晶対称操作)を、 後の3桁の数値は必要な平行移動を示しています。 対称操作は空間群によって定義され、並進移動は3つの単位格子の軸(a、b、c)方向の移動量で指定されます。 5 は平行移動しないことを、これより大きい数値は正方向への移動、小さい数値は負方向への移動を示します。 例えば、4_565 は 4番の対称移動が b の正方向に1単位格子分平行移動する内容であることを示しています。
- ウイルスカプシドの例(2bfu)
-
ウイルスや非結晶学的対称性を持つ複合体の場合、生物学的集合体はより複雑で、多くの部分集合体で構成される場合もあります。 pdbx_struct_assembly には考え得る全ての部分集合体が記載され、 その集合体の生成方法は pdbx_struct_assembly_gen に記されます。 pdbx_struct_oper_list カテゴリでは登録されている座標データから生物学的集合体を生成するのに必要な変換行列(結晶学的な操作もそうでない操作も共に)が列挙されています。ここには次の行列も含まれます。「P」は登録された座標から標準点枠?(standard point frame)への変換を、「X0」は登録された座標から結晶枠?(crystal frame2)へ移動させるのに必要な変換を含みます。つまり、登録された座標はこれらの変換により標準点や結晶枠へ移動させることができるのです。
以下に示すmmCIF/PDBMLの内容は全て、5番から58番の変換行列が省略されています。 また、非結晶学的対称(non-crystallographic symmetry、NCS)の対称操作は「struct_ncs_oper」カテゴリに記載してあります。
mmCIFフォーマットにおいて、各カテゴリのデータは下記のように記載されています。
_pdbx_struct_assemblyloop_ _pdbx_struct_assembly.id _pdbx_struct_assembly.details 1 'complete icosahedral assembly' 2 'icosahedral asymmetric unit' 3 'icosahedral pentamer' 4 'icosahedral 23 hexamer' PAU 'icosahedral asymmetric unit, std point frame' XAU 'crystal asymmetric unit, crystal frame'_pdbx_struct_assembly_genloop_ _pdbx_struct_assembly_gen.assembly_id _pdbx_struct_assembly_gen.oper_expression _pdbx_struct_assembly_gen.asym_id_list _pdbx_struct_assembly_gen.entity_inst_id 1 (1-60) A,B . 2 1 A,B . 3 (1-5) A,B . 4 (1,2,6,10,23,24) A,B . PAU P A,B . XAU (X0)(1-5) A,B ._pdbx_struct_oper_listloop_ _pdbx_struct_oper_list.id _pdbx_struct_oper_list.type _pdbx_struct_oper_list.matrix[1][1] _pdbx_struct_oper_list.matrix[1][2] _pdbx_struct_oper_list.matrix[1][3] _pdbx_struct_oper_list.vector[1] _pdbx_struct_oper_list.matrix[2][1] _pdbx_struct_oper_list.matrix[2][2] _pdbx_struct_oper_list.matrix[2][3] _pdbx_struct_oper_list.vector[2] _pdbx_struct_oper_list.matrix[3][1] _pdbx_struct_oper_list.matrix[3][2] _pdbx_struct_oper_list.matrix[3][3] _pdbx_struct_oper_list.vector[3] P 'transform to point frame' 0.30901699 -0.80901699 0.50000000 0.00000 0.80901699 0.50000000 0.30901699 -0.00000 -0.50000000 0.30901699 0.80901699 0.00000 X0 'transform to crystal frame' 1.00000000 0.00000000 0.00000000 0.00000 0.00000000 1.00000000 0.00000000 0.00000 0.00000000 0.00000000 1.00000000 0.00000 1 'point symmetry operation' 1.00000000 0.00000000 0.00000000 0.00000 0.00000000 1.00000000 0.00000000 0.00000 0.00000000 0.00000000 1.00000000 0.00000 2 'point symmetry operation' 0.30901699 -0.80901699 0.50000000 0.00000 0.80901699 0.50000000 0.30901699 0.00000 -0.50000000 0.30901699 0.80901699 0.00000 3 'point symmetry operation' -0.80901699 -0.50000000 0.30901699 0.00000 0.50000000 -0.30901699 0.80901699 0.00000 -0.30901699 0.80901699 0.50000000 0.00000 4 'point symmetry operation' -0.80901699 0.50000000 -0.30901699 0.00000 -0.50000000 -0.30901699 0.80901699 0.00000 0.30901699 0.80901699 0.50000000 0.00000 (中略) 59 'point symmetry operation' -0.30901699 -0.80901699 -0.50000000 0.00000 -0.80901699 0.50000000 -0.30901699 0.00000 0.50000000 0.30901699 -0.80901699 0.00000 60 'point symmetry operation' -0.50000000 -0.30901699 -0.80901699 0.00000 0.30901699 0.80901699 -0.50000000 0.00000 0.80901699 -0.50000000 -0.30901699 0.00000
_pdbx_point_symmetry_pdbx_point_symmetry.entry_id 2BFU _pdbx_point_symmetry.Schoenflies_symbol I
_struct_ncs_operloop_ _struct_ncs_oper.id _struct_ncs_oper.code _struct_ncs_oper.details _struct_ncs_oper.matrix[1][1] _struct_ncs_oper.matrix[1][2] _struct_ncs_oper.matrix[1][3] _struct_ncs_oper.matrix[2][1] _struct_ncs_oper.matrix[2][2] _struct_ncs_oper.matrix[2][3] _struct_ncs_oper.matrix[3][1] _struct_ncs_oper.matrix[3][2] _struct_ncs_oper.matrix[3][3] _struct_ncs_oper.vector[1] _struct_ncs_oper.vector[2] _struct_ncs_oper.vector[3] 1 given ? 1.00000000 0.00000000 0.00000000 0.00000000 1.00000000 0.00000000 0.00000000 0.00000000 1.00000000 0.00000 0.00000 0.00000 2 generate ? 0.30901699 -0.80901699 0.50000000 0.80901699 0.50000000 0.30901699 -0.50000000 0.30901699 0.80901699 0.00000 0.00000 0.00000 3 generate ? -0.80901699 -0.50000000 0.30901699 0.50000000 -0.30901699 0.80901699 -0.30901699 0.80901699 0.50000000 0.00000 0.00000 0.00000 4 generate ? -0.80901699 0.50000000 -0.30901699 -0.50000000 -0.30901699 0.80901699 0.30901699 0.80901699 0.50000000 0.00000 0.00000 0.00000 5 generate ? 0.30901699 0.80901699 -0.50000000 -0.80901699 0.50000000 0.30901699 0.50000000 0.30901699 0.80901699 0.00000 0.00000 0.00000PDBMLフォーマットでは、各々のカテゴリに対応するデータは下記のように記載されています。
pdbx_struct_assemblyCategory<PDBx:pdbx_struct_assemblyCategory> <PDBx:pdbx_struct_assembly id="1"> <PDBx:details>complete icosahedral assembly</PDBx:details> <PDBx:oligomeric_count>120</PDBx:oligomeric_count> <PDBx:oligomeric_details>120-MERIC</PDBx:oligomeric_details> </PDBx:pdbx_struct_assembly> <PDBx:pdbx_struct_assembly id="2"> <PDBx:details>icosahedral asymmetric unit</PDBx:details> </PDBx:pdbx_struct_assembly> <PDBx:pdbx_struct_assembly id="3"> <PDBx:details>icosahedral pentamer</PDBx:details> </PDBx:pdbx_struct_assembly> <PDBx:pdbx_struct_assembly id="4"> <PDBx:details>icosahedral 23 hexamer</PDBx:details> </PDBx:pdbx_struct_assembly> <PDBx:pdbx_struct_assembly id="PAU"> <PDBx:details>icosahedral asymmetric unit, std point frame</PDBx:details> </PDBx:pdbx_struct_assembly> <PDBx:pdbx_struct_assembly id="XAU"> <PDBx:details>crystal asymmetric unit, crystal frame</PDBx:details> </PDBx:pdbx_struct_assembly> </PDBx:pdbx_struct_assemblyCategory>pdbx_struct_assembly_genCategory<PDBx:pdbx_struct_assembly_genCategory> <PDBx:pdbx_struct_assembly_gen assembly_id="1" asym_id_list="A,B" oper_expression="(1-60)"></PDBx:pdbx_struct_assembly_gen> <PDBx:pdbx_struct_assembly_gen assembly_id="2" asym_id_list="A,B" oper_expression="1"></PDBx:pdbx_struct_assembly_gen> <PDBx:pdbx_struct_assembly_gen assembly_id="3" asym_id_list="A,B" oper_expression="(1-5)"></PDBx:pdbx_struct_assembly_gen> <PDBx:pdbx_struct_assembly_gen assembly_id="4" asym_id_list="A,B" oper_expression="(1,2,6,10,23,24)"></PDBx:pdbx_struct_assembly_gen> <PDBx:pdbx_struct_assembly_gen assembly_id="PAU" asym_id_list="A,B" oper_expression="P"></PDBx:pdbx_struct_assembly_gen> <PDBx:pdbx_struct_assembly_gen assembly_id="XAU" asym_id_list="A,B" oper_expression="(X0)(1-5)"></PDBx:pdbx_struct_assembly_gen> </PDBx:pdbx_struct_assembly_genCategory>pdbx_struct_oper_listCategory<PDBx:pdbx_struct_oper_listCategory> <PDBx:pdbx_struct_oper_list id="P"> <PDBx:matrix11>0.30901699</PDBx:matrix11> <PDBx:matrix12>-0.80901699</PDBx:matrix12> <PDBx:matrix13>0.50000000</PDBx:matrix13> <PDBx:matrix21>0.80901699</PDBx:matrix21> <PDBx:matrix22>0.50000000</PDBx:matrix22> <PDBx:matrix23>0.30901699</PDBx:matrix23> <PDBx:matrix31>-0.50000000</PDBx:matrix31> <PDBx:matrix32>0.30901699</PDBx:matrix32> <PDBx:matrix33>0.80901699</PDBx:matrix33> <PDBx:type>transform to point frame</PDBx:type> <PDBx:vector1>0.00000</PDBx:vector1> <PDBx:vector2>-0.00000</PDBx:vector2> <PDBx:vector3>0.00000</PDBx:vector3> </PDBx:pdbx_struct_oper_list> <PDBx:pdbx_struct_oper_list id="X0"> <PDBx:matrix11>1.00000000</PDBx:matrix11> <PDBx:matrix12>0.00000000</PDBx:matrix12> <PDBx:matrix13>0.00000000</PDBx:matrix13> <PDBx:matrix21>0.00000000</PDBx:matrix21> <PDBx:matrix22>1.00000000</PDBx:matrix22> <PDBx:matrix23>0.00000000</PDBx:matrix23> <PDBx:matrix31>0.00000000</PDBx:matrix31> <PDBx:matrix32>0.00000000</PDBx:matrix32> <PDBx:matrix33>1.00000000</PDBx:matrix33> <PDBx:type>transform to crystal frame</PDBx:type> <PDBx:vector1>0.00000</PDBx:vector1> <PDBx:vector2>0.00000</PDBx:vector2> <PDBx:vector3>0.00000</PDBx:vector3> </PDBx:pdbx_struct_oper_list> <PDBx:pdbx_struct_oper_list id="1"> <PDBx:matrix11>1.00000000</PDBx:matrix11> <PDBx:matrix12>0.00000000</PDBx:matrix12> <PDBx:matrix13>0.00000000</PDBx:matrix13> <PDBx:matrix21>0.00000000</PDBx:matrix21> <PDBx:matrix22>1.00000000</PDBx:matrix22> <PDBx:matrix23>0.00000000</PDBx:matrix23> <PDBx:matrix31>0.00000000</PDBx:matrix31> <PDBx:matrix32>0.00000000</PDBx:matrix32> <PDBx:matrix33>1.00000000</PDBx:matrix33> <PDBx:type>point symmetry operation</PDBx:type> <PDBx:vector1>0.00000</PDBx:vector1> <PDBx:vector2>0.00000</PDBx:vector2> <PDBx:vector3>0.00000</PDBx:vector3> </PDBx:pdbx_struct_oper_list> <PDBx:pdbx_struct_oper_list id="2"> <PDBx:matrix11>0.30901699</PDBx:matrix11> <PDBx:matrix12>-0.80901699</PDBx:matrix12> <PDBx:matrix13>0.50000000</PDBx:matrix13> <PDBx:matrix21>0.80901699</PDBx:matrix21> <PDBx:matrix22>0.50000000</PDBx:matrix22> <PDBx:matrix23>0.30901699</PDBx:matrix23> <PDBx:matrix31>-0.50000000</PDBx:matrix31> <PDBx:matrix32>0.30901699</PDBx:matrix32> <PDBx:matrix33>0.80901699</PDBx:matrix33> <PDBx:type>point symmetry operation</PDBx:type> <PDBx:vector1>0.00000</PDBx:vector1> <PDBx:vector2>0.00000</PDBx:vector2> <PDBx:vector3>0.00000</PDBx:vector3> </PDBx:pdbx_struct_oper_list> <PDBx:pdbx_struct_oper_list id="3"> <PDBx:matrix11>-0.80901699</PDBx:matrix11> <PDBx:matrix12>-0.50000000</PDBx:matrix12> <PDBx:matrix13>0.30901699</PDBx:matrix13> <PDBx:matrix21>0.50000000</PDBx:matrix21> <PDBx:matrix22>-0.30901699</PDBx:matrix22> <PDBx:matrix23>0.80901699</PDBx:matrix23> <PDBx:matrix31>-0.30901699</PDBx:matrix31> <PDBx:matrix32>0.80901699</PDBx:matrix32> <PDBx:matrix33>0.50000000</PDBx:matrix33> <PDBx:type>point symmetry operation</PDBx:type> <PDBx:vector1>0.00000</PDBx:vector1> <PDBx:vector2>0.00000</PDBx:vector2> <PDBx:vector3>0.00000</PDBx:vector3> </PDBx:pdbx_struct_oper_list> <PDBx:pdbx_struct_oper_list id="4"> <PDBx:matrix11>-0.80901699</PDBx:matrix11> <PDBx:matrix12>0.50000000</PDBx:matrix12> <PDBx:matrix13>-0.30901699</PDBx:matrix13> <PDBx:matrix21>-0.50000000</PDBx:matrix21> <PDBx:matrix22>-0.30901699</PDBx:matrix22> <PDBx:matrix23>0.80901699</PDBx:matrix23> <PDBx:matrix31>0.30901699</PDBx:matrix31> <PDBx:matrix32>0.80901699</PDBx:matrix32> <PDBx:matrix33>0.50000000</PDBx:matrix33> <PDBx:type>point symmetry operation</PDBx:type> <PDBx:vector1>0.00000</PDBx:vector1> <PDBx:vector2>0.00000</PDBx:vector2> <PDBx:vector3>0.00000</PDBx:vector3> </PDBx:pdbx_struct_oper_list> (中略) <PDBx:pdbx_struct_oper_list id="59"> <PDBx:matrix11>-0.30901699</PDBx:matrix11> <PDBx:matrix12>-0.80901699</PDBx:matrix12> <PDBx:matrix13>-0.50000000</PDBx:matrix13> <PDBx:matrix21>-0.80901699</PDBx:matrix21> <PDBx:matrix22>0.50000000</PDBx:matrix22> <PDBx:matrix23>-0.30901699</PDBx:matrix23> <PDBx:matrix31>0.50000000</PDBx:matrix31> <PDBx:matrix32>0.30901699</PDBx:matrix32> <PDBx:matrix33>-0.80901699</PDBx:matrix33> <PDBx:type>point symmetry operation</PDBx:type> <PDBx:vector1>0.00000</PDBx:vector1> <PDBx:vector2>0.00000</PDBx:vector2> <PDBx:vector3>0.00000</PDBx:vector3> </PDBx:pdbx_struct_oper_list> <PDBx:pdbx_struct_oper_list id="60"> <PDBx:matrix11>-0.50000000</PDBx:matrix11> <PDBx:matrix12>-0.30901699</PDBx:matrix12> <PDBx:matrix13>-0.80901699</PDBx:matrix13> <PDBx:matrix21>0.30901699</PDBx:matrix21> <PDBx:matrix22>0.80901699</PDBx:matrix22> <PDBx:matrix23>-0.50000000</PDBx:matrix23> <PDBx:matrix31>0.80901699</PDBx:matrix31> <PDBx:matrix32>-0.50000000</PDBx:matrix32> <PDBx:matrix33>-0.30901699</PDBx:matrix33> <PDBx:type>point symmetry operation</PDBx:type> <PDBx:vector1>0.00000</PDBx:vector1> <PDBx:vector2>0.00000</PDBx:vector2> <PDBx:vector3>0.00000</PDBx:vector3> </PDBx:pdbx_struct_oper_list> </PDBx:pdbx_struct_oper_listCategory>
pdbx_point_symmetryCategory<PDBx:pdbx_point_symmetryCategory> <PDBx:pdbx_point_symmetry entry_id="2BFU"> <PDBx:Schoenflies_symbol>I</PDBx:Schoenflies_symbol> </PDBx:pdbx_point_symmetry> </PDBx:pdbx_point_symmetryCategory>
struct_ncs_operCategory<PDBx:struct_ncs_operCategory> <PDBx:struct_ncs_oper id="1"> <PDBx:code>given</PDBx:code> <PDBx:matrix11>output">1.000000</PDBx:matrix11> <PDBx:matrix12>output">0.000000</PDBx:matrix12> <PDBx:matrix13>output">0.000000</PDBx:matrix13> <PDBx:matrix21>output">0.000000</PDBx:matrix21> <PDBx:matrix22>output">1.000000</PDBx:matrix22> <PDBx:matrix23>output">0.000000</PDBx:matrix23> <PDBx:matrix31>output">0.000000</PDBx:matrix31> <PDBx:matrix32>output">0.000000</PDBx:matrix32> <PDBx:matrix33>output">1.000000</PDBx:matrix33> <PDBx:vector1>output">0.00000</PDBx:vector1> <PDBx:vector2>output">0.00000</PDBx:vector2> <PDBx:vector3>output">0.00000</PDBx:vector3> </PDBx:struct_ncs_oper> <PDBx:struct_ncs_oper id="2"> <PDBx:code>generate</PDBx:code> <PDBx:matrix11>output">0.309017</PDBx:matrix11> <PDBx:matrix12>-0.809017</PDBx:matrix12> <PDBx:matrix13>output">0.500000</PDBx:matrix13> <PDBx:matrix21>output">0.809017</PDBx:matrix21> <PDBx:matrix22>output">0.500000</PDBx:matrix22> <PDBx:matrix23>output">0.309017</PDBx:matrix23> <PDBx:matrix31>-0.500000</PDBx:matrix31> <PDBx:matrix32>output">0.309017</PDBx:matrix32> <PDBx:matrix33>output">0.809017</PDBx:matrix33> <PDBx:vector1>output">0.00000</PDBx:vector1> <PDBx:vector2>output">0.00000</PDBx:vector2> <PDBx:vector3>output">0.00000</PDBx:vector3> </PDBx:struct_ncs_oper> <PDBx:struct_ncs_oper id="3"> <PDBx:code>generate</PDBx:code> <PDBx:matrix11>-0.809017</PDBx:matrix11> <PDBx:matrix12>-0.500000</PDBx:matrix12> <PDBx:matrix13>output">0.309017</PDBx:matrix13> <PDBx:matrix21>output">0.500000</PDBx:matrix21> <PDBx:matrix22>-0.309017</PDBx:matrix22> <PDBx:matrix23>output">0.809017</PDBx:matrix23> <PDBx:matrix31>-0.309017</PDBx:matrix31> <PDBx:matrix32>output">0.809017</PDBx:matrix32> <PDBx:matrix33>output">0.500000</PDBx:matrix33> <PDBx:vector1>output">0.00000</PDBx:vector1> <PDBx:vector2>output">0.00000</PDBx:vector2> <PDBx:vector3>output">0.00000</PDBx:vector3> </PDBx:struct_ncs_oper> <PDBx:struct_ncs_oper id="4"> <PDBx:code>generate</PDBx:code> <PDBx:matrix11>-0.809017</PDBx:matrix11> <PDBx:matrix12>output">0.500000</PDBx:matrix12> <PDBx:matrix13>-0.309017</PDBx:matrix13> <PDBx:matrix21>-0.500000</PDBx:matrix21> <PDBx:matrix22>-0.309017</PDBx:matrix22> <PDBx:matrix23>output">0.809017</PDBx:matrix23> <PDBx:matrix31>output">0.309017</PDBx:matrix31> <PDBx:matrix32>output">0.809017</PDBx:matrix32> <PDBx:matrix33>output">0.500000</PDBx:matrix33> <PDBx:vector1>output">0.00000</PDBx:vector1> <PDBx:vector2>output">0.00000</PDBx:vector2> <PDBx:vector3>output">0.00000</PDBx:vector3> </PDBx:struct_ncs_oper> <PDBx:struct_ncs_oper id="5"> <PDBx:code>generate</PDBx:code> <PDBx:matrix11>output">0.309017</PDBx:matrix11> <PDBx:matrix12>output">0.809017</PDBx:matrix12> <PDBx:matrix13>-0.500000</PDBx:matrix13> <PDBx:matrix21>-0.809017</PDBx:matrix21> <PDBx:matrix22>output">0.500000</PDBx:matrix22> <PDBx:matrix23>output">0.309017</PDBx:matrix23> <PDBx:matrix31>output">0.500000</PDBx:matrix31> <PDBx:matrix32>output">0.309017</PDBx:matrix32> <PDBx:matrix33>output">0.809017</PDBx:matrix33> <PDBx:vector1>output">0.00000</PDBx:vector1> <PDBx:vector2>output">0.00000</PDBx:vector2> <PDBx:vector3>output">0.00000</PDBx:vector3> </PDBx:struct_ncs_oper> </PDBx:struct_ncs_operCategory> - 複数のPDBIDに分けて登録されている場合
-
注)PDBフォーマットに収まらないという理由で1つの構造を複数のエントリーに分割する措置は2014年12月10日に廃止されました。詳細はPDBjニュースをご覧下さい。
リボソームのように巨大な構造の場合、PDBフォーマットファイルで多くの残基と鎖を記述できるようにするため2つ以上のエントリーに分割されていることがあります。 つまり、非対称単位や生物学的集合体の座標が複数のPDBエントリーに渡って保存されていることがあるということです。 このような場合、各PDBフォーマットファイルの「REMARK 300」「REMARK 350」に記された内容は本来の生物学的集合体全体を示している訳ではありません。
非対称単位を見るに当たって結合する必要のあるPDB IDはPDBフォーマットファイルの「SPLIT」の項目に記載されています。 以下の例は PDBエントリー 2j00の「SPLIT」項目です。
SPLIT 2J00 2J01 2J02 2J03
PDBjの構造概要ページでは、分割された個々のエントリの非対称単位が表示されています。分割された複数のエントリーを合成することで、完全な非対称単位が得られます。
生物学的集合体座標ファイルの表示とダウンロード
wwPDBが作成した生物学的集合体(生物学的単位)の座標は wwPDBのftpサイトや、その完全なミラーであるPDBjのPDBアーカイブに保存されています。
wwPDBのftpサイト:ftp://ftp.wwpdb.org/pub/pdb/data/biounit
PDBjアーカイブ:ftp://ftp.pdbj.org/pub/pdb/data/biounit
(PDBフォーマットで提供されているのは、通常のエントリーのみ。現在、PDBx/mmCIFフォーマットで提供されているのは、巨大構造のみ)
著者
Shuchismita Dutta, Rachel Kramer Green, and Catherine L. Lawson
参考文献
- E. Krissinel and K. Henrick (2007) Inference of macromolecular assemblies from crystalline state. J. Mol. Biol. 372: 774-797. [doi:10.1016/j.str.2008.12.014]
- C.L. Lawson, S. Dutta, J.D. Westbrook, K. Henrick, H.M. Berman (2008) Representation of viruses in the remediated PDB archive. Acta Cryst. D64: 874-882
- K. Henrick, J.M. Thornton (1998) PQS: a protein quaternary structure file server. Trends in Biochemical Sciences 23(9): 358-361. http://pqs.ebi.ac.uk







