+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7b49 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structural basis of reactivation of oncogenic p53 mutants by a small molecule: methylene quinuclidinone (MQ). Human p53DBD-R273H mutant bound to DNA and MQ: R273H-DNA-MQ | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  TRANSCRIPTION (転写 (生物学)) / TRANSCRIPTION (転写 (生物学)) /  P53 (P53遺伝子) / P53 (P53遺伝子) /  TUMOR SUPPRESSOR (がん抑制遺伝子) / TUMOR SUPPRESSOR (がん抑制遺伝子) /  DNA BINDING PROTEIN (DNA結合タンパク質) / PROTEIN DNA COMPLEX / DNA BINDING PROTEIN (DNA結合タンパク質) / PROTEIN DNA COMPLEX /  MICHAEL ACCEPTOR / MICHAEL ACCEPTOR /  MICHAEL REACTION / PROTEIN-DRUG COMPLEX / PROTEIN-DNA-DRUG COMPLEX / LOOP-SHEET-HELIX MOTIF / DNA TARGET / ACTIVATOR / HOOGSTEEN BASE-PAIRING MICHAEL REACTION / PROTEIN-DRUG COMPLEX / PROTEIN-DNA-DRUG COMPLEX / LOOP-SHEET-HELIX MOTIF / DNA TARGET / ACTIVATOR / HOOGSTEEN BASE-PAIRING | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Loss of function of TP53 in cancer due to loss of tetramerization ability / Regulation of TP53 Expression / signal transduction by p53 class mediator / negative regulation of G1 to G0 transition / negative regulation of glucose catabolic process to lactate via pyruvate / Transcriptional activation of cell cycle inhibitor p21 / regulation of intrinsic apoptotic signaling pathway by p53 class mediator / Activation of NOXA and translocation to mitochondria / negative regulation of pentose-phosphate shunt / ATP-dependent DNA/DNA annealing activity ...Loss of function of TP53 in cancer due to loss of tetramerization ability / Regulation of TP53 Expression / signal transduction by p53 class mediator / negative regulation of G1 to G0 transition / negative regulation of glucose catabolic process to lactate via pyruvate / Transcriptional activation of cell cycle inhibitor p21 / regulation of intrinsic apoptotic signaling pathway by p53 class mediator / Activation of NOXA and translocation to mitochondria / negative regulation of pentose-phosphate shunt / ATP-dependent DNA/DNA annealing activity / negative regulation of helicase activity / regulation of cell cycle G2/M phase transition / intrinsic apoptotic signaling pathway in response to hypoxia / regulation of fibroblast apoptotic process / oxidative stress-induced premature senescence / oligodendrocyte apoptotic process / negative regulation of miRNA processing / positive regulation of thymocyte apoptotic process / glucose catabolic process to lactate via pyruvate /  regulation of tissue remodeling / positive regulation of mitochondrial membrane permeability / negative regulation of mitophagy / positive regulation of programmed necrotic cell death / regulation of tissue remodeling / positive regulation of mitochondrial membrane permeability / negative regulation of mitophagy / positive regulation of programmed necrotic cell death /  転写 (生物学) / bone marrow development / circadian behavior / T cell proliferation involved in immune response / regulation of mitochondrial membrane permeability involved in apoptotic process / histone deacetylase regulator activity / RUNX3 regulates CDKN1A transcription / germ cell nucleus / regulation of DNA damage response, signal transduction by p53 class mediator / TP53 regulates transcription of additional cell cycle genes whose exact role in the p53 pathway remain uncertain / TP53 Regulates Transcription of Death Receptors and Ligands / Activation of PUMA and translocation to mitochondria / DNA damage response, signal transduction by p53 class mediator resulting in transcription of p21 class mediator / negative regulation of neuroblast proliferation / negative regulation of glial cell proliferation / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / Regulation of TP53 Activity through Association with Co-factors / positive regulation of execution phase of apoptosis / mitochondrial DNA repair / T cell lineage commitment / negative regulation of DNA replication / ER overload response / B cell lineage commitment / thymocyte apoptotic process / positive regulation of cardiac muscle cell apoptotic process / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / TP53 Regulates Transcription of Caspase Activators and Caspases / entrainment of circadian clock by photoperiod / cardiac septum morphogenesis / PI5P Regulates TP53 Acetylation / Association of TriC/CCT with target proteins during biosynthesis / Zygotic genome activation (ZGA) / necroptotic process / rRNA transcription / positive regulation of release of cytochrome c from mitochondria / TP53 Regulates Transcription of Genes Involved in Cytochrome C Release / TFIID-class transcription factor complex binding / 転写 (生物学) / bone marrow development / circadian behavior / T cell proliferation involved in immune response / regulation of mitochondrial membrane permeability involved in apoptotic process / histone deacetylase regulator activity / RUNX3 regulates CDKN1A transcription / germ cell nucleus / regulation of DNA damage response, signal transduction by p53 class mediator / TP53 regulates transcription of additional cell cycle genes whose exact role in the p53 pathway remain uncertain / TP53 Regulates Transcription of Death Receptors and Ligands / Activation of PUMA and translocation to mitochondria / DNA damage response, signal transduction by p53 class mediator resulting in transcription of p21 class mediator / negative regulation of neuroblast proliferation / negative regulation of glial cell proliferation / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / Regulation of TP53 Activity through Association with Co-factors / positive regulation of execution phase of apoptosis / mitochondrial DNA repair / T cell lineage commitment / negative regulation of DNA replication / ER overload response / B cell lineage commitment / thymocyte apoptotic process / positive regulation of cardiac muscle cell apoptotic process / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / TP53 Regulates Transcription of Caspase Activators and Caspases / entrainment of circadian clock by photoperiod / cardiac septum morphogenesis / PI5P Regulates TP53 Acetylation / Association of TriC/CCT with target proteins during biosynthesis / Zygotic genome activation (ZGA) / necroptotic process / rRNA transcription / positive regulation of release of cytochrome c from mitochondria / TP53 Regulates Transcription of Genes Involved in Cytochrome C Release / TFIID-class transcription factor complex binding /  マイトファジー / negative regulation of telomere maintenance via telomerase / SUMOylation of transcription factors / intrinsic apoptotic signaling pathway by p53 class mediator / neuroblast proliferation / general transcription initiation factor binding / cellular response to actinomycin D / Transcriptional Regulation by VENTX / DNA damage response, signal transduction by p53 class mediator / response to X-ray / マイトファジー / negative regulation of telomere maintenance via telomerase / SUMOylation of transcription factors / intrinsic apoptotic signaling pathway by p53 class mediator / neuroblast proliferation / general transcription initiation factor binding / cellular response to actinomycin D / Transcriptional Regulation by VENTX / DNA damage response, signal transduction by p53 class mediator / response to X-ray /  ヘイフリック限界 / chromosome organization / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / ヘイフリック限界 / chromosome organization / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress /  gastrulation / cellular response to UV-C / response to inorganic substance / hematopoietic stem cell differentiation / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / positive regulation of RNA polymerase II transcription preinitiation complex assembly / negative regulation of reactive oxygen species metabolic process / MDM2/MDM4 family protein binding / embryonic organ development / glial cell proliferation / gastrulation / cellular response to UV-C / response to inorganic substance / hematopoietic stem cell differentiation / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / positive regulation of RNA polymerase II transcription preinitiation complex assembly / negative regulation of reactive oxygen species metabolic process / MDM2/MDM4 family protein binding / embryonic organ development / glial cell proliferation /  Pyroptosis / cis-regulatory region sequence-specific DNA binding / hematopoietic progenitor cell differentiation / cellular response to glucose starvation / TP53 Regulates Transcription of Genes Involved in G1 Cell Cycle Arrest / Pyroptosis / cis-regulatory region sequence-specific DNA binding / hematopoietic progenitor cell differentiation / cellular response to glucose starvation / TP53 Regulates Transcription of Genes Involved in G1 Cell Cycle Arrest /  somitogenesis / type II interferon-mediated signaling pathway / DNA damage response, signal transduction by p53 class mediator resulting in cell cycle arrest / positive regulation of intrinsic apoptotic signaling pathway / negative regulation of stem cell proliferation / core promoter sequence-specific DNA binding / negative regulation of fibroblast proliferation / cardiac muscle cell apoptotic process / response to salt stress / transcription initiation-coupled chromatin remodeling / mitotic G1 DNA damage checkpoint signaling / Regulation of TP53 Activity through Acetylation somitogenesis / type II interferon-mediated signaling pathway / DNA damage response, signal transduction by p53 class mediator resulting in cell cycle arrest / positive regulation of intrinsic apoptotic signaling pathway / negative regulation of stem cell proliferation / core promoter sequence-specific DNA binding / negative regulation of fibroblast proliferation / cardiac muscle cell apoptotic process / response to salt stress / transcription initiation-coupled chromatin remodeling / mitotic G1 DNA damage checkpoint signaling / Regulation of TP53 Activity through Acetylation類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)synthetic construct (人工物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.42 Å 分子置換 / 解像度: 1.42 Å | ||||||
 データ登録者 データ登録者 | Rozenberg, H. / Degtjarik, O. / Shakked, Z. | ||||||
| 資金援助 | 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structural basis of reactivation of oncogenic p53 mutants by a small molecule: methylene quinuclidinone (MQ). 著者: Degtjarik, O. / Golovenko, D. / Diskin-Posner, Y. / Abrahmsen, L. / Rozenberg, H. / Shakked, Z. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7b49.cif.gz 7b49.cif.gz | 249.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7b49.ent.gz pdb7b49.ent.gz | 162.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7b49.json.gz 7b49.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b4/7b49 https://data.pdbj.org/pub/pdb/validation_reports/b4/7b49 ftp://data.pdbj.org/pub/pdb/validation_reports/b4/7b49 ftp://data.pdbj.org/pub/pdb/validation_reports/b4/7b49 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  6zncC  7b46C  7b47C  7b48C  7b4aC  7b4bC  7b4cC  7b4dC  7b4eC  7b4fC  7b4gC 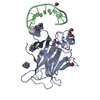 7b4hC  7b4nC  4ibsS C: 同じ文献を引用 ( S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 単位格子 |
| ||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
-タンパク質 / DNA鎖 , 2種, 3分子 ABC
| #1: タンパク質 |  P53遺伝子 / Antigen NY-CO-13 / Phosphoprotein p53 / Tumor suppressor p53 P53遺伝子 / Antigen NY-CO-13 / Phosphoprotein p53 / Tumor suppressor p53分子量: 22486.535 Da / 分子数: 2 / 断片: p53 human DNA binding domain / 変異: R273H / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: TP53, P53 / プラスミド: pET-27-b / 発現宿主: Homo sapiens (ヒト) / 遺伝子: TP53, P53 / プラスミド: pET-27-b / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: P04637 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: P04637#2: DNA鎖 | | 分子量: 6441.135 Da / 分子数: 1 / 断片: p53 DNA target / 変異: ' / 由来タイプ: 合成 / 詳細: IDT: Integrated DNA Technology / 由来: (合成) synthetic construct (人工物) |
|---|
-非ポリマー , 7種, 534分子 
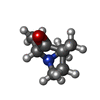
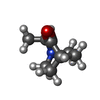










| #3: 化合物 | | #4: 化合物 | #5: 化合物 | #6: 化合物 | ChemComp-FMT /  ギ酸 ギ酸#7: 化合物 |  酢酸塩 酢酸塩#8: 化合物 | ChemComp-EDO /  エチレングリコール エチレングリコール#9: 水 | ChemComp-HOH / |  水 水 |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| 非ポリマーの詳細 | Two enantiomers, QNN and QN8 are produced by addition reaction of the michael acceptor compound "2- ...Two enantiomers, QNN and QN8 are produced by addition reaction of the michael acceptor compound "2-methylenequinuclidin-3-one" with cysteine or lysine. As such, QNN and QN8 bind covalently to the thiol group of cysteine or amino group of lysine. The chiral definitions of QNN and QN8 bound to cysteines are reversed to that of the pseudo free ligands. |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.27 Å3/Da / 溶媒含有率: 45.81 % |
|---|---|
結晶化 | 温度: 292 K / 手法: 蒸発脱水法 / pH: 6.1 詳細: Protein/DNA ratio 1:1.4, 0.075M Sodium formate, 7.5% w/v PEG 3350 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID23-1 / 波長: 0.972 Å / ビームライン: ID23-1 / 波長: 0.972 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2018年12月8日 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長 : 0.972 Å / 相対比: 1 : 0.972 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 1.38→39 Å / Num. obs: 93002 / % possible obs: 98 % / 冗長度: 5.5 % / Rmerge(I) obs: 0.119 / Rpim(I) all: 0.055 / Rrim(I) all: 0.132 / Χ2: 0.997 / Net I/σ(I): 3.6 / Num. measured all: 510562 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
-位相決定
位相決定 | 手法:  分子置換 分子置換 |
|---|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 4IBS 解像度: 1.42→34.11 Å / SU ML: 0.1895 / 交差検証法: FREE R-VALUE / σ(F): 1.35 / 位相誤差: 34.7122 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 21.79 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.42→34.11 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj
























































