| 登録情報 | データベース: PDB / ID: 6a6j
|
|---|
| タイトル | Crystal structure of Zebra fish Y-box protein1 (YB-1) Cold-shock domain in complex with 6mer m5C RNA |
|---|
 要素 要素 | - RNA (5'-R(P*CP*AP*UP*(5MC)P*U)-3')
- Zebra fish Y-box protein1 (YB-1)
|
|---|
 キーワード キーワード | RNA BINDING PROTEIN/RNA / OB-fold / RNA BINDING PROTEIN-RNA complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |    Danio rerio (ゼブラフィッシュ) Danio rerio (ゼブラフィッシュ)
  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2.255 Å シンクロトロン / 解像度: 2.255 Å |
|---|
 データ登録者 データ登録者 | Zhang, M.M. / Wu, B.X. / Huang, Y. / Ma, J.B. |
|---|
 引用 引用 |  ジャーナル: Mol.Cell / 年: 2019 ジャーナル: Mol.Cell / 年: 2019
タイトル: RNA 5-Methylcytosine Facilitates the Maternal-to-Zygotic Transition by Preventing Maternal mRNA Decay.
著者: Yang, Y. / Wang, L. / Han, X. / Yang, W.L. / Zhang, M. / Ma, H.L. / Sun, B.F. / Li, A. / Xia, J. / Chen, J. / Heng, J. / Wu, B. / Chen, Y.S. / Xu, J.W. / Yang, X. / Yao, H. / Sun, J. / Lyu, C. ...著者: Yang, Y. / Wang, L. / Han, X. / Yang, W.L. / Zhang, M. / Ma, H.L. / Sun, B.F. / Li, A. / Xia, J. / Chen, J. / Heng, J. / Wu, B. / Chen, Y.S. / Xu, J.W. / Yang, X. / Yao, H. / Sun, J. / Lyu, C. / Wang, H.L. / Huang, Y. / Sun, Y.P. / Zhao, Y.L. / Meng, A. / Ma, J. / Liu, F. / Yang, Y.G. |
|---|
| 履歴 | | 登録 | 2018年6月28日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2019年6月19日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年6月26日 | Group: Data collection / Database references ...Data collection / Database references / Source and taxonomy / Structure summary
カテゴリ: entity / entity_name_com ...entity / entity_name_com / entity_src_gen / struct_ref / struct_ref_seq
Item: _entity.pdbx_description / _entity.pdbx_fragment ..._entity.pdbx_description / _entity.pdbx_fragment / _entity_name_com.name / _entity_src_gen.gene_src_common_name / _entity_src_gen.pdbx_gene_src_gene / _entity_src_gen.pdbx_gene_src_ncbi_taxonomy_id / _entity_src_gen.pdbx_gene_src_scientific_name / _struct_ref.db_code / _struct_ref.pdbx_align_begin / _struct_ref.pdbx_db_accession / _struct_ref_seq.db_align_beg / _struct_ref_seq.db_align_end / _struct_ref_seq.pdbx_db_accession |
|---|
| 改定 1.2 | 2019年8月14日 | Group: Data collection / Database references / Structure summary
カテゴリ: audit_author / citation / citation_author
Item: _audit_author.name / _citation.country ..._audit_author.name / _citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.pdbx_database_id_DOI / _citation.title / _citation.year |
|---|
| 改定 1.3 | 2019年8月21日 | Group: Data collection / Database references / カテゴリ: citation / citation_author
Item: _citation.pdbx_database_id_PubMed / _citation.title / _citation_author.name |
|---|
| 改定 1.4 | 2019年10月2日 | Group: Data collection / Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.5 | 2024年3月27日 | Group: Data collection / Database references / Derived calculations
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_struct_conn_angle / struct_conn / struct_conn_type
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_ptnr1_label_alt_id / _struct_conn.pdbx_ptnr2_label_alt_id / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_conn_type.id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 messenger ribonucleoprotein complex / CRD-mediated mRNA stabilization / protein localization to cytoplasmic stress granule / eukaryotic initiation factor 4E binding / histone pre-mRNA 3'end processing complex / embryonic morphogenesis / U12-type spliceosomal complex / positive regulation of cytoplasmic translation / mRNA stabilization / cellular response to interleukin-7 / miRNA binding / mRNA Splicing - Minor Pathway / negative regulation of mRNA splicing, via spliceosome / negative regulation of cellular senescence / Processing of Capped Intron-Containing Pre-mRNA / positive regulation of cell division / epidermis development / negative regulation of translational initiation / mRNA Splicing - Major Pathway /
messenger ribonucleoprotein complex / CRD-mediated mRNA stabilization / protein localization to cytoplasmic stress granule / eukaryotic initiation factor 4E binding / histone pre-mRNA 3'end processing complex / embryonic morphogenesis / U12-type spliceosomal complex / positive regulation of cytoplasmic translation / mRNA stabilization / cellular response to interleukin-7 / miRNA binding / mRNA Splicing - Minor Pathway / negative regulation of mRNA splicing, via spliceosome / negative regulation of cellular senescence / Processing of Capped Intron-Containing Pre-mRNA / positive regulation of cell division / epidermis development / negative regulation of translational initiation / mRNA Splicing - Major Pathway /  RNA splicing /
RNA splicing /  P-body / SMAD2/SMAD3:SMAD4 heterotrimer regulates transcription /
P-body / SMAD2/SMAD3:SMAD4 heterotrimer regulates transcription /  転写後修飾 / cytoplasmic stress granule / Interferon gamma signaling / sequence-specific double-stranded DNA binding /
転写後修飾 / cytoplasmic stress granule / Interferon gamma signaling / sequence-specific double-stranded DNA binding /  single-stranded DNA binding /
single-stranded DNA binding /  GTPase binding /
GTPase binding /  double-stranded DNA binding /
double-stranded DNA binding /  遺伝子発現の調節 / in utero embryonic development / negative regulation of translation /
遺伝子発現の調節 / in utero embryonic development / negative regulation of translation /  nucleic acid binding /
nucleic acid binding /  ribonucleoprotein complex /
ribonucleoprotein complex /  mRNA binding / intracellular membrane-bounded organelle /
mRNA binding / intracellular membrane-bounded organelle /  シナプス /
シナプス /  chromatin binding / regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II /
chromatin binding / regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II /  小胞体 / positive regulation of transcription by RNA polymerase II /
小胞体 / positive regulation of transcription by RNA polymerase II /  DNA binding /
DNA binding /  RNA binding / extracellular exosome / extracellular region /
RNA binding / extracellular exosome / extracellular region /  核質 /
核質 /  細胞核 /
細胞核 /  細胞膜 /
細胞膜 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Danio rerio (ゼブラフィッシュ)
Danio rerio (ゼブラフィッシュ)
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン / 解像度: 2.255 Å
シンクロトロン / 解像度: 2.255 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Mol.Cell / 年: 2019
ジャーナル: Mol.Cell / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6a6j.cif.gz
6a6j.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6a6j.ent.gz
pdb6a6j.ent.gz PDB形式
PDB形式 6a6j.json.gz
6a6j.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/a6/6a6j
https://data.pdbj.org/pub/pdb/validation_reports/a6/6a6j ftp://data.pdbj.org/pub/pdb/validation_reports/a6/6a6j
ftp://data.pdbj.org/pub/pdb/validation_reports/a6/6a6j リンク
リンク 集合体
集合体
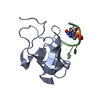
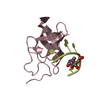
 要素
要素
 Danio rerio (ゼブラフィッシュ)
Danio rerio (ゼブラフィッシュ)
 Escherichia coli (大腸菌) / 参照: UniProt: B5DE31, UniProt: P67809*PLUS
Escherichia coli (大腸菌) / 参照: UniProt: B5DE31, UniProt: P67809*PLUS
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL17U1 / 波長: 0.97914 Å
/ ビームライン: BL17U1 / 波長: 0.97914 Å : 0.97914 Å / 相対比: 1
: 0.97914 Å / 相対比: 1  解析
解析 ムービー
ムービー コントローラー
コントローラー







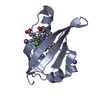





 PDBj
PDBj







