| 登録情報 | データベース: PDB / ID: 5zlp
|
|---|
| タイトル | Crystal structure of glutamine synthetase from helicobacter pylori |
|---|
 要素 要素 | Glutamine synthetase グルタミンシンテターゼ グルタミンシンテターゼ |
|---|
 キーワード キーワード |  LIGASE (リガーゼ) / LIGASE (リガーゼ) /  glutamine synthetase (グルタミンシンテターゼ) / glutamine synthetase (グルタミンシンテターゼ) /  enzyme (酵素) / GS / enzyme (酵素) / GS /  helicobacter pylori (ヘリコバクター・ピロリ) / helicobacter pylori (ヘリコバクター・ピロリ) /  STRUCTURAL PROTEIN (タンパク質) STRUCTURAL PROTEIN (タンパク質) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
nitrogen utilization /  グルタミンシンテターゼ / glutamine biosynthetic process / グルタミンシンテターゼ / glutamine biosynthetic process /  glutamine synthetase activity / glutamine synthetase activity /  ATP binding / ATP binding /  生体膜 / 生体膜 /  metal ion binding / metal ion binding /  細胞質類似検索 - 分子機能 細胞質類似検索 - 分子機能  Glutamine synthetase, N-terminal domain / Glutamine synthetase type I / Glutamine synthetase/guanido kinase, catalytic domain / Glutamine synthetase (GS) beta-grasp domain profile. / Glutamine synthetase (GS) catalytic domain profile. / Glutamine synthetase, N-terminal domain / Glutamine synthetase type I / Glutamine synthetase/guanido kinase, catalytic domain / Glutamine synthetase (GS) beta-grasp domain profile. / Glutamine synthetase (GS) catalytic domain profile. /  Glutamine synthetase, beta-Grasp domain / Glutamine synthetase, beta-Grasp domain /  Creatine Kinase; Chain A, domain 2 / Creatine Kinase; Chain A, domain 2 /  Glutamine synthetase, glycine-rich site / Glutamine synthetase putative ATP-binding region signature. / Glutamine synthetase, glycine-rich site / Glutamine synthetase putative ATP-binding region signature. /  Glutamine synthetase, N-terminal domain ... Glutamine synthetase, N-terminal domain ... Glutamine synthetase, N-terminal domain / Glutamine synthetase type I / Glutamine synthetase/guanido kinase, catalytic domain / Glutamine synthetase (GS) beta-grasp domain profile. / Glutamine synthetase (GS) catalytic domain profile. / Glutamine synthetase, N-terminal domain / Glutamine synthetase type I / Glutamine synthetase/guanido kinase, catalytic domain / Glutamine synthetase (GS) beta-grasp domain profile. / Glutamine synthetase (GS) catalytic domain profile. /  Glutamine synthetase, beta-Grasp domain / Glutamine synthetase, beta-Grasp domain /  Creatine Kinase; Chain A, domain 2 / Creatine Kinase; Chain A, domain 2 /  Glutamine synthetase, glycine-rich site / Glutamine synthetase putative ATP-binding region signature. / Glutamine synthetase, glycine-rich site / Glutamine synthetase putative ATP-binding region signature. /  Glutamine synthetase, N-terminal domain / Glutamine synthetase, N-terminal domain /  Glutamine synthetase, N-terminal domain superfamily / Glutamine synthetase, N-terminal domain superfamily /  Glutamine synthetase, catalytic domain / Glutamine synthetase, catalytic domain /  Glutamine synthetase, catalytic domain / Glutamine synthetase, catalytic domain /  Glutamine synthetase, catalytic domain / Glutamine synthetase/guanido kinase, catalytic domain / Ubiquitin-like (UB roll) / Roll / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 Glutamine synthetase, catalytic domain / Glutamine synthetase/guanido kinase, catalytic domain / Ubiquitin-like (UB roll) / Roll / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性ADENOSINE-5'-DIPHOSPHATE / ADENOSINE-5'-TRIPHOSPHATE / Chem-P3P /  グルホシネート / グルホシネート /  グルタミンシンテターゼ類似検索 - 構成要素 グルタミンシンテターゼ類似検索 - 構成要素 |
|---|
| 生物種 |  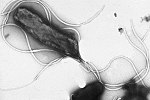  Helicobacter pylori (ピロリ菌) Helicobacter pylori (ピロリ菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2.93 Å シンクロトロン / 解像度: 2.93 Å |
|---|
 データ登録者 データ登録者 | Joo, H.K. / Lee, J.Y. |
|---|
| 資金援助 |  韓国, 2件 韓国, 2件 | 組織 | 認可番号 | 国 |
|---|
| National Research Foundation (Korea) | 2017R1D1A1B03032109 |  韓国 韓国 | | 710013-03-1-SB120 |  韓国 韓国 |
|
|---|
 引用 引用 |  ジャーナル: Sci Rep / 年: 2018 ジャーナル: Sci Rep / 年: 2018
タイトル: Structural Analysis of Glutamine Synthetase from Helicobacter pylori.
著者: Joo, H.K. / Park, Y.W. / Jang, Y.Y. / Lee, J.Y. |
|---|
| 履歴 | | 登録 | 2018年3月29日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2018年8月29日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年3月27日 | Group: Data collection / Database references / Derived calculations
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 グルタミンシンテターゼ
グルタミンシンテターゼ  キーワード
キーワード LIGASE (リガーゼ) /
LIGASE (リガーゼ) /  glutamine synthetase (グルタミンシンテターゼ) /
glutamine synthetase (グルタミンシンテターゼ) /  enzyme (酵素) / GS /
enzyme (酵素) / GS /  helicobacter pylori (ヘリコバクター・ピロリ) /
helicobacter pylori (ヘリコバクター・ピロリ) /  STRUCTURAL PROTEIN (タンパク質)
STRUCTURAL PROTEIN (タンパク質) 機能・相同性情報
機能・相同性情報 グルタミンシンテターゼ / glutamine biosynthetic process /
グルタミンシンテターゼ / glutamine biosynthetic process /  glutamine synthetase activity /
glutamine synthetase activity /  ATP binding /
ATP binding /  生体膜 /
生体膜 /  metal ion binding /
metal ion binding /  細胞質
細胞質
 Helicobacter pylori (ピロリ菌)
Helicobacter pylori (ピロリ菌) X線回折 /
X線回折 /  シンクロトロン / 解像度: 2.93 Å
シンクロトロン / 解像度: 2.93 Å  データ登録者
データ登録者 韓国, 2件
韓国, 2件  引用
引用 ジャーナル: Sci Rep / 年: 2018
ジャーナル: Sci Rep / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5zlp.cif.gz
5zlp.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5zlp.ent.gz
pdb5zlp.ent.gz PDB形式
PDB形式 5zlp.json.gz
5zlp.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/zl/5zlp
https://data.pdbj.org/pub/pdb/validation_reports/zl/5zlp ftp://data.pdbj.org/pub/pdb/validation_reports/zl/5zlp
ftp://data.pdbj.org/pub/pdb/validation_reports/zl/5zlp リンク
リンク 集合体
集合体
 要素
要素 グルタミンシンテターゼ / GS / Glutamate--ammonia ligase / Glutamine synthetase I beta / GSI beta
グルタミンシンテターゼ / GS / Glutamate--ammonia ligase / Glutamine synthetase I beta / GSI beta
 Helicobacter pylori (strain ATCC 700392 / 26695) (ピロリ菌)
Helicobacter pylori (strain ATCC 700392 / 26695) (ピロリ菌)
 Escherichia coli (大腸菌) / 参照: UniProt: P94845,
Escherichia coli (大腸菌) / 参照: UniProt: P94845,  グルタミンシンテターゼ
グルタミンシンテターゼ








 アデノシン三リン酸
アデノシン三リン酸 アデノシン二リン酸
アデノシン二リン酸 グルホシネート
グルホシネート 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト: PAL/PLS
シンクロトロン / サイト: PAL/PLS  / ビームライン: 5C (4A) / 波長: 0.9595 Å
/ ビームライン: 5C (4A) / 波長: 0.9595 Å : 0.9595 Å / 相対比: 1
: 0.9595 Å / 相対比: 1  解析
解析 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj





