| 登録情報 | データベース: PDB / ID: 5nm2
|
|---|
| タイトル | A2A Adenosine receptor cryo structure |
|---|
 要素 要素 | Adenosine receptor A2a,Soluble cytochrome b562,Adenosine receptor A2a |
|---|
 キーワード キーワード |  SIGNALING PROTEIN / room-temperature / serial crystallography / SIGNALING PROTEIN / room-temperature / serial crystallography /  hydrolase (加水分解酵素) hydrolase (加水分解酵素) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of acetylcholine secretion, neurotransmission / positive regulation of circadian sleep/wake cycle, sleep / regulation of norepinephrine secretion / negative regulation of alpha-beta T cell activation / Adenosine P1 receptors / G protein-coupled adenosine receptor activity / G protein-coupled adenosine receptor signaling pathway / response to purine-containing compound /  知覚 / positive regulation of urine volume ...positive regulation of acetylcholine secretion, neurotransmission / positive regulation of circadian sleep/wake cycle, sleep / regulation of norepinephrine secretion / negative regulation of alpha-beta T cell activation / Adenosine P1 receptors / G protein-coupled adenosine receptor activity / G protein-coupled adenosine receptor signaling pathway / response to purine-containing compound / 知覚 / positive regulation of urine volume ...positive regulation of acetylcholine secretion, neurotransmission / positive regulation of circadian sleep/wake cycle, sleep / regulation of norepinephrine secretion / negative regulation of alpha-beta T cell activation / Adenosine P1 receptors / G protein-coupled adenosine receptor activity / G protein-coupled adenosine receptor signaling pathway / response to purine-containing compound /  知覚 / positive regulation of urine volume / NGF-independant TRKA activation / Surfactant metabolism / protein kinase C-activating G protein-coupled receptor signaling pathway / 知覚 / positive regulation of urine volume / NGF-independant TRKA activation / Surfactant metabolism / protein kinase C-activating G protein-coupled receptor signaling pathway /  synaptic transmission, dopaminergic / synaptic transmission, dopaminergic /  inhibitory postsynaptic potential / negative regulation of vascular permeability / type 5 metabotropic glutamate receptor binding / positive regulation of glutamate secretion / inhibitory postsynaptic potential / negative regulation of vascular permeability / type 5 metabotropic glutamate receptor binding / positive regulation of glutamate secretion /  循環器 / 循環器 /  synaptic transmission, cholinergic / response to caffeine / synaptic transmission, cholinergic / response to caffeine /  中間径フィラメント / 中間径フィラメント /  eating behavior / presynaptic active zone / alpha-actinin binding / eating behavior / presynaptic active zone / alpha-actinin binding /  脱分極 / regulation of calcium ion transport / asymmetric synapse / 脱分極 / regulation of calcium ion transport / asymmetric synapse /  axolemma / response to inorganic substance / cellular defense response / axolemma / response to inorganic substance / cellular defense response /  prepulse inhibition / prepulse inhibition /  食作用 / positive regulation of apoptotic signaling pathway / response to amphetamine / 食作用 / positive regulation of apoptotic signaling pathway / response to amphetamine /  excitatory postsynaptic potential / presynaptic modulation of chemical synaptic transmission / positive regulation of synaptic transmission, glutamatergic / neuron projection morphogenesis / regulation of mitochondrial membrane potential / locomotory behavior / excitatory postsynaptic potential / presynaptic modulation of chemical synaptic transmission / positive regulation of synaptic transmission, glutamatergic / neuron projection morphogenesis / regulation of mitochondrial membrane potential / locomotory behavior /  central nervous system development / central nervous system development /  synaptic transmission, glutamatergic / positive regulation of long-term synaptic potentiation / astrocyte activation / apoptotic signaling pathway / positive regulation of synaptic transmission, GABAergic / positive regulation of protein secretion / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / negative regulation of inflammatory response / synaptic transmission, glutamatergic / positive regulation of long-term synaptic potentiation / astrocyte activation / apoptotic signaling pathway / positive regulation of synaptic transmission, GABAergic / positive regulation of protein secretion / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / negative regulation of inflammatory response /  vasodilation / vasodilation /  凝固・線溶系 / cell-cell signaling / 凝固・線溶系 / cell-cell signaling /  presynaptic membrane / G alpha (s) signalling events / presynaptic membrane / G alpha (s) signalling events /  postsynaptic membrane / negative regulation of neuron apoptotic process / postsynaptic membrane / negative regulation of neuron apoptotic process /  electron transfer activity / electron transfer activity /  ペリプラズム / ペリプラズム /  calmodulin binding / response to xenobiotic stimulus / calmodulin binding / response to xenobiotic stimulus /  炎症 / iron ion binding / negative regulation of cell population proliferation / neuronal cell body / 炎症 / iron ion binding / negative regulation of cell population proliferation / neuronal cell body /  樹状突起 / glutamatergic synapse / apoptotic process / 樹状突起 / glutamatergic synapse / apoptotic process /  lipid binding / lipid binding /  heme binding / protein-containing complex binding / regulation of DNA-templated transcription / heme binding / protein-containing complex binding / regulation of DNA-templated transcription /  enzyme binding / enzyme binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜類似検索 - 分子機能 細胞膜類似検索 - 分子機能  Adenosine A2A receptor / Adenosine A2A receptor /  アデノシン受容体 / Cytochrome b562 / Cytochrome b562 / Cytochrome c/b562 / Serpentine type 7TM GPCR chemoreceptor Srsx / G-protein coupled receptors family 1 signature. / G protein-coupled receptor, rhodopsin-like / アデノシン受容体 / Cytochrome b562 / Cytochrome b562 / Cytochrome c/b562 / Serpentine type 7TM GPCR chemoreceptor Srsx / G-protein coupled receptors family 1 signature. / G protein-coupled receptor, rhodopsin-like /  GPCR, rhodopsin-like, 7TM / G-protein coupled receptors family 1 profile. / 7 transmembrane receptor (rhodopsin family)類似検索 - ドメイン・相同性 GPCR, rhodopsin-like, 7TM / G-protein coupled receptors family 1 profile. / 7 transmembrane receptor (rhodopsin family)類似検索 - ドメイン・相同性 コレステロール / コレステロール /  オレイン酸 / (2S)-2,3-dihydroxypropyl (9Z)-octadec-9-enoate / (2R)-2,3-dihydroxypropyl (9Z)-octadec-9-enoate / Chem-ZMA / Soluble cytochrome b562 / オレイン酸 / (2S)-2,3-dihydroxypropyl (9Z)-octadec-9-enoate / (2R)-2,3-dihydroxypropyl (9Z)-octadec-9-enoate / Chem-ZMA / Soluble cytochrome b562 /  Adenosine receptor A2a類似検索 - 構成要素 Adenosine receptor A2a類似検索 - 構成要素 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)
   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.948 Å 分子置換 / 解像度: 1.948 Å |
|---|
 データ登録者 データ登録者 | Weinert, T. / Cheng, R. / James, D. / Gashi, D. / Nogly, P. / Jaeger, K. / Dore, A.S. / Geng, T. / Cooke, R. / Hennig, M. / Standfuss, J. |
|---|
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017
タイトル: Serial millisecond crystallography for routine room-temperature structure determination at synchrotrons.
著者: Weinert, T. / Olieric, N. / Cheng, R. / Brunle, S. / James, D. / Ozerov, D. / Gashi, D. / Vera, L. / Marsh, M. / Jaeger, K. / Dworkowski, F. / Panepucci, E. / Basu, S. / Skopintsev, P. / ...著者: Weinert, T. / Olieric, N. / Cheng, R. / Brunle, S. / James, D. / Ozerov, D. / Gashi, D. / Vera, L. / Marsh, M. / Jaeger, K. / Dworkowski, F. / Panepucci, E. / Basu, S. / Skopintsev, P. / Dore, A.S. / Geng, T. / Cooke, R.M. / Liang, M. / Prota, A.E. / Panneels, V. / Nogly, P. / Ermler, U. / Schertler, G. / Hennig, M. / Steinmetz, M.O. / Wang, M. / Standfuss, J. |
|---|
| 履歴 | | 登録 | 2017年4月5日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2017年9月27日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年8月14日 | Group: Data collection / カテゴリ: reflns / reflns_shell
Item: _reflns.pdbx_Rrim_I_all / _reflns_shell.pdbx_Rrim_I_all |
|---|
| 改定 1.2 | 2024年1月17日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード SIGNALING PROTEIN / room-temperature / serial crystallography /
SIGNALING PROTEIN / room-temperature / serial crystallography /  hydrolase (加水分解酵素)
hydrolase (加水分解酵素) 機能・相同性情報
機能・相同性情報 知覚 / positive regulation of urine volume ...positive regulation of acetylcholine secretion, neurotransmission / positive regulation of circadian sleep/wake cycle, sleep / regulation of norepinephrine secretion / negative regulation of alpha-beta T cell activation / Adenosine P1 receptors / G protein-coupled adenosine receptor activity / G protein-coupled adenosine receptor signaling pathway / response to purine-containing compound /
知覚 / positive regulation of urine volume ...positive regulation of acetylcholine secretion, neurotransmission / positive regulation of circadian sleep/wake cycle, sleep / regulation of norepinephrine secretion / negative regulation of alpha-beta T cell activation / Adenosine P1 receptors / G protein-coupled adenosine receptor activity / G protein-coupled adenosine receptor signaling pathway / response to purine-containing compound /  知覚 / positive regulation of urine volume / NGF-independant TRKA activation / Surfactant metabolism / protein kinase C-activating G protein-coupled receptor signaling pathway /
知覚 / positive regulation of urine volume / NGF-independant TRKA activation / Surfactant metabolism / protein kinase C-activating G protein-coupled receptor signaling pathway /  synaptic transmission, dopaminergic /
synaptic transmission, dopaminergic /  inhibitory postsynaptic potential / negative regulation of vascular permeability / type 5 metabotropic glutamate receptor binding / positive regulation of glutamate secretion /
inhibitory postsynaptic potential / negative regulation of vascular permeability / type 5 metabotropic glutamate receptor binding / positive regulation of glutamate secretion /  循環器 /
循環器 /  synaptic transmission, cholinergic / response to caffeine /
synaptic transmission, cholinergic / response to caffeine /  中間径フィラメント /
中間径フィラメント /  eating behavior / presynaptic active zone / alpha-actinin binding /
eating behavior / presynaptic active zone / alpha-actinin binding /  脱分極 / regulation of calcium ion transport / asymmetric synapse /
脱分極 / regulation of calcium ion transport / asymmetric synapse /  axolemma / response to inorganic substance / cellular defense response /
axolemma / response to inorganic substance / cellular defense response /  prepulse inhibition /
prepulse inhibition /  食作用 / positive regulation of apoptotic signaling pathway / response to amphetamine /
食作用 / positive regulation of apoptotic signaling pathway / response to amphetamine /  excitatory postsynaptic potential / presynaptic modulation of chemical synaptic transmission / positive regulation of synaptic transmission, glutamatergic / neuron projection morphogenesis / regulation of mitochondrial membrane potential / locomotory behavior /
excitatory postsynaptic potential / presynaptic modulation of chemical synaptic transmission / positive regulation of synaptic transmission, glutamatergic / neuron projection morphogenesis / regulation of mitochondrial membrane potential / locomotory behavior /  central nervous system development /
central nervous system development /  synaptic transmission, glutamatergic / positive regulation of long-term synaptic potentiation / astrocyte activation / apoptotic signaling pathway / positive regulation of synaptic transmission, GABAergic / positive regulation of protein secretion / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / negative regulation of inflammatory response /
synaptic transmission, glutamatergic / positive regulation of long-term synaptic potentiation / astrocyte activation / apoptotic signaling pathway / positive regulation of synaptic transmission, GABAergic / positive regulation of protein secretion / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / negative regulation of inflammatory response /  vasodilation /
vasodilation /  凝固・線溶系 / cell-cell signaling /
凝固・線溶系 / cell-cell signaling /  presynaptic membrane / G alpha (s) signalling events /
presynaptic membrane / G alpha (s) signalling events /  postsynaptic membrane / negative regulation of neuron apoptotic process /
postsynaptic membrane / negative regulation of neuron apoptotic process /  electron transfer activity /
electron transfer activity /  ペリプラズム /
ペリプラズム /  calmodulin binding / response to xenobiotic stimulus /
calmodulin binding / response to xenobiotic stimulus /  炎症 / iron ion binding / negative regulation of cell population proliferation / neuronal cell body /
炎症 / iron ion binding / negative regulation of cell population proliferation / neuronal cell body /  樹状突起 / glutamatergic synapse / apoptotic process /
樹状突起 / glutamatergic synapse / apoptotic process /  lipid binding /
lipid binding /  heme binding / protein-containing complex binding / regulation of DNA-templated transcription /
heme binding / protein-containing complex binding / regulation of DNA-templated transcription /  enzyme binding /
enzyme binding /  生体膜 / identical protein binding /
生体膜 / identical protein binding /  細胞膜
細胞膜
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.948 Å
分子置換 / 解像度: 1.948 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat Commun / 年: 2017
ジャーナル: Nat Commun / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5nm2.cif.gz
5nm2.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5nm2.ent.gz
pdb5nm2.ent.gz PDB形式
PDB形式 5nm2.json.gz
5nm2.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/nm/5nm2
https://data.pdbj.org/pub/pdb/validation_reports/nm/5nm2 ftp://data.pdbj.org/pub/pdb/validation_reports/nm/5nm2
ftp://data.pdbj.org/pub/pdb/validation_reports/nm/5nm2 リンク
リンク 集合体
集合体
 要素
要素
 Homo sapiens (ヒト), (組換発現)
Homo sapiens (ヒト), (組換発現) 
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌)
 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P29274, UniProt: P0ABE7
Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P29274, UniProt: P0ABE7
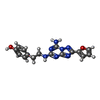

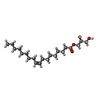









 ZM-241,385
ZM-241,385 オレイン酸
オレイン酸 コレステロール
コレステロール 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06SA / 波長: 1 Å
/ ビームライン: X06SA / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj






















