| 登録情報 | データベース: PDB / ID: 5mvb
|
|---|
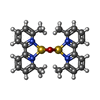
| タイトル | Solution structure of a human G-Quadruplex hybrid-2 form in complex with a Gold-ligand. |
|---|
 要素 要素 | DNA (26-MER) |
|---|
 キーワード キーワード |  DNA (デオキシリボ核酸) / G-Quadruplex hybrid / 2 Auoxo6 DNA (デオキシリボ核酸) / G-Quadruplex hybrid / 2 Auoxo6 |
|---|
| 機能・相同性 | Chem-AUZ / : /  デオキシリボ核酸 / DNA (> 10) デオキシリボ核酸 / DNA (> 10) 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  溶液NMR / 溶液NMR /  simulated annealing simulated annealing |
|---|
 データ登録者 データ登録者 | Wirmer-Bartoschek, J. / Jonker, H.R.A. / Bendel, L.E. / Gruen, T. / Bazzicalupi, C. / Messori, L. / Gratteri, P. / Schwalbe, H. |
|---|
| 資金援助 |  ドイツ, 1件 ドイツ, 1件 | 組織 | 認可番号 | 国 |
|---|
| Land Hessen | LOEWE SynChemBio |  ドイツ ドイツ |
|
|---|
 引用 引用 |  ジャーナル: Angew. Chem. Int. Ed. Engl. / 年: 2017 ジャーナル: Angew. Chem. Int. Ed. Engl. / 年: 2017
タイトル: Solution NMR Structure of a Ligand/Hybrid-2-G-Quadruplex Complex Reveals Rearrangements that Affect Ligand Binding.
著者: Wirmer-Bartoschek, J. / Bendel, L.E. / Jonker, H.R.A. / Grun, J.T. / Papi, F. / Bazzicalupi, C. / Messori, L. / Gratteri, P. / Schwalbe, H. |
|---|
| 履歴 | | 登録 | 2017年1月16日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2017年5月31日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年6月14日 | Group: Database references / カテゴリ: citation
Item: _citation.country / _citation.journal_id_ASTM ..._citation.country / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.2 | 2018年1月17日 | Group: Structure summary / カテゴリ: struct / Item: _struct.title |
|---|
| 改定 1.3 | 2019年5月8日 | Group: Data collection
カテゴリ: pdbx_nmr_software / pdbx_seq_map_depositor_info
Item: _pdbx_nmr_software.name / _pdbx_seq_map_depositor_info.one_letter_code_mod |
|---|
| 改定 1.4 | 2023年6月14日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Other
カテゴリ: database_2 / pdbx_database_status ...database_2 / pdbx_database_status / pdbx_nmr_spectrometer / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data / _pdbx_nmr_spectrometer.model / _struct_conn.pdbx_dist_value / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード DNA (デオキシリボ核酸) / G-Quadruplex hybrid / 2 Auoxo6
DNA (デオキシリボ核酸) / G-Quadruplex hybrid / 2 Auoxo6 デオキシリボ核酸 / DNA (> 10)
デオキシリボ核酸 / DNA (> 10) 機能・相同性情報
機能・相同性情報
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR /
溶液NMR /  simulated annealing
simulated annealing  データ登録者
データ登録者 ドイツ, 1件
ドイツ, 1件  引用
引用 ジャーナル: Angew. Chem. Int. Ed. Engl. / 年: 2017
ジャーナル: Angew. Chem. Int. Ed. Engl. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5mvb.cif.gz
5mvb.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5mvb.ent.gz
pdb5mvb.ent.gz PDB形式
PDB形式 5mvb.json.gz
5mvb.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/mv/5mvb
https://data.pdbj.org/pub/pdb/validation_reports/mv/5mvb ftp://data.pdbj.org/pub/pdb/validation_reports/mv/5mvb
ftp://data.pdbj.org/pub/pdb/validation_reports/mv/5mvb リンク
リンク 集合体
集合体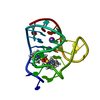
 要素
要素
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR
溶液NMR 試料調製
試料調製 解析
解析 simulated annealing / ソフトェア番号: 6
simulated annealing / ソフトェア番号: 6  ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj