+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4cgb | ||||||
|---|---|---|---|---|---|---|---|
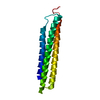
| タイトル | Crystal structure of the trimerization domain of EML2 | ||||||
 要素 要素 | ECHINODERM MICROTUBULE-ASSOCIATED PROTEIN-LIKE 2 | ||||||
 キーワード キーワード |  CELL CYCLE (細胞周期) / CELL CYCLE (細胞周期) /  COILED-COIL (コイルドコイル) COILED-COIL (コイルドコイル) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 regulation of microtubule nucleation / regulation of microtubule nucleation /  : / microtubule associated complex / negative regulation of microtubule polymerization / : / microtubule associated complex / negative regulation of microtubule polymerization /  視覚 / 視覚 /  tubulin binding / sensory perception of sound / microtubule cytoskeleton organization / spindle / tubulin binding / sensory perception of sound / microtubule cytoskeleton organization / spindle /  microtubule binding ... microtubule binding ... regulation of microtubule nucleation / regulation of microtubule nucleation /  : / microtubule associated complex / negative regulation of microtubule polymerization / : / microtubule associated complex / negative regulation of microtubule polymerization /  視覚 / 視覚 /  tubulin binding / sensory perception of sound / microtubule cytoskeleton organization / spindle / tubulin binding / sensory perception of sound / microtubule cytoskeleton organization / spindle /  microtubule binding / microtubule binding /  微小管 / 微小管 /  signaling receptor binding / signaling receptor binding /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.154 Å 分子置換 / 解像度: 2.154 Å | ||||||
 データ登録者 データ登録者 | Richards, M.W. / Bayliss, R. | ||||||
 引用 引用 |  ジャーナル: Biochem.J. / 年: 2015 ジャーナル: Biochem.J. / 年: 2015タイトル: Microtubule Association of Eml Proteins and the Eml4-Alk Variant 3 Oncoprotein Require an N-Terminal Trimerization Domain. 著者: Richards, M.W. / O'Regan, L. / Roth, D. / Montgomery, J.M. / Straube, A. / Fry, A.M. / Bayliss, R. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4cgb.cif.gz 4cgb.cif.gz | 111.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4cgb.ent.gz pdb4cgb.ent.gz | 90.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4cgb.json.gz 4cgb.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cg/4cgb https://data.pdbj.org/pub/pdb/validation_reports/cg/4cgb ftp://data.pdbj.org/pub/pdb/validation_reports/cg/4cgb ftp://data.pdbj.org/pub/pdb/validation_reports/cg/4cgb | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 5601.340 Da / 分子数: 6 / 断片: TRIMERIZATION DOMAIN, RESIDUES 11-60 / 由来タイプ: 組換発現 / 由来: (組換発現)   HOMO SAPIENS (ヒト) / 器官: BRAIN AND SPINAL CORD / プラスミド: PET30TEV / 発現宿主: HOMO SAPIENS (ヒト) / 器官: BRAIN AND SPINAL CORD / プラスミド: PET30TEV / 発現宿主:   ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): RIL / 参照: UniProt: O95834 ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): RIL / 参照: UniProt: O95834#2: 化合物 | #3: 化合物 | ChemComp-GOL / |  グリセリン グリセリン#4: 水 | ChemComp-HOH / |  水 水配列の詳細 | THE N-TERMINAL GLYCINE RESIDUE IS NOT PART OF THE NATIVE SEQUENCE AND DERIVES FROM THE TEV CLEAVAGE ...THE N-TERMINAL GLYCINE RESIDUE IS NOT PART OF THE NATIVE SEQUENCE AND DERIVES FROM THE TEV CLEAVAGE SITE. SEQUENCE USED HERE IS THE ISOFORM-2 OF O95834 | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.86 Å3/Da / 溶媒含有率: 33.7 % / 解説: NONE |
|---|---|
結晶化 | 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6.5 詳細: CRYSTALS WERE OBTAINED BY THE HANGING DROP VAPOUR DIFFUSION METHOD USING 100 MM BIS-TRIS-PROPANE PH 6.5, 20% PEG 3350, 200 MM POTASSIUM THIOCYANATE IN THE RESERVOIR. |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I04 / 波長: 0.9795 / ビームライン: I04 / 波長: 0.9795 |
| 検出器 | タイプ: DECTRIS PILATUS / 検出器: PIXEL |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9795 Å / 相対比: 1 : 0.9795 Å / 相対比: 1 |
| 反射 | 解像度: 2.15→50.86 Å / Num. obs: 14593 / % possible obs: 99.8 % / Observed criterion σ(I): 2 / 冗長度: 7 % / Biso Wilson estimate: 28.61 Å2 / Rmerge(I) obs: 0.08 / Net I/σ(I): 14.2 |
| 反射 シェル | 解像度: 2.15→2.32 Å / 冗長度: 7.3 % / Rmerge(I) obs: 0.64 / Mean I/σ(I) obs: 3.2 / % possible all: 99.9 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 4CGC 解像度: 2.154→50.846 Å / SU ML: 0.2 / σ(F): 1.02 / 位相誤差: 23.5 / 立体化学のターゲット値: ML 詳細: IN CHAIN A RESIDUES 14-57 ARE ORDERED. IN CHAIN B RESIDUES 14-58 ARE ORDERED. IN CHAIN C RESIDUES 13-53 ARE ORDERED. IN CHAIN D RESIDUES 13-57 ARE ORDERED. IN CHAIN E RESIDUES 13-52 ARE ...詳細: IN CHAIN A RESIDUES 14-57 ARE ORDERED. IN CHAIN B RESIDUES 14-58 ARE ORDERED. IN CHAIN C RESIDUES 13-53 ARE ORDERED. IN CHAIN D RESIDUES 13-57 ARE ORDERED. IN CHAIN E RESIDUES 13-52 ARE ORDERED. IN CHAIN F RESIDUES 13-57 ARE ORDERED.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.98 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL / Bsol: 43.888 Å2 / ksol: 0.378 e/Å3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.154→50.846 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj





