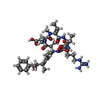
登録情報 データベース : PDB / ID : 3e7aタイトル Crystal Structure of Protein Phosphatase-1 Bound to the natural toxin Nodularin-R Serine/threonine-protein phosphatase PP1-alpha catalytic subunit nodularin R キーワード / / / / / / / / / / / 機能・相同性 生物種 Homo sapiens (ヒト)Nodularia spumigena (バクテリア)手法 / / / 解像度 : 1.63 Å データ登録者 Kelker, M.S. / Page, R. / Peti, W. ジャーナル : J.Mol.Biol. / 年 : 2009タイトル : Crystal structures of protein phosphatase-1 bound to nodularin-R and tautomycin: a novel scaffold for structure-based drug design of serine/threonine phosphatase inhibitors著者 : Kelker, M.S. / Page, R. / Peti, W. 履歴 登録 2008年8月18日 登録サイト / 処理サイト 改定 1.0 2008年11月4日 Provider / タイプ 改定 1.1 2011年7月13日 Group Advisory / Atomic model ... Advisory / Atomic model / Database references / Derived calculations / Non-polymer description / Structure summary / Version format compliance 改定 1.2 2013年2月27日 Group 改定 1.3 2013年4月10日 Group 改定 1.4 2017年10月25日 Group / Refinement description / カテゴリ / software改定 1.5 2023年8月30日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_asym_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_ref_seq_dif.details 改定 2.0 2023年11月15日 Group / Data collection / Derived calculationsカテゴリ atom_site / chem_comp_atom ... atom_site / chem_comp_atom / chem_comp_bond / pdbx_validate_main_chain_plane / pdbx_validate_peptide_omega / struct_conn Item _atom_site.auth_atom_id / _atom_site.label_atom_id ... _atom_site.auth_atom_id / _atom_site.label_atom_id / _chem_comp_atom.atom_id / _chem_comp_bond.atom_id_1 / _chem_comp_bond.atom_id_2 / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr2_label_atom_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード Carbohydrate metabolism (炭水化物代謝) /
Carbohydrate metabolism (炭水化物代謝) /  Cell cycle (細胞周期) /
Cell cycle (細胞周期) /  Cell division (細胞分裂) / Glycogen metabolism /
Cell division (細胞分裂) / Glycogen metabolism /  Hydrolase (加水分解酵素) /
Hydrolase (加水分解酵素) /  Iron (鉄) /
Iron (鉄) /  Manganese (マンガン) / Metal-binding /
Manganese (マンガン) / Metal-binding /  Phosphoprotein /
Phosphoprotein /  Protein phosphatase (プロテインホスファターゼ) / HYDROLASE-HYDROLASE INHIBITOR COMPLEX
Protein phosphatase (プロテインホスファターゼ) / HYDROLASE-HYDROLASE INHIBITOR COMPLEX 機能・相同性情報
機能・相同性情報 グリコーゲン / regulation of glycogen biosynthetic process /
グリコーゲン / regulation of glycogen biosynthetic process /  protein phosphatase 1 binding / cadherin binding involved in cell-cell adhesion / positive regulation of extrinsic apoptotic signaling pathway in absence of ligand / regulation of canonical Wnt signaling pathway / regulation of translational initiation / myosin phosphatase activity ...regulation of glycogen catabolic process / PTW/PP1 phosphatase complex /
protein phosphatase 1 binding / cadherin binding involved in cell-cell adhesion / positive regulation of extrinsic apoptotic signaling pathway in absence of ligand / regulation of canonical Wnt signaling pathway / regulation of translational initiation / myosin phosphatase activity ...regulation of glycogen catabolic process / PTW/PP1 phosphatase complex /  グリコーゲン / regulation of glycogen biosynthetic process /
グリコーゲン / regulation of glycogen biosynthetic process /  protein phosphatase 1 binding / cadherin binding involved in cell-cell adhesion / positive regulation of extrinsic apoptotic signaling pathway in absence of ligand / regulation of canonical Wnt signaling pathway / regulation of translational initiation / myosin phosphatase activity /
protein phosphatase 1 binding / cadherin binding involved in cell-cell adhesion / positive regulation of extrinsic apoptotic signaling pathway in absence of ligand / regulation of canonical Wnt signaling pathway / regulation of translational initiation / myosin phosphatase activity /  protein serine/threonine phosphatase activity / branching morphogenesis of an epithelial tube / glycogen metabolic process / protein-serine/threonine phosphatase / entrainment of circadian clock by photoperiod / Triglyceride catabolism /
protein serine/threonine phosphatase activity / branching morphogenesis of an epithelial tube / glycogen metabolic process / protein-serine/threonine phosphatase / entrainment of circadian clock by photoperiod / Triglyceride catabolism /  phosphatase activity /
phosphatase activity /  phosphoprotein phosphatase activity / DARPP-32 events / ribonucleoprotein complex binding /
phosphoprotein phosphatase activity / DARPP-32 events / ribonucleoprotein complex binding /  脱リン酸化 / protein dephosphorylation / Downregulation of TGF-beta receptor signaling / response to lead ion /
脱リン酸化 / protein dephosphorylation / Downregulation of TGF-beta receptor signaling / response to lead ion /  接着結合 / lung development / circadian regulation of gene expression /
接着結合 / lung development / circadian regulation of gene expression /  regulation of circadian rhythm /
regulation of circadian rhythm /  Circadian Clock / presynapse /
Circadian Clock / presynapse /  perikaryon /
perikaryon /  樹状突起スパイン /
樹状突起スパイン /  細胞周期 /
細胞周期 /  細胞分裂 / glutamatergic synapse /
細胞分裂 / glutamatergic synapse /  核小体 / extracellular exosome /
核小体 / extracellular exosome /  核質 /
核質 /  metal ion binding /
metal ion binding /  細胞核 /
細胞核 /  細胞膜 /
細胞膜 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) Nodularia spumigena (バクテリア)
Nodularia spumigena (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.63 Å
分子置換 / 解像度: 1.63 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 2009
ジャーナル: J.Mol.Biol. / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3e7a.cif.gz
3e7a.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3e7a.ent.gz
pdb3e7a.ent.gz PDB形式
PDB形式 3e7a.json.gz
3e7a.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/e7/3e7a
https://data.pdbj.org/pub/pdb/validation_reports/e7/3e7a ftp://data.pdbj.org/pub/pdb/validation_reports/e7/3e7a
ftp://data.pdbj.org/pub/pdb/validation_reports/e7/3e7a リンク
リンク 集合体
集合体

 要素
要素
 Homo sapiens (ヒト) / 遺伝子: PPP1CA, PPP1A / プラスミド: in house derived / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: PPP1CA, PPP1A / プラスミド: in house derived / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21 (DE3)
Escherichia coli (大腸菌) / 株 (発現宿主): BL21 (DE3) ノジュラリン
ノジュラリン










 ヨウ化物
ヨウ化物 塩化物
塩化物 グリセリン
グリセリン アジ化物
アジ化物 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X6A / 波長: 1 Å
/ ビームライン: X6A / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1 
 分子置換
分子置換 解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj