+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2ymn | ||||||
|---|---|---|---|---|---|---|---|
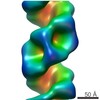
| タイトル | Organization of the Influenza Virus Replication Machinery | ||||||
 要素 要素 | NUCLEOPROTEIN 核タンパク質 核タンパク質 | ||||||
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / RNP / VIRAL PROTEIN (ウイルスタンパク質) / RNP /  POLYMERASE (ポリメラーゼ) POLYMERASE (ポリメラーゼ) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報helical viral capsid / viral penetration into host nucleus / viral nucleocapsid /  ribonucleoprotein complex / symbiont entry into host cell / host cell nucleus / structural molecule activity / ribonucleoprotein complex / symbiont entry into host cell / host cell nucleus / structural molecule activity /  RNA binding / identical protein binding RNA binding / identical protein binding類似検索 - 分子機能 | ||||||
| 生物種 |    INFLUENZA A VIRUS (A型インフルエンザウイルス) INFLUENZA A VIRUS (A型インフルエンザウイルス) | ||||||
| 手法 |  電子顕微鏡法 / らせん対称体再構成法 / 電子顕微鏡法 / らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 20 Å クライオ電子顕微鏡法 / 解像度: 20 Å | ||||||
 データ登録者 データ登録者 | Moeller, A. / Kirchdoerfer, R.N. / Potter, C.S. / Carragher, B. / Wilson, I.A. | ||||||
 引用 引用 |  ジャーナル: Science / 年: 2012 ジャーナル: Science / 年: 2012タイトル: Organization of the influenza virus replication machinery. 著者: Arne Moeller / Robert N Kirchdoerfer / Clinton S Potter / Bridget Carragher / Ian A Wilson /  要旨: Influenza virus ribonucleoprotein complexes (RNPs) are central to the viral life cycle and in adaptation to new host species. RNPs are composed of the viral genome, viral polymerase, and many copies ...Influenza virus ribonucleoprotein complexes (RNPs) are central to the viral life cycle and in adaptation to new host species. RNPs are composed of the viral genome, viral polymerase, and many copies of the viral nucleoprotein. In vitro cell expression of all RNP protein components with four of the eight influenza virus gene segments enabled structural determination of native influenza virus RNPs by means of cryogenic electron microscopy (cryo-EM). The cryo-EM structure reveals the architecture and organization of the native RNP, defining the attributes of its largely helical structure and how polymerase interacts with nucleoprotein and the viral genome. Observations of branched-RNP structures in negative-stain electron microscopy and their putative identification as replication intermediates suggest a mechanism for viral replication by a second polymerase on the RNP template. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2ymn.cif.gz 2ymn.cif.gz | 461.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2ymn.ent.gz pdb2ymn.ent.gz | 389.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2ymn.json.gz 2ymn.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ym/2ymn https://data.pdbj.org/pub/pdb/validation_reports/ym/2ymn ftp://data.pdbj.org/pub/pdb/validation_reports/ym/2ymn ftp://data.pdbj.org/pub/pdb/validation_reports/ym/2ymn | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  核タンパク質 / NUCLEOCAPSID PROTEIN / PROTEIN N 核タンパク質 / NUCLEOCAPSID PROTEIN / PROTEIN N分子量: 56454.793 Da / 分子数: 6 / 由来タイプ: 組換発現 詳細: HELICAL ASSEMBLY OF NUCLEOPROTEINS WITHIN INFLUENZA RNP FILAMENT 由来: (組換発現)   INFLUENZA A VIRUS (A/PUERTO RICO/8/1934(H1N1)) (A型インフルエンザウイルス) INFLUENZA A VIRUS (A/PUERTO RICO/8/1934(H1N1)) (A型インフルエンザウイルス)細胞株 (発現宿主): HEK 293T17 / 発現宿主:   HOMO SAPIENS (ヒト) / 参照: UniProt: Q1I2B5, UniProt: Q1K9H2*PLUS HOMO SAPIENS (ヒト) / 参照: UniProt: Q1I2B5, UniProt: Q1K9H2*PLUS |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: らせん対称体再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: INFLUENZA VIRUS RIBONUCLEOPROTEIN COMPLEX CENTRAL FILAMENT REGION タイプ: VIRUS |
|---|---|
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
| 試料支持 | 詳細: HOLEY CARBON |
急速凍結 | 装置: FEI VITROBOT MARK II / 凍結剤: ETHANE 詳細: VITRIFICATION 1 -- CRYOGEN- ETHANE, HUMIDITY- 85, INSTRUMENT- FEI VITROBOT MARK II, |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: FEI TECNAI 20 / 日付: 2010年8月27日 |
|---|---|
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 120 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 120 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 50000 X Bright-field microscopy / 倍率(公称値): 50000 X |
| 撮影 | 電子線照射量: 15 e/Å2 / フィルム・検出器のモデル: GENERIC CCD |
| 放射波長 | 相対比: 1 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | 詳細: WHOLEIMAGE | ||||||||||||||||||||||||||||||||
3次元再構成 | 手法: IHRSR / 解像度: 20 Å / 粒子像の数: 31573 / ピクセルサイズ(公称値): 1.64 Å / ピクセルサイズ(実測値): 1.64 Å 詳細: SUBMISSION BASED ON EXPERIMENTAL DATA FROM EMDB EMD-2209.(DEPOSITION ID: 11133) 対称性のタイプ: HELICAL | ||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: OTHER / 空間: REAL / 詳細: REFINEMENT PROTOCOL--X-RAY | ||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 2IQH | ||||||||||||||||||||||||||||||||
| 精密化 | 最高解像度: 20 Å | ||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 20 Å
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj
