+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2y65 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of Drosophila melanogaster kinesin-1 motor domain dimer-tail complex | ||||||
 要素 要素 | (KINESIN HEAVY CHAIN キネシン) x 2 キネシン) x 2 | ||||||
 キーワード キーワード |  MOTOR PROTEIN (モータータンパク質) MOTOR PROTEIN (モータータンパク質) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報actin filament bundle organization / anterograde axonal transport of mitochondrion / anterograde dendritic transport / ovarian nurse cell to oocyte transport / mitochondrion distribution / larval locomotory behavior / oocyte microtubule cytoskeleton polarization / eye photoreceptor cell differentiation / regulation of pole plasm oskar mRNA localization / pole plasm oskar mRNA localization ...actin filament bundle organization / anterograde axonal transport of mitochondrion / anterograde dendritic transport / ovarian nurse cell to oocyte transport / mitochondrion distribution / larval locomotory behavior / oocyte microtubule cytoskeleton polarization / eye photoreceptor cell differentiation / regulation of pole plasm oskar mRNA localization / pole plasm oskar mRNA localization / oocyte dorsal/ventral axis specification / pole plasm assembly / microtubule sliding / dorsal appendage formation / larval somatic muscle development / COPI-dependent Golgi-to-ER retrograde traffic / Kinesins / transport along microtubule / centrosome separation / anterograde dendritic transport of neurotransmitter receptor complex / actin cap / microtubule plus-end / plus-end-directed microtubule motor activity / axo-dendritic transport / stress granule disassembly / nuclear migration / dendrite morphogenesis /  kinesin complex / synaptic vesicle transport / kinesin complex / synaptic vesicle transport /  microtubule motor activity / microtubule motor activity /  tropomyosin binding / intracellular distribution of mitochondria / microtubule-based movement / tropomyosin binding / intracellular distribution of mitochondria / microtubule-based movement /  微小管 / cytoskeletal motor activity / axon cytoplasm / dendrite cytoplasm / 微小管 / cytoskeletal motor activity / axon cytoplasm / dendrite cytoplasm /  軸索誘導 / 軸索誘導 /  軸索誘導 / 軸索誘導 /  microtubule binding / microtubule binding /  微小管 / 微小管 /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   DROSOPHILA MELANOGASTER (キイロショウジョウバエ) DROSOPHILA MELANOGASTER (キイロショウジョウバエ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.2 Å 分子置換 / 解像度: 2.2 Å | ||||||
 データ登録者 データ登録者 | Kaan, H.Y.K. / Hackney, D.D. / Kozielski, F. | ||||||
 引用 引用 |  ジャーナル: Science / 年: 2011 ジャーナル: Science / 年: 2011タイトル: The Structure of the Kinesin-1 Motor-Tail Complex Reveals the Mechanism of Autoinhibition. 著者: Kaan, H.Y.K. / Hackney, D.D. / Kozielski, F. | ||||||
| 履歴 |
| ||||||
| Remark 700 | SHEET THE SHEET STRUCTURE OF THIS MOLECULE IS BIFURCATED. IN ORDER TO REPRESENT THIS FEATURE IN ... SHEET THE SHEET STRUCTURE OF THIS MOLECULE IS BIFURCATED. IN ORDER TO REPRESENT THIS FEATURE IN THE SHEET RECORDS BELOW, TWO SHEETS ARE DEFINED. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2y65.cif.gz 2y65.cif.gz | 301.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2y65.ent.gz pdb2y65.ent.gz | 243.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2y65.json.gz 2y65.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/y6/2y65 https://data.pdbj.org/pub/pdb/validation_reports/y6/2y65 ftp://data.pdbj.org/pub/pdb/validation_reports/y6/2y65 ftp://data.pdbj.org/pub/pdb/validation_reports/y6/2y65 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 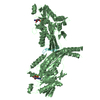
| ||||||||
| 3 | 
| ||||||||
| 単位格子 |
| ||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: given Matrix: (-0.9805, 0.0458, -0.1911), ベクター  : : 詳細 | OWING TO ITS AMINO ACID SEQUENCE, CHAINS X AND Y ARE ALMOST SYMMETRICAL, IN TERMS OF SIDE CHAIN PROPERTIES, ABOUT THE LYS 944 RESIDUE. THIS DISTINCTIVE FEATURE OF THE CHAINS X AND Y ALLOWS THEM TO BIND IN TWO DIRECTIONS BETWEEN THE DIMERS A-A' AND B-B', WHICH ALSO EXHIBIT TWO-FOLD SYMMETRY. | |
- 要素
要素
| #1: タンパク質 |  キネシン / KINESIN キネシン / KINESIN分子量: 40885.301 Da / 分子数: 4 / 断片: MOTOR DOMAIN, RESIDUES 1-365 / 由来タイプ: 組換発現 由来: (組換発現)   DROSOPHILA MELANOGASTER (キイロショウジョウバエ) DROSOPHILA MELANOGASTER (キイロショウジョウバエ)発現宿主:   ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21 / Variant (発現宿主): CODONPLUS / 参照: UniProt: P17210 ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21 / Variant (発現宿主): CODONPLUS / 参照: UniProt: P17210#2: タンパク質・ペプチド |  キネシン / KINESIN キネシン / KINESIN分子量: 1913.098 Da / 分子数: 3 / 断片: TAIL DOMAIN RESIDUES 937-952 / 由来タイプ: 組換発現 由来: (組換発現)   DROSOPHILA MELANOGASTER (キイロショウジョウバエ) DROSOPHILA MELANOGASTER (キイロショウジョウバエ)発現宿主:   ESCHERICHIA COLI (大腸菌) / 参照: UniProt: P17210 ESCHERICHIA COLI (大腸菌) / 参照: UniProt: P17210#3: 化合物 | ChemComp-ADP /  アデノシン二リン酸 アデノシン二リン酸#4: 化合物 | ChemComp-MG / #5: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.15 Å3/Da / 溶媒含有率: 42.87 % / 解説: NONE |
|---|---|
結晶化 | pH: 7.4 詳細: 18% POLYETHYLENE GLYCOL-3350, 0.2M POTASSIUM CHLORIDE, 0.1M HEPES SODIUM PH 7.4 |
-データ収集
| 回折 | 平均測定温度: 93 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X06SA / 波長: 1 / ビームライン: X06SA / 波長: 1 |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2010年10月16日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.2→30 Å / Num. obs: 80517 / % possible obs: 99.9 % / Observed criterion σ(I): 2 / 冗長度: 8.8 % / Biso Wilson estimate: 41.383 Å2 / Rmerge(I) obs: 0.06 / Net I/σ(I): 21.7 |
| 反射 シェル | 解像度: 2.2→2.32 Å / 冗長度: 8.3 % / Rmerge(I) obs: 0.37 / Mean I/σ(I) obs: 5.5 / % possible all: 99.6 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 2Y5W 解像度: 2.2→50 Å / Cor.coef. Fo:Fc: 0.955 / Cor.coef. Fo:Fc free: 0.924 / SU B: 5.611 / SU ML: 0.147 / 交差検証法: THROUGHOUT / ESU R: 0.276 / ESU R Free: 0.221 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. U VALUES REFINED INDIVIDUALLY. PSEUDOSYMMETRY IN THE CRYSTAL STRUCTURE, WHERE CHAINS X AND Y LIE ON THE TWO-FOLD CRYSTALLOGRAPHIC SYMMETRY AXIS.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 42.405 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.2→50 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj








