| 登録情報 | データベース: PDB / ID: 2kft
|
|---|
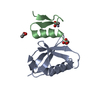
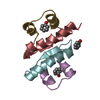
| タイトル | NMR Solution structure of the first PHD finger domain of human Autoimmune Regulator (AIRE) in complex with Histone H3(1-20Cys) Peptide |
|---|
 要素 要素 | - Autoimmune regulator
 - Histone H3
 ヒストンH3 ヒストンH3
|
|---|
 キーワード キーワード | TRANSCRIPTION/PROTEIN BINDING /  PHD Finger / PHD Finger /  Histone Code / AIRE / Histone Code / AIRE /  APECED / APECED /  Transcription (転写 (生物学)) / Transcription (転写 (生物学)) /  Alternative splicing (選択的スプライシング) / Alternative splicing (選択的スプライシング) /  Cytoplasm (細胞質) / Disease mutation / DNA-binding / Metal-binding / Cytoplasm (細胞質) / Disease mutation / DNA-binding / Metal-binding /  Nucleus (Nucleus) / Nucleus (Nucleus) /  Phosphoprotein / Polymorphism / Phosphoprotein / Polymorphism /  Transcription regulation / Transcription regulation /  Zinc (亜鉛) / Zinc (亜鉛) /  Zinc-finger (ジンクフィンガー) / Zinc-finger (ジンクフィンガー) /  Chromosomal protein (染色体) / Nucleosome core / TRANSCRIPTION-PROTEIN BINDING COMPLEX Chromosomal protein (染色体) / Nucleosome core / TRANSCRIPTION-PROTEIN BINDING COMPLEX |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
peripheral T cell tolerance induction / central tolerance induction to self antigen / regulation of thymocyte migration / thymus epithelium morphogenesis / negative thymic T cell selection / female germ cell nucleus /  液性免疫 / translation regulator activity / positive regulation of chemokine production / male germ cell nucleus ...peripheral T cell tolerance induction / central tolerance induction to self antigen / regulation of thymocyte migration / thymus epithelium morphogenesis / negative thymic T cell selection / female germ cell nucleus / 液性免疫 / translation regulator activity / positive regulation of chemokine production / male germ cell nucleus ...peripheral T cell tolerance induction / central tolerance induction to self antigen / regulation of thymocyte migration / thymus epithelium morphogenesis / negative thymic T cell selection / female germ cell nucleus /  液性免疫 / translation regulator activity / positive regulation of chemokine production / male germ cell nucleus / RNA polymerase II transcription regulatory region sequence-specific DNA binding / 液性免疫 / translation regulator activity / positive regulation of chemokine production / male germ cell nucleus / RNA polymerase II transcription regulatory region sequence-specific DNA binding /  histone binding / transcription by RNA polymerase II / histone binding / transcription by RNA polymerase II /  nuclear body / nuclear body /  免疫応答 / 免疫応答 /  chromatin binding / regulation of DNA-templated transcription / positive regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II / zinc ion binding / identical protein binding / chromatin binding / regulation of DNA-templated transcription / positive regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II / zinc ion binding / identical protein binding /  細胞核 / 細胞核 /  細胞質類似検索 - 分子機能 細胞質類似検索 - 分子機能  Autoimmune regulator, AIRE / AIRE, PHD finger 2 / HSR domain / Autoimmune regulator, AIRE / AIRE, PHD finger 2 / HSR domain /  Nuclear body protein Sp110/Sp140/Sp140L / HSR domain / HSR domain profile. / Nuclear body protein Sp110/Sp140/Sp140L / HSR domain / HSR domain profile. /  SAND domain / SAND domain /  SAND domain / SAND domain profile. / SAND domain / SAND domain profile. /  SAND domain ... SAND domain ... Autoimmune regulator, AIRE / AIRE, PHD finger 2 / HSR domain / Autoimmune regulator, AIRE / AIRE, PHD finger 2 / HSR domain /  Nuclear body protein Sp110/Sp140/Sp140L / HSR domain / HSR domain profile. / Nuclear body protein Sp110/Sp140/Sp140L / HSR domain / HSR domain profile. /  SAND domain / SAND domain /  SAND domain / SAND domain profile. / SAND domain / SAND domain profile. /  SAND domain / SAND-like domain superfamily / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 / SAND domain / SAND-like domain superfamily / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 /  Zinc finger, PHD-type, conserved site / PHD-finger / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. / Zinc finger, PHD-type, conserved site / PHD-finger / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. /  Zinc finger, PHD-finger / Zinc finger, PHD-finger /  Zinc finger, PHD-type / PHD zinc finger / Zinc finger, PHD-type / PHD zinc finger /  Zinc finger, FYVE/PHD-type / Zinc finger, FYVE/PHD-type /  Zinc finger, RING/FYVE/PHD-type / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 Zinc finger, RING/FYVE/PHD-type / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  溶液NMR / 溶液NMR /  simulated annealing simulated annealing |
|---|
| Model details | lowest energy, model 20 |
|---|
 データ登録者 データ登録者 | Chakravarty, S. / Zeng, L. / Zhou, M. |
|---|
 引用 引用 |  ジャーナル: Structure / 年: 2009 ジャーナル: Structure / 年: 2009
タイトル: Structure and Site-Specific Recognition of Histone H3 by the PHD Finger of Human Autoimmune Regulator.
著者: Chakravarty, S. / Zeng, L. / Zhou, M.M. |
|---|
| 履歴 | | 登録 | 2009年2月27日 | 登録サイト: BMRB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2009年4月28日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2022年3月16日 | Group: Database references / Derived calculations
カテゴリ: database_2 / pdbx_struct_assembly ...database_2 / pdbx_struct_assembly / pdbx_struct_oper_list / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード PHD Finger /
PHD Finger /  Histone Code / AIRE /
Histone Code / AIRE /  APECED /
APECED /  Transcription (転写 (生物学)) /
Transcription (転写 (生物学)) /  Alternative splicing (選択的スプライシング) /
Alternative splicing (選択的スプライシング) /  Cytoplasm (細胞質) / Disease mutation / DNA-binding / Metal-binding /
Cytoplasm (細胞質) / Disease mutation / DNA-binding / Metal-binding /  Nucleus (Nucleus) /
Nucleus (Nucleus) /  Phosphoprotein / Polymorphism /
Phosphoprotein / Polymorphism /  Transcription regulation /
Transcription regulation /  Zinc (亜鉛) /
Zinc (亜鉛) /  Zinc-finger (ジンクフィンガー) /
Zinc-finger (ジンクフィンガー) /  Chromosomal protein (染色体) / Nucleosome core / TRANSCRIPTION-PROTEIN BINDING COMPLEX
Chromosomal protein (染色体) / Nucleosome core / TRANSCRIPTION-PROTEIN BINDING COMPLEX 機能・相同性情報
機能・相同性情報 液性免疫 / translation regulator activity / positive regulation of chemokine production / male germ cell nucleus ...peripheral T cell tolerance induction / central tolerance induction to self antigen / regulation of thymocyte migration / thymus epithelium morphogenesis / negative thymic T cell selection / female germ cell nucleus /
液性免疫 / translation regulator activity / positive regulation of chemokine production / male germ cell nucleus ...peripheral T cell tolerance induction / central tolerance induction to self antigen / regulation of thymocyte migration / thymus epithelium morphogenesis / negative thymic T cell selection / female germ cell nucleus /  液性免疫 / translation regulator activity / positive regulation of chemokine production / male germ cell nucleus / RNA polymerase II transcription regulatory region sequence-specific DNA binding /
液性免疫 / translation regulator activity / positive regulation of chemokine production / male germ cell nucleus / RNA polymerase II transcription regulatory region sequence-specific DNA binding /  histone binding / transcription by RNA polymerase II /
histone binding / transcription by RNA polymerase II /  nuclear body /
nuclear body /  免疫応答 /
免疫応答 /  chromatin binding / regulation of DNA-templated transcription / positive regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II / zinc ion binding / identical protein binding /
chromatin binding / regulation of DNA-templated transcription / positive regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II / zinc ion binding / identical protein binding /  細胞核 /
細胞核 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR /
溶液NMR /  simulated annealing
simulated annealing  データ登録者
データ登録者 引用
引用 ジャーナル: Structure / 年: 2009
ジャーナル: Structure / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2kft.cif.gz
2kft.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2kft.ent.gz
pdb2kft.ent.gz PDB形式
PDB形式 2kft.json.gz
2kft.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/kf/2kft
https://data.pdbj.org/pub/pdb/validation_reports/kf/2kft ftp://data.pdbj.org/pub/pdb/validation_reports/kf/2kft
ftp://data.pdbj.org/pub/pdb/validation_reports/kf/2kft リンク
リンク 集合体
集合体
 要素
要素 / Autoimmune polyendocrinopathy candidiasis ectodermal dystrophy protein / APECED protein
/ Autoimmune polyendocrinopathy candidiasis ectodermal dystrophy protein / APECED protein
 Homo sapiens (ヒト) / 遺伝子: AIRE, APECED / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: AIRE, APECED / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: O43918
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: O43918 ヒストンH3
ヒストンH3 溶液NMR
溶液NMR 試料調製
試料調製 : DRX / 磁場強度: 800 MHz
: DRX / 磁場強度: 800 MHz 解析
解析 simulated annealing / ソフトェア番号: 1
simulated annealing / ソフトェア番号: 1  ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj

