| 登録情報 | データベース: PDB / ID: 2gku
|
|---|
| タイトル | Monomeric human telomere DNA tetraplex with 3+1 strand fold topology, two edgewise loops and double-chain reversal loop, NMR, 12 structures |
|---|
 要素 要素 | Human telomere DNA |
|---|
 キーワード キーワード |  DNA (デオキシリボ核酸) / G-TETRAD / 3+1 STRAND FOLDING / DNA (デオキシリボ核酸) / G-TETRAD / 3+1 STRAND FOLDING /  EDGEWISE / DOUBLE-CHAIN REVERSAL LOOPS / EDGEWISE / DOUBLE-CHAIN REVERSAL LOOPS /  NMR (核磁気共鳴) / 12 STRUCTURES NMR (核磁気共鳴) / 12 STRUCTURES |
|---|
| 機能・相同性 |  デオキシリボ核酸 / DNA (> 10) デオキシリボ核酸 / DNA (> 10) 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  溶液NMR / TORSION ANGLE DYNAMICS CARTESIAN DYNAMICS MATRIX RELAXATION 溶液NMR / TORSION ANGLE DYNAMICS CARTESIAN DYNAMICS MATRIX RELAXATION |
|---|
 データ登録者 データ登録者 | Luu, K.N. / Phan, A.T. / Kuryavyi, V.V. / Lacroix, L. / Patel, D.J. |
|---|
 引用 引用 |  ジャーナル: J.Am.Chem.Soc. / 年: 2006 ジャーナル: J.Am.Chem.Soc. / 年: 2006
タイトル: Structure of the human telomere in k(+) solution: an intramolecular (3 + 1) g-quadruplex scaffold.
著者: Luu, K.N. / Phan, A.T. / Kuryavyi, V.V. / Lacroix, L. / Patel, D.J. |
|---|
| 履歴 | | 登録 | 2006年4月3日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2006年8月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年5月1日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2022年3月9日 | Group: Data collection / Database references / Derived calculations
カテゴリ: database_2 / pdbx_nmr_software ...database_2 / pdbx_nmr_software / pdbx_nmr_spectrometer / pdbx_struct_assembly / pdbx_struct_oper_list
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード DNA (デオキシリボ核酸) / G-TETRAD / 3+1 STRAND FOLDING /
DNA (デオキシリボ核酸) / G-TETRAD / 3+1 STRAND FOLDING /  EDGEWISE / DOUBLE-CHAIN REVERSAL LOOPS /
EDGEWISE / DOUBLE-CHAIN REVERSAL LOOPS /  NMR (核磁気共鳴) / 12 STRUCTURES
NMR (核磁気共鳴) / 12 STRUCTURES デオキシリボ核酸 / DNA (> 10)
デオキシリボ核酸 / DNA (> 10) 機能・相同性情報
機能・相同性情報
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR / TORSION ANGLE DYNAMICS CARTESIAN DYNAMICS MATRIX RELAXATION
溶液NMR / TORSION ANGLE DYNAMICS CARTESIAN DYNAMICS MATRIX RELAXATION  データ登録者
データ登録者 引用
引用 ジャーナル: J.Am.Chem.Soc. / 年: 2006
ジャーナル: J.Am.Chem.Soc. / 年: 2006 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2gku.cif.gz
2gku.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2gku.ent.gz
pdb2gku.ent.gz PDB形式
PDB形式 2gku.json.gz
2gku.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/gk/2gku
https://data.pdbj.org/pub/pdb/validation_reports/gk/2gku ftp://data.pdbj.org/pub/pdb/validation_reports/gk/2gku
ftp://data.pdbj.org/pub/pdb/validation_reports/gk/2gku リンク
リンク 集合体
集合体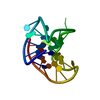
 要素
要素
 Homo sapiens (ヒト) / 発現宿主:
Homo sapiens (ヒト) / 発現宿主: 
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) 溶液NMR
溶液NMR 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj






































