+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2b6g | ||||||
|---|---|---|---|---|---|---|---|
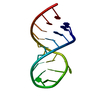
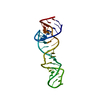
| タイトル | RNA recognition by the Vts1 SAM domain | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  RNA BINDING PROTEIN (RNA結合タンパク質) / RNA BINDING PROTEIN (RNA結合タンパク質) /  alpha-helix (Αヘリックス) / pentaloop / hairpin alpha-helix (Αヘリックス) / pentaloop / hairpin | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報flap-structured DNA binding / nuclear-transcribed mRNA poly(A) tail shortening / positive regulation of DNA metabolic process / nuclear-transcribed mRNA catabolic process /  P-body / P-body /  protein transport / protein transport /  nucleotide binding / nucleotide binding /  mRNA binding / mRNA binding /  RNA binding / RNA binding /  細胞核 / 細胞核 /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | ||||||
| 手法 |  溶液NMR / 溶液NMR /  simulated annealing simulated annealing | ||||||
 データ登録者 データ登録者 | Donaldson, L.W. / Johnson, P.E. | ||||||
 引用 引用 |  ジャーナル: Nat.Struct.Mol.Biol. / 年: 2006 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2006タイトル: RNA recognition by the Vts1p SAM domain 著者: Johnson, P.E. / Donaldson, L.W. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2b6g.cif.gz 2b6g.cif.gz | 48.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2b6g.ent.gz pdb2b6g.ent.gz | 36.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2b6g.json.gz 2b6g.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b6/2b6g https://data.pdbj.org/pub/pdb/validation_reports/b6/2b6g ftp://data.pdbj.org/pub/pdb/validation_reports/b6/2b6g ftp://data.pdbj.org/pub/pdb/validation_reports/b6/2b6g | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
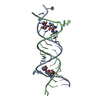
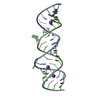
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 6086.633 Da / 分子数: 1 / 断片: Smaug Recognition Element / 由来タイプ: 合成 詳細: RNA was produced by T7 polymerase based in vitro transcription |
|---|---|
| #2: タンパク質 | 分子量: 13096.857 Da / 分子数: 1 / 断片: SAM domain / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: Vts1p / プラスミド: pGEX / 生物種 (発現宿主): Escherichia coli / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照:  GenBank: 6324935, UniProt: Q08831*PLUS GenBank: 6324935, UniProt: Q08831*PLUS |
-実験情報
-実験
| 実験 | 手法:  溶液NMR 溶液NMR | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | 内容: 0.2-0.8 mM U-15N,13C; 20 mM sodium phosphate buffer, pH 7.8, 150 mM sodium chloride, 0.02 % sodium azide 溶媒系: 90% H2O/10% D2O |
|---|---|
| 試料状態 | イオン強度: 150 mM NaCl / pH: 7.8 / 圧: ambient / 温度: 293 K |
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射波長 | 相対比: 1 | ||||||||||||||||||||
| NMRスペクトロメーター |
|
- 解析
解析
| NMR software |
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法:  simulated annealing / ソフトェア番号: 1 simulated annealing / ソフトェア番号: 1 詳細: Protein NOE restraints were calibrated from peak volumes to distances ranging from 1.8-5.0 using the CANDID module of CYANA v2.1. Initially, 100 structures were calculated with XPLOR-NIH v2. ...詳細: Protein NOE restraints were calibrated from peak volumes to distances ranging from 1.8-5.0 using the CANDID module of CYANA v2.1. Initially, 100 structures were calculated with XPLOR-NIH v2.11.0 starting from a partially docked protein-RNA complex. A simulated annealing approach with internal torsion angle dynamics, delphic database potentials and RNA planarity restraints was used. From the initial ensemble of structures, 20 were selected based upon low energy, no NOE violations > 0.5 angstroms and no dihedral angle violations > 5 degrees. Restraints used for the structure calculation: 882 intraresidue protein-protein NOE, 435 sequential protein-protein NOE, 335 medium range protein-protein NOE, 263 long range protein-protein NOE, 80 protein hydrogen bonds, 60 RNA-RNA NOE, 23 protein-RNA NOE, 140 protein dihedral angle, 93 RNA dihedral angle. | ||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj





























