+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1914 | ||||||
|---|---|---|---|---|---|---|---|
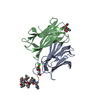
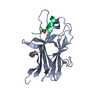
| タイトル | SIGNAL RECOGNITION PARTICLE ALU RNA BINDING HETERODIMER, SRP9/14 | ||||||
 要素 要素 | SIGNAL RECOGNITION PARTICLE 9/14 FUSION PROTEIN | ||||||
 キーワード キーワード | ALU DOMAIN /  RNA BINDING (リボ核酸) / SIGNAL RECOGNITION PARTICLE (SRP) / TRANSLATION REGULATION RNA BINDING (リボ核酸) / SIGNAL RECOGNITION PARTICLE (SRP) / TRANSLATION REGULATION | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報SRP-dependent cotranslational protein targeting to membrane / endoplasmic reticulum signal peptide binding /  signal recognition particle, endoplasmic reticulum targeting / response to xenobiotic stimulus => GO:0009410 / protein targeting to ER / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / Neutrophil degranulation / signal recognition particle, endoplasmic reticulum targeting / response to xenobiotic stimulus => GO:0009410 / protein targeting to ER / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / Neutrophil degranulation /  細胞核 細胞核類似検索 - 分子機能 | ||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多重同系置換 / 解像度: 2.53 Å 多重同系置換 / 解像度: 2.53 Å | ||||||
 データ登録者 データ登録者 | Birse, D. / Kapp, U. / Strub, K. / Cusack, S. / Aberg, A. | ||||||
 引用 引用 |  ジャーナル: EMBO J. / 年: 1997 ジャーナル: EMBO J. / 年: 1997タイトル: The crystal structure of the signal recognition particle Alu RNA binding heterodimer, SRP9/14. 著者: Birse, D.E. / Kapp, U. / Strub, K. / Cusack, S. / Aberg, A. #1:  ジャーナル: FEBS Lett. / 年: 1996 ジャーナル: FEBS Lett. / 年: 1996タイトル: Crystallization and Preliminary Crystallographic Analysis of the Signal Recognition Particle Srpphi14-9 Fusion Protein 著者: Birse, D.E. / Doublie, S. / Kapp, U. / Strub, K. / Cusack, S. / Aberg, A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1914.cif.gz 1914.cif.gz | 45.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1914.ent.gz pdb1914.ent.gz | 35.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1914.json.gz 1914.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/91/1914 https://data.pdbj.org/pub/pdb/validation_reports/91/1914 ftp://data.pdbj.org/pub/pdb/validation_reports/91/1914 ftp://data.pdbj.org/pub/pdb/validation_reports/91/1914 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 26389.631 Da / 分子数: 1 / 断片: ALU RNA BINDING HETERODIMER / 由来タイプ: 組換発現 / 由来: (組換発現)   Mus musculus (ハツカネズミ) / 細胞株: BL21 PLYSS / 細胞内の位置: CYTOPLASM Mus musculus (ハツカネズミ) / 細胞株: BL21 PLYSS / 細胞内の位置: CYTOPLASM 細胞質 / プラスミド: PET3C / 発現宿主: 細胞質 / プラスミド: PET3C / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P16254 Escherichia coli (大腸菌) / 参照: UniProt: P16254 |
|---|---|
| #2: 化合物 | ChemComp-PO4 /  リン酸塩 リン酸塩 |
| #3: 化合物 | ChemComp-BME /  2-メルカプトエタノール 2-メルカプトエタノール |
| #4: 水 | ChemComp-HOH /  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 10 X線回折 / 使用した結晶の数: 10 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3 Å3/Da / 溶媒含有率: 34 % 解説: DATA WERE COLLECTED ON SELENIUM, MERCURY AND PLATINUM DERIVATIVES TO SOLVE MIR STRUCTURE | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | 温度: 277 K / 手法: ハンギングドロップ法 / pH: 7.7 詳細: THE SRPPHI14-9 PROTEIN WAS CRYSTALLIZED (BIRSE ET AL., FEBS LETTERS, 1996) BY THE HANGING DROP METHOD IN 2.0 M NAH2/K2H PO4, PH 7.7, 2% MPD, 1.0 MM NAN3 AT 4 DEGREES C WITH A FINAL PROTEIN ...詳細: THE SRPPHI14-9 PROTEIN WAS CRYSTALLIZED (BIRSE ET AL., FEBS LETTERS, 1996) BY THE HANGING DROP METHOD IN 2.0 M NAH2/K2H PO4, PH 7.7, 2% MPD, 1.0 MM NAN3 AT 4 DEGREES C WITH A FINAL PROTEIN CONCENTRATION OF 5-8 MG ML-1. CRYSTALS FORMED OVER 2-3 WEEKS AND WERE TYPICALLY 150 X 150 X 300 MM3 IN SPACE, hanging drop, temperature 277K | ||||||||||||||||||||||||||||||
| 結晶 | *PLUS | ||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 4 ℃ / 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID2 / 波長: 0.9 / 波長: 0.900, 1.100 / ビームライン: ID2 / 波長: 0.9 / 波長: 0.900, 1.100 | |||||||||
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1996年9月1日 / 詳細: TOROIDAL MIRRORS | |||||||||
| 放射 | モノクロメーター: CU FILTER / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||
| 放射波長 |
| |||||||||
| 反射 | 解像度: 2.53→30 Å / Num. obs: 36596 / % possible obs: 98.7 % / Observed criterion σ(I): 3 / 冗長度: 5 % / Rmerge(I) obs: 0.069 / Rsym value: 0.069 / Net I/σ(I): 5.8 | |||||||||
| 反射 シェル | 解像度: 2.53→2.6 Å / 冗長度: 4.2 % / Rmerge(I) obs: 0.0218 / Mean I/σ(I) obs: 2.4 / Rsym value: 0.0218 / % possible all: 97.2 | |||||||||
| 反射 | *PLUS Num. obs: 7634 / Num. measured all: 36956 | |||||||||
| 反射 シェル | *PLUS 最低解像度: 2.64 Å / % possible obs: 97.2 % |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  多重同系置換 / 解像度: 2.53→20 Å / Data cutoff high absF: 10000000 / Data cutoff low absF: 0.001 / 交差検証法: THROUGHOUT / σ(F): 2 多重同系置換 / 解像度: 2.53→20 Å / Data cutoff high absF: 10000000 / Data cutoff low absF: 0.001 / 交差検証法: THROUGHOUT / σ(F): 2 詳細: THE MODEL INCLUDES 77 RESIDUES OF SRP9 (4-81) AND 84 RESIDUES OF SRP14 (1-34) AND (47-97). THE SRP14 LOOP ELECTRON DENSITY CONNECTING 14B1-14B2 (34-47) IS WEAK SUGGESTING THAT THE LOOP IS ...詳細: THE MODEL INCLUDES 77 RESIDUES OF SRP9 (4-81) AND 84 RESIDUES OF SRP14 (1-34) AND (47-97). THE SRP14 LOOP ELECTRON DENSITY CONNECTING 14B1-14B2 (34-47) IS WEAK SUGGESTING THAT THE LOOP IS FLEXIBLE. 5 C-TERMINAL AND 3 N-TERMINAL RESIDUES OF SRP9 AND 13 C-TERMINAL RESIDUES OF SRP14 HAVE POORLY DEFINED DENSITY. IN ADDITION, THE MODEL DOES NOT INCLUDE THE SRPF14-9 N-TERMINAL F EXTENSION (18 RESIDUES) NOR THE ENTIRE LINKER REGION OF WHICH ONLY 8 OF 17 RESIDUES ARE ORDERED.
| |||||||||||||||||||||
| Refine analyze | Luzzati d res low obs: 20 Å | |||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.53→20 Å
| |||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.53→2.64 Å / % reflection obs: 97.2 % | |||||||||||||||||||||
| Xplor file |
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj















