+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9002 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-EM structure of human TRPML1 with ML-SA1 and PI35P2 | |||||||||
 マップデータ マップデータ | primary map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of lysosome organization / iron ion transmembrane transporter activity / calcium ion export / intracellularly phosphatidylinositol-3,5-bisphosphate-gated monatomic cation channel activity / NAADP-sensitive calcium-release channel activity / phagosome maturation / transferrin transport / Transferrin endocytosis and recycling / cellular response to pH / ligand-gated calcium channel activity ...positive regulation of lysosome organization / iron ion transmembrane transporter activity / calcium ion export / intracellularly phosphatidylinositol-3,5-bisphosphate-gated monatomic cation channel activity / NAADP-sensitive calcium-release channel activity / phagosome maturation / transferrin transport / Transferrin endocytosis and recycling / cellular response to pH / ligand-gated calcium channel activity /  TRPチャネル / phagocytic cup / autophagosome maturation / monoatomic cation transport / localization / monoatomic cation channel activity / release of sequestered calcium ion into cytosol / cellular response to calcium ion / cell projection / calcium ion transmembrane transport / TRPチャネル / phagocytic cup / autophagosome maturation / monoatomic cation transport / localization / monoatomic cation channel activity / release of sequestered calcium ion into cytosol / cellular response to calcium ion / cell projection / calcium ion transmembrane transport /  calcium channel activity / phagocytic vesicle membrane / late endosome / late endosome membrane / protein homotetramerization / calcium channel activity / phagocytic vesicle membrane / late endosome / late endosome membrane / protein homotetramerization /  獲得免疫系 / 獲得免疫系 /  リソソーム / リソソーム /  receptor complex / endosome membrane / lysosomal membrane / intracellular membrane-bounded organelle / receptor complex / endosome membrane / lysosomal membrane / intracellular membrane-bounded organelle /  lipid binding / lipid binding /  ゴルジ体 / ゴルジ体 /  核質 / 核質 /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.73 Å クライオ電子顕微鏡法 / 解像度: 3.73 Å | |||||||||
 データ登録者 データ登録者 | Schmiege P / Li X | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2018 ジャーナル: Nat Commun / 年: 2018タイトル: Structural basis for PtdInsP-mediated human TRPML1 regulation. 著者: Michael Fine / Philip Schmiege / Xiaochun Li /  要旨: Transient receptor potential mucolipin 1 (TRPML1), a lysosomal channel, maintains the low pH and calcium levels for lysosomal function. Several small molecules modulate TRPML1 activity. ML-SA1, a ...Transient receptor potential mucolipin 1 (TRPML1), a lysosomal channel, maintains the low pH and calcium levels for lysosomal function. Several small molecules modulate TRPML1 activity. ML-SA1, a synthetic agonist, binds to the pore region and phosphatidylinositol-3,5-bisphosphate (PtdIns(3,5)P), a natural lipid, stimulates channel activity to a lesser extent than ML-SA1; moreover, PtdIns(4,5)P, another natural lipid, prevents TRPML1-mediated calcium release. Notably, PtdIns(3,5)P and ML-SA1 cooperate further increasing calcium efflux. Here we report the structures of human TRPML1 at pH 5.0 with PtdIns(3,5)P, PtdIns(4,5)P, or ML-SA1 and PtdIns(3,5)P, revealing a unique lipid-binding site. PtdIns(3,5)P and PtdIns(4,5)P bind to the extended helices of S1, S2, and S3. The phosphate group of PtdIns(3,5)P induces Y355 to form a π-cation interaction with R403, moving the S4-S5 linker, thus allosterically activating the channel. Our structures and electrophysiological characterizations reveal an allosteric site and provide molecular insight into how lipids regulate TRP channels. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9002.map.gz emd_9002.map.gz | 87.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9002-v30.xml emd-9002-v30.xml emd-9002.xml emd-9002.xml | 10.5 KB 10.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9002.png emd_9002.png | 65.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9002 http://ftp.pdbj.org/pub/emdb/structures/EMD-9002 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9002 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9002 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9002.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9002.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | primary map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TRPML1
| 全体 | 名称: TRPML1 MCOLN1 MCOLN1 |
|---|---|
| 要素 |
|
-超分子 #1: TRPML1
| 超分子 | 名称: TRPML1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Mucolipin-1
| 分子 | 名称: Mucolipin-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 65.084996 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MTAPAGPRGS ETERLLTPNP GYGTQAGPSP APPTPPEEED LRRRLKYFFM SPCDKFRAKG RKPCKLMLQV VKILVVTVQL ILFGLSNQL AVTFREENTI AFRHLFLLGY SDGADDTFAA YTREQLYQAI FHAVDQYLAL PDVSLGRYAY VRGGGDPWTN G SGLALCQR ...文字列: MTAPAGPRGS ETERLLTPNP GYGTQAGPSP APPTPPEEED LRRRLKYFFM SPCDKFRAKG RKPCKLMLQV VKILVVTVQL ILFGLSNQL AVTFREENTI AFRHLFLLGY SDGADDTFAA YTREQLYQAI FHAVDQYLAL PDVSLGRYAY VRGGGDPWTN G SGLALCQR YYHRGHVDPA NDTFDIDPMV VTDCIQVDPP ERPPPPPSDD LTLLESSSSY KNLTLKFHKL VNVTIHFRLK TI NLQSLIN NEIPDCYTFS VLITFDNKAH SGRIPISLET QAHIQECKHP SVFQHGDNSF RLLFDVVVIL TCSLSFLLCA RSL LRGFLL QNEFVGFMWR QRGRVISLWE RLEFVNGWYI LLVTSDVLTI SGTIMKIGIE AKNLASYDVC SILLGTSTLL VWVG VIRYL TFFHNYNILI ATLRVALPSV MRFCCCVAVI YLGYCFCGWI VLGPYHVKFR SLSMVSECLF SLINGDDMFV TFAAM QAQQ GRSSLVWLFS QLYLYSFISL FIYMVLSLFI ALITGAYDTI KHPGGAGAEE SELQAYIAQC QDSPTSGKFR RGSGSA CSL LCCCGRDPSE EHSLLVN |
-分子 #2: (1R,2S,3S,4R,5S,6R)-5-{[(R)-[(2R)-2,3-bis{[(1S)-1-hydroxyoctyl]ox...
| 分子 | 名称: (1R,2S,3S,4R,5S,6R)-5-{[(R)-[(2R)-2,3-bis{[(1S)-1-hydroxyoctyl]oxy}propoxy](hydroxy)phosphoryl]oxy}-2,4,6-trihydroxycyclohexane-1,3-diyl bis[dihydrogen (phosphate)] タイプ: ligand / ID: 2 / コピー数: 4 / 式: HZ7 |
|---|---|
| 分子量 | 理論値: 750.598 Da |
| Chemical component information | 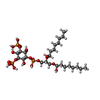 ChemComp-HZ7: |
-分子 #3: 2-{2-oxo-2-[(4S)-2,2,4-trimethyl-3,4-dihydroquinolin-1(2H)-yl]eth...
| 分子 | 名称: 2-{2-oxo-2-[(4S)-2,2,4-trimethyl-3,4-dihydroquinolin-1(2H)-yl]ethyl}-1H-isoindole-1,3(2H)-dione タイプ: ligand / ID: 3 / コピー数: 4 / 式: AQV |
|---|---|
| 分子量 | 理論値: 362.422 Da |
| Chemical component information | 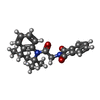 ChemComp-AQV: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 7 mg/mL |
|---|---|
| 緩衝液 | pH: 7 |
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 1.6 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: CTFFIND |
|---|---|
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.73 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: FREALIX / 使用した粒子像数: 45481 |
 ムービー
ムービー コントローラー
コントローラー

















