+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4576 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 2tRNAs-bound Elongator catalytic subcomplex, 2tRNA-Elp123. | |||||||||
 マップデータ マップデータ | Postprocessed map of the full Elongator catalytic subcomplex bound to 2 tRNAs, Elp123-2tRNAs, from yeast at 6.7A resolution. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
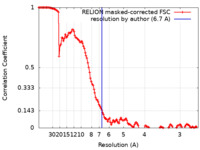
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.7 Å クライオ電子顕微鏡法 / 解像度: 6.7 Å | |||||||||
 データ登録者 データ登録者 | Dauden MI / Weis F | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2019 ジャーナル: Sci Adv / 年: 2019タイトル: Molecular basis of tRNA recognition by the Elongator complex. 著者: Maria I Dauden / Marcin Jaciuk / Felix Weis / Ting-Yu Lin / Carolin Kleindienst / Nour El Hana Abbassi / Heena Khatter / Rościsław Krutyhołowa / Karin D Breunig / Jan Kosinski / Christoph ...著者: Maria I Dauden / Marcin Jaciuk / Felix Weis / Ting-Yu Lin / Carolin Kleindienst / Nour El Hana Abbassi / Heena Khatter / Rościsław Krutyhołowa / Karin D Breunig / Jan Kosinski / Christoph W Müller / Sebastian Glatt /   要旨: The highly conserved Elongator complex modifies transfer RNAs (tRNAs) in their wobble base position, thereby regulating protein synthesis and ensuring proteome stability. The precise mechanisms of ...The highly conserved Elongator complex modifies transfer RNAs (tRNAs) in their wobble base position, thereby regulating protein synthesis and ensuring proteome stability. The precise mechanisms of tRNA recognition and its modification reaction remain elusive. Here, we show cryo-electron microscopy structures of the catalytic subcomplex of Elongator and its tRNA-bound state at resolutions of 3.3 and 4.4 Å. The structures resolve details of the catalytic site, including the substrate tRNA, the iron-sulfur cluster, and a SAM molecule, which are all validated by mutational analyses in vitro and in vivo. tRNA binding induces conformational rearrangements, which precisely position the targeted anticodon base in the active site. Our results provide the molecular basis for substrate recognition of Elongator, essential to understand its cellular function and role in neurodegenerative diseases and cancer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4576.map.gz emd_4576.map.gz | 9.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4576-v30.xml emd-4576-v30.xml emd-4576.xml emd-4576.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4576_fsc.xml emd_4576_fsc.xml | 13.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4576.png emd_4576.png | 316.1 KB | ||
| その他 |  emd_4576_half_map_1.map.gz emd_4576_half_map_1.map.gz emd_4576_half_map_2.map.gz emd_4576_half_map_2.map.gz | 148.7 MB 148.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4576 http://ftp.pdbj.org/pub/emdb/structures/EMD-4576 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4576 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4576 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4576.map.gz / 形式: CCP4 / 大きさ: 190.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4576.map.gz / 形式: CCP4 / 大きさ: 190.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed map of the full Elongator catalytic subcomplex bound to 2 tRNAs, Elp123-2tRNAs, from yeast at 6.7A resolution. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map of the full Elongator catalytic subcomplex...
| ファイル | emd_4576_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of the full Elongator catalytic subcomplex bound to 2 tRNAs, Elp123-2tRNAs, from yeast. | ||||||||||||
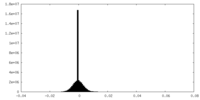
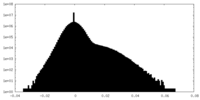
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of the full Elongator catalytic subcomplex...
| ファイル | emd_4576_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of the full Elongator catalytic subcomplex bound to 2 tRNAs, Elp123-2tRNAs, from yeast. | ||||||||||||
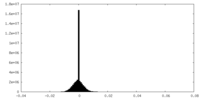
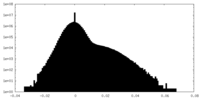
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 2tRNAs-bound Elongator catalytic subcomplex, 2tRNAs-Elp123.
| 全体 | 名称: 2tRNAs-bound Elongator catalytic subcomplex, 2tRNAs-Elp123. |
|---|---|
| 要素 |
|
-超分子 #1: 2tRNAs-bound Elongator catalytic subcomplex, 2tRNAs-Elp123.
| 超分子 | 名称: 2tRNAs-bound Elongator catalytic subcomplex, 2tRNAs-Elp123. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The EM map corresponds to the full Elp123 subcomplex bound to two tRNAs, one on each lobe. |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 665 KDa |
-分子 #1: Elongator complex protein 1, Elp1
| 分子 | 名称: Elongator complex protein 1, Elp1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 配列 | 文字列: MVEHDKSGSK RQELRSNMRN LITLNKGKFK PTASTAEGDE DDLSFTLLDS VFDTLSDSIT CVLGSTDIG AIEVQQFMKD GSRNVLASFN IQTFDDKLLS FVHFADINQL VFVFEQGDII T ATYDPVSL DPAETLIEIM GTIDNGIAAA QWSYDEETLA MVTKDRNVVV ...文字列: MVEHDKSGSK RQELRSNMRN LITLNKGKFK PTASTAEGDE DDLSFTLLDS VFDTLSDSIT CVLGSTDIG AIEVQQFMKD GSRNVLASFN IQTFDDKLLS FVHFADINQL VFVFEQGDII T ATYDPVSL DPAETLIEIM GTIDNGIAAA QWSYDEETLA MVTKDRNVVV LSKLFEPISE YH LEVDDLK ISKHVTVGWG KKETQFRGKG ARAMEREALA SLKASGLVGN QLRDPTMPYM VDT GDVTAL DSHEITISWR GDCDYFAVSS VEEVPDEDDE TKSIKRRAFR VFSREGQLDS ASEP VTGME HQLSWKPQGS LIASIQRKTD LGEEDSVDVI FFERNGLRHG EFDTRLPLDE KVESV CWNS NSEALAVVLA NRIQLWTSKN YHWYLKQELY ASDISYVKWH PEKDFTLMFS DAGFIN IVD FAYKMAQGPT LEPFDNGTSL VVDGRTVNIT PLALANVPPP MYYRDFETPG NVLDVAC SF SNEIYAAINK DVLIFAAVPS IEEMKKGKHP SIVCEFPKSE FTSEVDSLRQ VAFINDSI V GVLLDTDNLS RIALLDIQDI TQPTLITIVE VYDKIVLLRS DFDYNHLVYE TRDGTVCQL DAEGQLMEIT KFPQLVRDFR VKRVHNTSAE DDDNWSAESS ELVAFGITNN GKLFANQVLL ASAVTSLEI TDSFLLFTTA QHNLQFVHLN STDFKPLPLV EEGVEDERVR AIERGSILVS V IPSKSSVV LQATRGNLET IYPRIMVLAE VRKNIMAKRY KEAFIVCRTH RINLDILHDY AP ELFIENL EVFINQIGRV DYLNLFISCL SEDDVTKTKY KETLYSGISK SFGMEPAPLT EMQ IYMKKK MFDPKTSKVN KICDAVLNVL LSNPEYKKKY LQTIITAYAS QNPQNLSAAL KLIS ELENS EEKDSCVTYL CFLQDVNVVY KSALSLYDVS LALLVAQKSQ MDPREYLPFL QELQD NEPL RRKFLIDDYL GNYEKALEHL SEIDKDGNVS EEVIDYVESH DLYKHGLALY RYDSEK QNV IYNIYAKHLS SNQMYTDAAV AYEMLGKLKE AMGAYQSAKR WREAMSIAVQ KFPEEVE SV AEELISSLTF EHRYVDAADI QLEYLDNVKE AVALYCKAYR YDIASLVAIK AKKDELLE E VVDPGLGEGF GIIAELLADC KGQINSQLRR LRELRAKKEE NPYAFYGQET EQADDVSVA PSETSTQESF FTRYTGKTGG TAKTGASRRT AKNKRREERK RARGKKGTIY EEEYLVQSVG RLIERLNQT KPDAVRVVEG LCRRNMREQA HQIQKNFVEV LDLLKANVKE IYSISEKDRE R VNENGEVY YIPEIPVPEI HDFPKSHIVD F |
-分子 #2: Elongator complex protein 2, Elp2
| 分子 | 名称: Elongator complex protein 2, Elp2 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 配列 | 文字列: MVECITPEAI FIGANKQTQV SDIHKVKKIV AFGAGKTIAL WDPIEPNNKG VYATLKGHEA EVTCVRFVP DSDFMVSASE DHHVKIWKFT DYSHLQCIQT IQHYSKTIVA LSALPSLISV G CADGTISI WRQNIQNDEF GLAHEFTIKK GFFYPLCLSL SKVEEKKYLL ...文字列: MVECITPEAI FIGANKQTQV SDIHKVKKIV AFGAGKTIAL WDPIEPNNKG VYATLKGHEA EVTCVRFVP DSDFMVSASE DHHVKIWKFT DYSHLQCIQT IQHYSKTIVA LSALPSLISV G CADGTISI WRQNIQNDEF GLAHEFTIKK GFFYPLCLSL SKVEEKKYLL AIGGTNVNVF IA SFILSDS GIEKCRVVAE LEGHEDWVKS LAFRHQETPG DYLLCSGSQD RYIRLWRIRI NDL IDDSEE DSKKLTLLSN KQYKFQIDDE LRVGINFEAL IMGHDDWISS LQWHESRLQL LAAT ADTSL MVWEPDETSG IWVCSLRLGE MSSKGASTAT GSSGGFWSCL WFTHERMDFF LTNGK TGSW RMWATKDNII CDQRLGISGA TKDVTDIAWS PSGEYLLATS LDQTTRLFAP WIYDAS GRK REIATWHEFS RPQIHGYDMI CVETVTDTRF VSGGDEKILR SFDLPKGVAG MLQKFVG IQ FEEKSEMPDS ATVPVLGLSN KAGEDDANED DEEEEGGNKE TPDITDPLSL LECPPMED Q LQRHLLWPEV EKLYGHGFEI TCLDISPDQK LIASACRSNN VQNAVIRIFS TENWLEIKP ALPFHSLTIT RLKFSKDGKF LLSVCRDRKW ALWERNMEDN TFELRFKNEK PHTRIIWDAD WAPLEFGNV FVTASRDKTV KVWRHQKEPA DDYVLEASIK HTKAVTAISI HDSMIREKIL I SVGLENGE IYLYSYTLGK FELITQLNED ITPADKITRL RWSHLKRNGK LFLGVGSSDL ST RIYSLAY E |
-分子 #3: Elongator complex protein 3, Elp3
| 分子 | 名称: Elongator complex protein 3, Elp3 / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 配列 | 文字列: MARHGKGPKT NKKKLAPEKE RFIQCCADIT LELTDSLTSG TTREINLNGL ITKYSKKYKL KQQPRLTDI INSIPDQYKK YLLPKLKAKP VRTASGIAVV AVMCKPHRCP HIAYTGNICV Y CPGGPDSD FEYSTQSYTG YEPTSMRAIR ARYDPYEQAR GRVEQLKQLG ...文字列: MARHGKGPKT NKKKLAPEKE RFIQCCADIT LELTDSLTSG TTREINLNGL ITKYSKKYKL KQQPRLTDI INSIPDQYKK YLLPKLKAKP VRTASGIAVV AVMCKPHRCP HIAYTGNICV Y CPGGPDSD FEYSTQSYTG YEPTSMRAIR ARYDPYEQAR GRVEQLKQLG HSIDKVEYVL MG GTFMSLP KEYREDFIVK LHNALSGFNG NDIDEAILYS QQSLTKCVGI TIETRPDYCT QTH LDDMLK YGCTRLEIGV QSLYEDVARD TNRGHTVRSV CETFAVSKDA GYKVVSHMMP DLPN VGMER DIEQFKEYFE NPDFRTDGLK IYPTLVIRGT GLYELWKTGR YKSYSANALV DLVAR ILAL VPPWTRIYRV QRDIPMPLVT SGVDNGNLRE LALARMKDLG TTCRDVRTRE VGIQEV HHK VQPDQVELIR RDYYANGGWE TFLSYEDPKK DILIGLLRLR KASKKYTYRK EFTSQRT SI VRELHVYGSV VPLHSRDPRK FQHQGFGTLL MEEAERIAKE EHGSEKISVI SGVGVRNY Y GKLGYELDGP YMSKRI |
-分子 #4: tRNA Ala UGC
| 分子 | 名称: tRNA Ala UGC / タイプ: rna / ID: 4 |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 配列 | 文字列: GGGCACAUGG CGCAGUUGGU AGCGCGCUUC CCUUGCAAGG AAGAGGUCAU CGGUUCGAUU CCGGUUGCGU CCA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 詳細: Pelco EasyGlow glow discharger | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: 2.5 ul of sample, blotting parameters: wait time 15 s, blot force 5, blot time 5-8 s.. | |||||||||||||||||||||
| 詳細 | Pure Elp123 was incubated with in vitro transcribed tRNA Ala UGC at 1:5 molar ratio for 30 min at 25 degrees, and then immediately plunged. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 詳細 | Gatan Quantum energy filter and a K2 Summit direct detector |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 5412 / 平均電子線量: 39.8 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X