+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3747 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Poliovirus type 3 (strain Saukett) stabilized virus-like particle | |||||||||
 マップデータ マップデータ | MRC format map file. Contour level 0.0725 determined by visual inspection of map in UCSF Chimera. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報caveolin-mediated endocytosis of virus by host cell / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid ...caveolin-mediated endocytosis of virus by host cell / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity / picornain 3C / T=pseudo3 icosahedral viral capsid ...caveolin-mediated endocytosis of virus by host cell / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane /  カプシド / カプシド /  : / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity / : / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity /  RNA helicase activity / RNA helicase activity /  DNA複製 / induction by virus of host autophagy / DNA複製 / induction by virus of host autophagy /  RNA依存性RNAポリメラーゼ / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity / RNA依存性RNAポリメラーゼ / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity /  RNA-dependent RNA polymerase activity / DNA-templated transcription / host cell nucleus / virion attachment to host cell / structural molecule activity / RNA-dependent RNA polymerase activity / DNA-templated transcription / host cell nucleus / virion attachment to host cell / structural molecule activity /  タンパク質分解 / タンパク質分解 /  RNA binding / RNA binding /  ATP binding / ATP binding /  生体膜 / 生体膜 /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Human poliovirus 3 (ポリオウイルス) Human poliovirus 3 (ポリオウイルス) | |||||||||
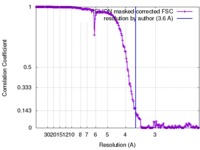
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Bahar MW / Kotecha A / Fry EE / Stuart DI | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: Plant-made polio type 3 stabilized VLPs-a candidate synthetic polio vaccine. 著者: Johanna Marsian / Helen Fox / Mohammad W Bahar / Abhay Kotecha / Elizabeth E Fry / David I Stuart / Andrew J Macadam / David J Rowlands / George P Lomonossoff /  要旨: Poliovirus (PV) is the causative agent of poliomyelitis, a crippling human disease known since antiquity. PV occurs in two distinct antigenic forms, D and C, of which only the D form elicits a robust ...Poliovirus (PV) is the causative agent of poliomyelitis, a crippling human disease known since antiquity. PV occurs in two distinct antigenic forms, D and C, of which only the D form elicits a robust neutralizing response. Developing a synthetically produced stabilized virus-like particle (sVLP)-based vaccine with D antigenicity, without the drawbacks of current vaccines, will be a major step towards the final eradication of poliovirus. Such a sVLP would retain the native antigenic conformation and the repetitive structure of the original virus particle, but lack infectious genomic material. In this study, we report the production of synthetically stabilized PV VLPs in plants. Mice carrying the gene for the human PV receptor are protected from wild-type PV when immunized with the plant-made PV sVLPs. Structural analysis of the stabilized mutant at 3.6 Å resolution by cryo-electron microscopy and single-particle reconstruction reveals a structure almost indistinguishable from wild-type PV3.Despite the success of current vaccination against poliomyelitis, safe, cheap and effective vaccines remain sought for continuing eradication effort. Here the authors use plants to express stabilized virus-like particles of type 3 poliovirus that can induce a protective immune response in mice transgenic for the human poliovirus receptor. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3747.map.gz emd_3747.map.gz | 472.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3747-v30.xml emd-3747-v30.xml emd-3747.xml emd-3747.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3747_fsc.xml emd_3747_fsc.xml | 17.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3747.png emd_3747.png | 328.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3747 http://ftp.pdbj.org/pub/emdb/structures/EMD-3747 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3747 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3747 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3747.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3747.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MRC format map file. Contour level 0.0725 determined by visual inspection of map in UCSF Chimera. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human poliovirus 3
| 全体 | 名称:   Human poliovirus 3 (ポリオウイルス) Human poliovirus 3 (ポリオウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Human poliovirus 3
| 超分子 | 名称: Human poliovirus 3 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Poliovirus type 3 (Saukett strain) virus-like particle produced in plant expression system. NCBI-ID: 12086 / 生物種: Human poliovirus 3 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:   Human poliovirus 3 (ポリオウイルス) / 株: Saukett Human poliovirus 3 (ポリオウイルス) / 株: Saukett |
| Host system | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ)組換プラスミド: pEAQ-HT |
| 分子量 | 理論値: 5.8 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Capsid / 直径: 327.0 Å / T番号(三角分割数): 1 |
-分子 #1: Capsid proteins, VP1
| 分子 | 名称: Capsid proteins, VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human poliovirus 3 (ポリオウイルス) / 株: Saukett Human poliovirus 3 (ポリオウイルス) / 株: Saukett |
| 分子量 | 理論値: 33.562785 KDa |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 配列 | 文字列: GIEDLITEVA QGALTLSLPK QQDSLPDTKA SGPAHSKEVP ALTAVETGAT NPLVPSDTVQ TRHVIQRRSR SESTIESFFA RGACVAIIE VDNEEPTTRA QKLFAMWRIT YKDTVQLRRK LEFFTYSRFD MELTFVVTAN FTNTNNGHAL NQVYQIMYIP P GAPTPKSW ...文字列: GIEDLITEVA QGALTLSLPK QQDSLPDTKA SGPAHSKEVP ALTAVETGAT NPLVPSDTVQ TRHVIQRRSR SESTIESFFA RGACVAIIE VDNEEPTTRA QKLFAMWRIT YKDTVQLRRK LEFFTYSRFD MELTFVVTAN FTNTNNGHAL NQVYQIMYIP P GAPTPKSW DDYTWQTSSN PSIFYTYGAA PARISVPYVG LANAYSHFYD GFAKVPLKTD ANDQIGDSLY SAMTVDDFGV LA IRVVNDH NPTKVTSKVR IYMKPKHVRV WCPRPPRAVP YYGPGVDYKD NLNPLSEKGL TTY |
-分子 #2: Capsid proteins, VP2
| 分子 | 名称: Capsid proteins, VP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human poliovirus 3 (ポリオウイルス) / 株: Saukett Human poliovirus 3 (ポリオウイルス) / 株: Saukett |
| 分子量 | 理論値: 30.188982 KDa |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 配列 | 文字列: SPNVEACGYS DRVLQLTIGN STITTQEAAN SVVAYGRWPE FIRDDEANPV DQPTEPDVAT CRFYTLDTVM WGKESKGWWW KLPDALRDM GLFGQNMYYH YLGRSGYTVH VQCNASKFHQ GALGVFAIPE YCLAGDSDKQ RYTSYANANP GEKGGKFYSQ F NRDTAVTS ...文字列: SPNVEACGYS DRVLQLTIGN STITTQEAAN SVVAYGRWPE FIRDDEANPV DQPTEPDVAT CRFYTLDTVM WGKESKGWWW KLPDALRDM GLFGQNMYYH YLGRSGYTVH VQCNASKFHQ GALGVFAIPE YCLAGDSDKQ RYTSYANANP GEKGGKFYSQ F NRDTAVTS PKREFCPVDY LLGCGVLLGN AFVYPHQIIN LRTNNSATIV LPYVNAMAID SMVKHNNWGI AILPLSPLDF AQ ESSVEIP ITVTIAPMCS EFNGLRNVTA PKFQ |
-分子 #3: Capsid proteins, VP3
| 分子 | 名称: Capsid proteins, VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human poliovirus 3 (ポリオウイルス) / 株: Saukett Human poliovirus 3 (ポリオウイルス) / 株: Saukett |
| 分子量 | 理論値: 26.3151 KDa |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 配列 | 文字列: GLPVLNTPGS NQYLTSDNYQ SPCAIPEFDV TPPIDIPGEV KNMMELAEID TMIPLNLENT KRNTMDMYRV TLSDSADLSQ PILCFSLSP ASDPRLSHTM LGEVLNYYTH WAGSLKFTFL FCGSMMATGK ILVAYAPPGA QPPTSRKEAM LGTHVIWDLG L QSSCTMVV ...文字列: GLPVLNTPGS NQYLTSDNYQ SPCAIPEFDV TPPIDIPGEV KNMMELAEID TMIPLNLENT KRNTMDMYRV TLSDSADLSQ PILCFSLSP ASDPRLSHTM LGEVLNYYTH WAGSLKFTFL FCGSMMATGK ILVAYAPPGA QPPTSRKEAM LGTHVIWDLG L QSSCTMVV PWISNVTYRQ TTQDSFTEGG YISMFYQTRI VVPLSTPKSM SMLGFVSACN DFSVRLLRDT THISQSALPQ |
-分子 #4: Capsid proteins, VP4
| 分子 | 名称: Capsid proteins, VP4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human poliovirus 3 (ポリオウイルス) / 株: Saukett Human poliovirus 3 (ポリオウイルス) / 株: Saukett |
| 分子量 | 理論値: 7.452113 KDa |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 配列 | 文字列: MGAQVSSQKV GAHENSNRAY GGSTINYTTI NYYKDSASNA ASKQDYSQDP SKFTEPLKDV LIKTAPALN |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7 / 詳細: PBS pH 7.0 |
| グリッド | モデル: C-flat CF-2/1-2C / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK I / 詳細: Blot for 4 seconds before plunging. |
| 詳細 | Virus-like particles for polio type 3 (strain Saukett) were purified by Nycodenz gradients and assessed for monodispersity by negative stain EM analysis. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 133333 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4086 pixel / デジタル化 - サイズ - 縦: 4086 pixel / デジタル化 - サンプリング間隔: 14.0 µm / デジタル化 - 画像ごとのフレーム数: 2-33 / 撮影したグリッド数: 1 / 実像数: 2768 / 平均露光時間: 0.2 sec. / 平均電子線量: 1.5 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 詳細 | phenix.real_space_refine was used to refine the atomic model in the cryo-em map. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: Cross-correlation coefficient |
| 得られたモデル |  PDB-5o5b: |
 ムービー
ムービー コントローラー
コントローラー