+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3570 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of deformed wing virus, a honeybee pathogen. | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Deformed wing virus / Picornavirales / Iflaviridae / Iflavirus / viral protein / bee pathogen | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral capsid / RNA helicase activity / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / structural molecule activity / proteolysis / RNA binding / ATP binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Deformed wing virus (ウイルス) Deformed wing virus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Skubnik K / Novacek J | |||||||||
| 資金援助 | 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2017 ジャーナル: Proc Natl Acad Sci U S A / 年: 2017タイトル: Structure of deformed wing virus, a major honey bee pathogen. 著者: Karel Škubník / Jiří Nováček / Tibor Füzik / Antonín Přidal / Robert J Paxton / Pavel Plevka /   要旨: The worldwide population of western honey bees () is under pressure from habitat loss, environmental stress, and pathogens, particularly viruses that cause lethal epidemics. Deformed wing virus (DWV) ...The worldwide population of western honey bees () is under pressure from habitat loss, environmental stress, and pathogens, particularly viruses that cause lethal epidemics. Deformed wing virus (DWV) from the family , together with its vector, the mite , is likely the major threat to the world's honey bees. However, lack of knowledge of the atomic structures of iflaviruses has hindered the development of effective treatments against them. Here, we present the virion structures of DWV determined to a resolution of 3.1 Å using cryo-electron microscopy and 3.8 Å by X-ray crystallography. The C-terminal extension of capsid protein VP3 folds into a globular protruding (P) domain, exposed on the virion surface. The P domain contains an Asp-His-Ser catalytic triad that is, together with five residues that are spatially close, conserved among iflaviruses. These residues may participate in receptor binding or provide the protease, lipase, or esterase activity required for entry of the virus into a host cell. Furthermore, nucleotides of the DWV RNA genome interact with VP3 subunits. The capsid protein residues involved in the RNA binding are conserved among honey bee iflaviruses, suggesting a putative role of the genome in stabilizing the virion or facilitating capsid assembly. Identifying the RNA-binding and putative catalytic sites within the DWV virion structure enables future analyses of how DWV and other iflaviruses infect insect cells and also opens up possibilities for the development of antiviral treatments. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3570.map.gz emd_3570.map.gz | 59.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3570-v30.xml emd-3570-v30.xml emd-3570.xml emd-3570.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3570_fsc.xml emd_3570_fsc.xml | 22.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3570.png emd_3570.png | 258.2 KB | ||
| Filedesc metadata |  emd-3570.cif.gz emd-3570.cif.gz | 6.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3570 http://ftp.pdbj.org/pub/emdb/structures/EMD-3570 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3570 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3570 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3570_validation.pdf.gz emd_3570_validation.pdf.gz | 291.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3570_full_validation.pdf.gz emd_3570_full_validation.pdf.gz | 290.2 KB | 表示 | |
| XML形式データ |  emd_3570_validation.xml.gz emd_3570_validation.xml.gz | 16.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3570 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3570 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3570 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3570 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5mupMC  3574C  3575C  4009C  4014C  5g51C  5g52C  5l7qC  5l8qC  5mv5C  5mv6C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3570.map.gz / 形式: CCP4 / 大きさ: 620.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3570.map.gz / 形式: CCP4 / 大きさ: 620.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.054 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Deformed wing virus
| 全体 | 名称:   Deformed wing virus (ウイルス) Deformed wing virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Deformed wing virus
| 超分子 | 名称: Deformed wing virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 / 詳細: Virus was purified from honeybee pupae / NCBI-ID: 198112 / 生物種: Deformed wing virus / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
| ウイルス殻 | Shell ID: 1 / 直径: 390.0 Å / T番号(三角分割数): 3 |
-分子 #1: VP1
| 分子 | 名称: VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Deformed wing virus (ウイルス) Deformed wing virus (ウイルス) |
| 分子量 | 理論値: 28.679273 KDa |
| 配列 | 文字列: GEESRNTTVL DTTTTLQSSG FGRAFFGEAF NDLKTLMRRY QLYGQLLLSV TTDKDIDHCM FTFPCLPQGL ALDIGSAGSP HEIFNRCRD GIIPLIASGY RFYRGDLRYK IVFPSNVNSN IWVQHRPDRR LEGWSAAKIV NCDAVSTGQG VYNHGYASHI Q ITRVNNVI ...文字列: GEESRNTTVL DTTTTLQSSG FGRAFFGEAF NDLKTLMRRY QLYGQLLLSV TTDKDIDHCM FTFPCLPQGL ALDIGSAGSP HEIFNRCRD GIIPLIASGY RFYRGDLRYK IVFPSNVNSN IWVQHRPDRR LEGWSAAKIV NCDAVSTGQG VYNHGYASHI Q ITRVNNVI ELEVPFYNAT CYNYLQAFNA SSAASSYAVS LGEISVGFQA TSDDIASIVN KPVTIYYSIG DGMQFSQWVG YQ PMMILDQ LPAPVVRAVP E UniProtKB: Genome polyprotein |
-分子 #2: VP2
| 分子 | 名称: VP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Deformed wing virus (ウイルス) Deformed wing virus (ウイルス) |
| 分子量 | 理論値: 28.3609 KDa |
| 配列 | 文字列: MDNPNPGPDG EGEVELEKDS NVVLTTQRDP STSIPAPVSV KWSRWTSNDV VDDYATITSR WYQIAEFVWS KDDPFDKELA RLILPRALL SSIEANSDAI CDVPNTIPFK VHAYWRGDME VRVQINSNKF QVGQLQATWY YSDHENLNIS SKRSVYGFSQ M DHALISAS ...文字列: MDNPNPGPDG EGEVELEKDS NVVLTTQRDP STSIPAPVSV KWSRWTSNDV VDDYATITSR WYQIAEFVWS KDDPFDKELA RLILPRALL SSIEANSDAI CDVPNTIPFK VHAYWRGDME VRVQINSNKF QVGQLQATWY YSDHENLNIS SKRSVYGFSQ M DHALISAS ASNEAKLVIP FKHVYPFLPT RIVPDWTTGI LDMGALNIRV IAPLRMSATG PTTCNVVVFI KLNNSEFTGT SS GKFYASQ IRAKPE UniProtKB: Genome polyprotein |
-分子 #3: VP3
| 分子 | 名称: VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Deformed wing virus (ウイルス) Deformed wing virus (ウイルス) |
| 分子量 | 理論値: 46.697582 KDa |
| 配列 | 文字列: DNPSYQQSPR HFVPTGMHSL ALGTNLVEPL HALRLDAAGT TQHPVGCAPD EDMTVSSIAS RYGLIRRVQW KKDHAKGSLL LQLDADPFV EQRIEGTNPI SLYWFAPVGV VSSMFMQWRG SLEYRFDIIA SQFHTGRLIV GYVPGLTASL QLQMDYMKLK S SSYVVFDL ...文字列: DNPSYQQSPR HFVPTGMHSL ALGTNLVEPL HALRLDAAGT TQHPVGCAPD EDMTVSSIAS RYGLIRRVQW KKDHAKGSLL LQLDADPFV EQRIEGTNPI SLYWFAPVGV VSSMFMQWRG SLEYRFDIIA SQFHTGRLIV GYVPGLTASL QLQMDYMKLK S SSYVVFDL QESNSFTFEV PYVSYRPWWV RKYGGNYLPS STDAPSTLFM YVQVPLIPME AVSDTIDINV YVRGGSSFEV CV PVQPSLG LNWNTDFILR NDEEYRAKTG YAPYYAGVWH SFNNSNSLVF RWGSASDQIA QWPTISVPRG ELAFLRIKDG KQA AVGTQP WRTMVVWPSG HGYNIGIPTY NAERARQLAQ HLYGGGSLTD EKAKQLFVPA NQQGPGKVSN GNPVWEVMRA PLAT QRAHI QDFEFIEAIP E UniProtKB: Genome polyprotein |
-分子 #4: URIDINE-5'-MONOPHOSPHATE
| 分子 | 名称: URIDINE-5'-MONOPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: U5P |
|---|---|
| 分子量 | 理論値: 324.181 Da |
| Chemical component information | 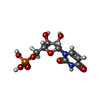 ChemComp-U: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: Dulbeccos Phosphate Buffered Saline D8537 sigma aldrich |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: NITROGEN / 前処理 - 気圧: 0.007 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Virus was incubated in high salt solution containing 0.8 M potassium dihydrogen phosphate, 0.8 M sodium dihydrogen phosphate, 0.1 M sodium HEPES, pH 7.5. After 12 hours incubation was virus dialysed into PBS buffer. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - 画像ごとのフレーム数: 2-16 / 撮影したグリッド数: 1 / 平均露光時間: 1.0 sec. / 平均電子線量: 21.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 74235 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER / 当てはまり具合の基準: R-factor |
|---|---|
| 得られたモデル |  PDB-5mup: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)