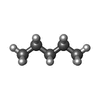+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23361 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | apo SERT reconstituted in lipid nanodisc in KCl | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of cerebellar granule cell precursor proliferation / regulation of thalamus size / Serotonin clearance from the synaptic cleft / serotonergic synapse / positive regulation of serotonin secretion /  cocaine binding / sperm ejaculation / serotonin:sodium:chloride symporter activity / neurotransmitter transmembrane transporter activity / negative regulation of synaptic transmission, dopaminergic ...negative regulation of cerebellar granule cell precursor proliferation / regulation of thalamus size / Serotonin clearance from the synaptic cleft / serotonergic synapse / positive regulation of serotonin secretion / cocaine binding / sperm ejaculation / serotonin:sodium:chloride symporter activity / neurotransmitter transmembrane transporter activity / negative regulation of synaptic transmission, dopaminergic ...negative regulation of cerebellar granule cell precursor proliferation / regulation of thalamus size / Serotonin clearance from the synaptic cleft / serotonergic synapse / positive regulation of serotonin secretion /  cocaine binding / sperm ejaculation / serotonin:sodium:chloride symporter activity / neurotransmitter transmembrane transporter activity / negative regulation of synaptic transmission, dopaminergic / serotonin uptake / cocaine binding / sperm ejaculation / serotonin:sodium:chloride symporter activity / neurotransmitter transmembrane transporter activity / negative regulation of synaptic transmission, dopaminergic / serotonin uptake /  enteric nervous system development / monoamine transmembrane transporter activity / cellular response to cGMP / monoamine transport / negative regulation of organ growth / enteric nervous system development / monoamine transmembrane transporter activity / cellular response to cGMP / monoamine transport / negative regulation of organ growth /  sodium ion binding / sodium ion binding /  serotonin binding / serotonin binding /  血管収縮 / 血管収縮 /  神経伝達物質輸送体 / brain morphogenesis / 神経伝達物質輸送体 / brain morphogenesis /  antiporter activity / antiporter activity /  syntaxin-1 binding / syntaxin-1 binding /  nitric-oxide synthase binding / nitric-oxide synthase binding /  脱分極 / 脱分極 /  social behavior / sodium ion transmembrane transport / negative regulation of neuron differentiation / social behavior / sodium ion transmembrane transport / negative regulation of neuron differentiation /  細胞内膜系 / positive regulation of cell cycle / cellular response to retinoic acid / monoatomic cation channel activity / response to nutrient / 細胞内膜系 / positive regulation of cell cycle / cellular response to retinoic acid / monoatomic cation channel activity / response to nutrient /  記憶 / response to toxic substance / 記憶 / response to toxic substance /  血小板 / 血小板 /  概日リズム / 概日リズム /  actin filament binding / actin filament binding /  integrin binding / response to estradiol / integrin binding / response to estradiol /  presynaptic membrane / presynaptic membrane /  postsynaptic membrane / response to hypoxia / endosome membrane / neuron projection / response to xenobiotic stimulus / postsynaptic membrane / response to hypoxia / endosome membrane / neuron projection / response to xenobiotic stimulus /  脂質ラフト / 脂質ラフト /  focal adhesion / focal adhesion /  シナプス / positive regulation of gene expression / identical protein binding / シナプス / positive regulation of gene expression / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   mouse (ネズミ) mouse (ネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Yang D / Gouaux E | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: Illumination of serotonin transporter mechanism and role of the allosteric site. 著者: Dongxue Yang / Eric Gouaux /  要旨: The serotonin transporter (SERT) terminates serotonin signaling by using sodium and chloride gradients to drive reuptake of serotonin into presynaptic neurons and is the target of widely used ...The serotonin transporter (SERT) terminates serotonin signaling by using sodium and chloride gradients to drive reuptake of serotonin into presynaptic neurons and is the target of widely used medications to treat neuropsychiatric disorders. Despite decades of study, the molecular mechanism of serotonin transport, the coupling to ion gradients, and the role of the allosteric site have remained elusive. Here, we present cryo–electron microscopy structures of SERT in serotonin-bound and serotonin-free states, in the presence of sodium or potassium, resolving all fundamental states of the transport cycle. From the SERT-serotonin complex, we localize the substrate-bound allosteric site, formed by an aromatic pocket positioned in the scaffold domain in the extracellular vestibule, connected to the central site via a short tunnel. Together with elucidation of multiple apo state conformations, we provide previously unseen structural understanding of allosteric modulation, demonstrating how SERT binds serotonin from synaptic volumes and promotes unbinding into the presynaptic neurons. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23361.map.gz emd_23361.map.gz | 229.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23361-v30.xml emd-23361-v30.xml emd-23361.xml emd-23361.xml | 15.2 KB 15.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23361.png emd_23361.png | 122.6 KB | ||
| その他 |  emd_23361_additional_1.map.gz emd_23361_additional_1.map.gz | 120.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23361 http://ftp.pdbj.org/pub/emdb/structures/EMD-23361 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23361 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23361 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23361.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23361.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.661 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: unsharpened map
| ファイル | emd_23361_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
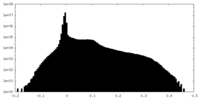
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : apo state of human serotonin transporter in complex with 15B8 Fab...
+超分子 #1: apo state of human serotonin transporter in complex with 15B8 Fab...
+分子 #1: Sodium-dependent serotonin transporter
+分子 #2: variable domain of 15B8 antibody Fab heavy chain
+分子 #3: variable domain of 15B8 antibody Fab light chain
+分子 #5: CHLORIDE ION
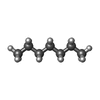
+分子 #6: HEXADECANE
+分子 #7: HEPTANE
+分子 #8: DECANE
+分子 #9: DODECANE
+分子 #10: PENTANE
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 52.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: Ab initio |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 707210 |
 ムービー
ムービー コントローラー
コントローラー



























 Z
Z Y
Y X
X