+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20836 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | neurotensin receptor and arrestin2 complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 neuropeptide receptor binding / G protein-coupled neurotensin receptor activity / inositol phosphate catabolic process / neuropeptide receptor binding / G protein-coupled neurotensin receptor activity / inositol phosphate catabolic process /  angiotensin receptor binding / positive regulation of inhibitory postsynaptic potential / symmetric synapse / D-aspartate import across plasma membrane / positive regulation of gamma-aminobutyric acid secretion / Activation of SMO / negative regulation of interleukin-8 production ... angiotensin receptor binding / positive regulation of inhibitory postsynaptic potential / symmetric synapse / D-aspartate import across plasma membrane / positive regulation of gamma-aminobutyric acid secretion / Activation of SMO / negative regulation of interleukin-8 production ... neuropeptide receptor binding / G protein-coupled neurotensin receptor activity / inositol phosphate catabolic process / neuropeptide receptor binding / G protein-coupled neurotensin receptor activity / inositol phosphate catabolic process /  angiotensin receptor binding / positive regulation of inhibitory postsynaptic potential / symmetric synapse / D-aspartate import across plasma membrane / positive regulation of gamma-aminobutyric acid secretion / Activation of SMO / negative regulation of interleukin-8 production / L-glutamate import across plasma membrane / positive regulation of arachidonic acid secretion / regulation of respiratory gaseous exchange / neuropeptide hormone activity / negative regulation of systemic arterial blood pressure / arrestin family protein binding / G protein-coupled receptor internalization / negative regulation of release of sequestered calcium ion into cytosol / angiotensin receptor binding / positive regulation of inhibitory postsynaptic potential / symmetric synapse / D-aspartate import across plasma membrane / positive regulation of gamma-aminobutyric acid secretion / Activation of SMO / negative regulation of interleukin-8 production / L-glutamate import across plasma membrane / positive regulation of arachidonic acid secretion / regulation of respiratory gaseous exchange / neuropeptide hormone activity / negative regulation of systemic arterial blood pressure / arrestin family protein binding / G protein-coupled receptor internalization / negative regulation of release of sequestered calcium ion into cytosol /  enzyme inhibitor activity / positive regulation of glutamate secretion / positive regulation of inositol phosphate biosynthetic process / enzyme inhibitor activity / positive regulation of glutamate secretion / positive regulation of inositol phosphate biosynthetic process /  temperature homeostasis / Lysosome Vesicle Biogenesis / positive regulation of Rho protein signal transduction / Golgi Associated Vesicle Biogenesis / response to lipid / negative regulation of NF-kappaB transcription factor activity / temperature homeostasis / Lysosome Vesicle Biogenesis / positive regulation of Rho protein signal transduction / Golgi Associated Vesicle Biogenesis / response to lipid / negative regulation of NF-kappaB transcription factor activity /  stress fiber assembly / stress fiber assembly /  regulation of membrane depolarization / negative regulation of Notch signaling pathway / regulation of membrane depolarization / negative regulation of Notch signaling pathway /  仮足 / detection of temperature stimulus involved in sensory perception of pain / negative regulation of interleukin-6 production / positive regulation of receptor internalization / neuropeptide signaling pathway / axon terminus / 仮足 / detection of temperature stimulus involved in sensory perception of pain / negative regulation of interleukin-6 production / positive regulation of receptor internalization / neuropeptide signaling pathway / axon terminus /  クラスリン / negative regulation of protein ubiquitination / クラスリン / negative regulation of protein ubiquitination /  小胞 / 小胞 /  insulin-like growth factor receptor binding / insulin-like growth factor receptor binding /  視覚 / Activated NOTCH1 Transmits Signal to the Nucleus / blood vessel diameter maintenance / 視覚 / Activated NOTCH1 Transmits Signal to the Nucleus / blood vessel diameter maintenance /  GTPase activator activity / Peptide ligand-binding receptors / adult locomotory behavior / positive regulation of release of sequestered calcium ion into cytosol / dendritic shaft / GTPase activator activity / Peptide ligand-binding receptors / adult locomotory behavior / positive regulation of release of sequestered calcium ion into cytosol / dendritic shaft /  学習 / G protein-coupled receptor binding / G protein-coupled receptor activity / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / cytoplasmic vesicle membrane / 学習 / G protein-coupled receptor binding / G protein-coupled receptor activity / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / cytoplasmic vesicle membrane /  terminal bouton / cytoplasmic side of plasma membrane / Signaling by RAF1 mutants / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / Signaling by BRAF and RAF1 fusions / Thrombin signalling through proteinase activated receptors (PARs) / terminal bouton / cytoplasmic side of plasma membrane / Signaling by RAF1 mutants / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / Signaling by BRAF and RAF1 fusions / Thrombin signalling through proteinase activated receptors (PARs) /  protein transport / Cargo recognition for clathrin-mediated endocytosis / protein transport / Cargo recognition for clathrin-mediated endocytosis /  Clathrin-mediated endocytosis / positive regulation of NF-kappaB transcription factor activity / ubiquitin-dependent protein catabolic process / Clathrin-mediated endocytosis / positive regulation of NF-kappaB transcription factor activity / ubiquitin-dependent protein catabolic process /  perikaryon / chemical synaptic transmission / cytoplasmic vesicle / G alpha (s) signalling events / G alpha (q) signalling events / proteasome-mediated ubiquitin-dependent protein catabolic process / perikaryon / chemical synaptic transmission / cytoplasmic vesicle / G alpha (s) signalling events / G alpha (q) signalling events / proteasome-mediated ubiquitin-dependent protein catabolic process /  樹状突起スパイン / 樹状突起スパイン /  transcription coactivator activity / transcription coactivator activity /  receptor ligand activity / positive regulation of ERK1 and ERK2 cascade / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / protein ubiquitination / receptor ligand activity / positive regulation of ERK1 and ERK2 cascade / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / protein ubiquitination /  nuclear body / Ub-specific processing proteases / positive regulation of protein phosphorylation / positive regulation of apoptotic process / nuclear body / Ub-specific processing proteases / positive regulation of protein phosphorylation / positive regulation of apoptotic process /  脂質ラフト / G protein-coupled receptor signaling pathway / lysosomal membrane / 脂質ラフト / G protein-coupled receptor signaling pathway / lysosomal membrane /  ゴルジ体 / negative regulation of gene expression / intracellular membrane-bounded organelle / ゴルジ体 / negative regulation of gene expression / intracellular membrane-bounded organelle /  ubiquitin protein ligase binding / ubiquitin protein ligase binding /  クロマチン / protein-containing complex binding / positive regulation of gene expression / regulation of transcription by RNA polymerase II / negative regulation of apoptotic process / クロマチン / protein-containing complex binding / positive regulation of gene expression / regulation of transcription by RNA polymerase II / negative regulation of apoptotic process /  ゴルジ体 / ゴルジ体 /  細胞膜 / 細胞膜 /  小胞体 / 小胞体 /  シグナル伝達 / positive regulation of transcription by RNA polymerase II シグナル伝達 / positive regulation of transcription by RNA polymerase II類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.2 Å クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Qu QH / Huang W / Masureel M / Janetzko J / Kobilka BK / Skiniotis G | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Structure of the neurotensin receptor 1 in complex with β-arrestin 1. 著者: Weijiao Huang / Matthieu Masureel / Qianhui Qu / John Janetzko / Asuka Inoue / Hideaki E Kato / Michael J Robertson / Khanh C Nguyen / Jeffrey S Glenn / Georgios Skiniotis / Brian K Kobilka /   要旨: Arrestin proteins bind to active, phosphorylated G-protein-coupled receptors (GPCRs), thereby preventing G-protein coupling, triggering receptor internalization and affecting various downstream ...Arrestin proteins bind to active, phosphorylated G-protein-coupled receptors (GPCRs), thereby preventing G-protein coupling, triggering receptor internalization and affecting various downstream signalling pathways. Although there is a wealth of structural information detailing the interactions between GPCRs and G proteins, less is known about how arrestins engage GPCRs. Here we report a cryo-electron microscopy structure of full-length human neurotensin receptor 1 (NTSR1) in complex with truncated human β-arrestin 1 (βarr1(ΔCT)). We find that phosphorylation of NTSR1 is critical for the formation of a stable complex with βarr1(ΔCT), and identify phosphorylated sites in both the third intracellular loop and the C terminus that may promote this interaction. In addition, we observe a phosphatidylinositol-4,5-bisphosphate molecule forming a bridge between the membrane side of NTSR1 transmembrane segments 1 and 4 and the C-lobe of arrestin. Compared with a structure of a rhodopsin-arrestin-1 complex, in our structure arrestin is rotated by approximately 85° relative to the receptor. These findings highlight both conserved aspects and plasticity among arrestin-receptor interactions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20836.map.gz emd_20836.map.gz | 3.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20836-v30.xml emd-20836-v30.xml emd-20836.xml emd-20836.xml | 13.2 KB 13.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20836.png emd_20836.png | 43.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20836 http://ftp.pdbj.org/pub/emdb/structures/EMD-20836 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20836 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20836 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20836.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20836.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : NTSR_Arrestin
| 全体 | 名称: NTSR_Arrestin |
|---|---|
| 要素 |
|
-超分子 #1: NTSR_Arrestin
| 超分子 | 名称: NTSR_Arrestin / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
-分子 #1: ARG-ARG-PRO-TYR-ILE-LEU
| 分子 | 名称: ARG-ARG-PRO-TYR-ILE-LEU / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 819.007 Da |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: RRPYIL |
-分子 #2: Beta-arrestin-1
| 分子 | 名称: Beta-arrestin-1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 39.06284 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: VFKKASPNGK LTVYLGKRDF VDHIDLVDPV DGVVLVDPEY LKERRVYVTL TVAFRYGRED LDVLGLTFRK DLFVANVQSF PPAPEDKKP LTRLQERLIK KLGEHAYPFT FEIPPNLPSS VTLQPGPEDT GKALGVDYEV KAFVAENLEE KIHKRNSVRL V IRKVQYAP ...文字列: VFKKASPNGK LTVYLGKRDF VDHIDLVDPV DGVVLVDPEY LKERRVYVTL TVAFRYGRED LDVLGLTFRK DLFVANVQSF PPAPEDKKP LTRLQERLIK KLGEHAYPFT FEIPPNLPSS VTLQPGPEDT GKALGVDYEV KAFVAENLEE KIHKRNSVRL V IRKVQYAP ERPGPQPTAE TTRQFLMSDK PLHLEASLDK EIYYHGEPIS VNVHVTNNTN KTVKKIKISV RQYADIVLFN TA QYKVPVA MEEADDTVAP SSTFSKVYTL TPFLANNREK RGLALDGKLK HEDTNLASST LLREGANREI LGIIVSYKVK VKL VVSRGG LLGDLASSDV AVELPFTLMH PK |
-分子 #3: Neurotensin receptor type 1
| 分子 | 名称: Neurotensin receptor type 1 / タイプ: protein_or_peptide / ID: 3 詳細: missing regions were not modeled due to local low resolution/flexibility. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 37.360656 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: PSSELDVNTD IYSKVLVTAV YLALFVVGTV GNTVTAFTLA RKKSLQSLQS TVHYHLGSLA LSDLLTLLLA MPVELYNFIW VHHPWAFGD AGCRGYYFLR DACTYATALN VASLSVERYL AICHPFKAKT LMSRSRTKKF ISAIWLASAL LAVPMLFTMG E QNRSADGQ ...文字列: PSSELDVNTD IYSKVLVTAV YLALFVVGTV GNTVTAFTLA RKKSLQSLQS TVHYHLGSLA LSDLLTLLLA MPVELYNFIW VHHPWAFGD AGCRGYYFLR DACTYATALN VASLSVERYL AICHPFKAKT LMSRSRTKKF ISAIWLASAL LAVPMLFTMG E QNRSADGQ HAGGLVCTPT IHTATVKVVI QVNTFMSFIF PMVVISVLNT IIANKLTVMV RQAAEQGQVC TVGGEHSTFS MA IEPGRVQ ALRHGVRVLR AVVIAFVVCW LPYHVRRLMF CYISDEQWTP FLYDFYHYFY MVTNALFYVS STINPILYNL VSA NFRHIF LATLACLC |
-分子 #4: unidentified peptide
| 分子 | 名称: unidentified peptide / タイプ: protein_or_peptide / ID: 4 詳細: actual sequence for this poly-A exists, however, the local resolution for this region is not sufficient to register the amino acids. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 783.958 Da |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #5: [(2R)-2-octanoyloxy-3-[oxidanyl-[(1R,2R,3S,4R,5R,6S)-2,3,6-tris(o...
| 分子 | 名称: [(2R)-2-octanoyloxy-3-[oxidanyl-[(1R,2R,3S,4R,5R,6S)-2,3,6-tris(oxidanyl)-4,5-diphosphonooxy-cyclohexyl]oxy-phosphoryl]oxy-propyl] octanoate タイプ: ligand / ID: 5 / コピー数: 1 / 式: PIO |
|---|---|
| 分子量 | 理論値: 746.566 Da |
| Chemical component information | 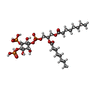 ChemComp-PIO: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: OTHER |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 56.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 254725 |
 ムービー
ムービー コントローラー
コントローラー
































