+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20806 | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single-Particle Cryo-EM Structure of Plasmodium falciparum Chloroquine Resistance Transporter (PfCRT) 7G8 Isoform | ||||||||||||||||||||||||||||||
 マップデータ マップデータ | Sharpened map | ||||||||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||||||||
| 機能・相同性 | Chloroquine-resistance transporter-like / Chloroquine resistance transporter / CRT-like, chloroquine-resistance transporter-like / vacuolar membrane / xenobiotic transmembrane transporter activity / membrane => GO:0016020 / Chloroquine resistance transporter 機能・相同性情報 機能・相同性情報 | ||||||||||||||||||||||||||||||
| 生物種 |   Plasmodium falciparum 7G8 (マラリア病原虫) / Plasmodium falciparum 7G8 (マラリア病原虫) /   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Plasmodium falciparum (isolate 7G8) (マラリア病原虫) Plasmodium falciparum (isolate 7G8) (マラリア病原虫) | ||||||||||||||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Kim J / Tan YZ / Wicht KJ / Erramilli SK / Dhingra SK / Okombo J / Vendome J / Hagenah LM / Giacometti SI / Warren AL ...Kim J / Tan YZ / Wicht KJ / Erramilli SK / Dhingra SK / Okombo J / Vendome J / Hagenah LM / Giacometti SI / Warren AL / Nosol K / Roepe PD / Potter CS / Carragher B / Kossiakoff AA / Quick M / Fidock DA / Mancia F | ||||||||||||||||||||||||||||||
| 資金援助 |  米国, 9件 米国, 9件
| ||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: Structure and drug resistance of the Plasmodium falciparum transporter PfCRT. 著者: Jonathan Kim / Yong Zi Tan / Kathryn J Wicht / Satchal K Erramilli / Satish K Dhingra / John Okombo / Jeremie Vendome / Laura M Hagenah / Sabrina I Giacometti / Audrey L Warren / Kamil Nosol ...著者: Jonathan Kim / Yong Zi Tan / Kathryn J Wicht / Satchal K Erramilli / Satish K Dhingra / John Okombo / Jeremie Vendome / Laura M Hagenah / Sabrina I Giacometti / Audrey L Warren / Kamil Nosol / Paul D Roepe / Clinton S Potter / Bridget Carragher / Anthony A Kossiakoff / Matthias Quick / David A Fidock / Filippo Mancia /  要旨: The emergence and spread of drug-resistant Plasmodium falciparum impedes global efforts to control and eliminate malaria. For decades, treatment of malaria has relied on chloroquine (CQ), a safe and ...The emergence and spread of drug-resistant Plasmodium falciparum impedes global efforts to control and eliminate malaria. For decades, treatment of malaria has relied on chloroquine (CQ), a safe and affordable 4-aminoquinoline that was highly effective against intra-erythrocytic asexual blood-stage parasites, until resistance arose in Southeast Asia and South America and spread worldwide. Clinical resistance to the chemically related current first-line combination drug piperaquine (PPQ) has now emerged regionally, reducing its efficacy. Resistance to CQ and PPQ has been associated with distinct sets of point mutations in the P. falciparum CQ-resistance transporter PfCRT, a 49-kDa member of the drug/metabolite transporter superfamily that traverses the membrane of the acidic digestive vacuole of the parasite. Here we present the structure, at 3.2 Å resolution, of the PfCRT isoform of CQ-resistant, PPQ-sensitive South American 7G8 parasites, using single-particle cryo-electron microscopy and antigen-binding fragment technology. Mutations that contribute to CQ and PPQ resistance localize primarily to moderately conserved sites on distinct helices that line a central negatively charged cavity, indicating that this cavity is the principal site of interaction with the positively charged CQ and PPQ. Binding and transport studies reveal that the 7G8 isoform binds both drugs with comparable affinities, and that these drugs are mutually competitive. The 7G8 isoform transports CQ in a membrane potential- and pH-dependent manner, consistent with an active efflux mechanism that drives CQ resistance, but does not transport PPQ. Functional studies on the newly emerging PfCRT F145I and C350R mutations, associated with decreased PPQ susceptibility in Asia and South America, respectively, reveal their ability to mediate PPQ transport in 7G8 variant proteins and to confer resistance in gene-edited parasites. Structural, functional and in silico analyses suggest that distinct mechanistic features mediate the resistance to CQ and PPQ in PfCRT variants. These data provide atomic-level insights into the molecular mechanism of this key mediator of antimalarial treatment failures. | ||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20806.map.gz emd_20806.map.gz | 95.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20806-v30.xml emd-20806-v30.xml emd-20806.xml emd-20806.xml | 45.7 KB 45.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
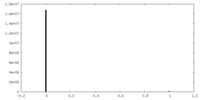
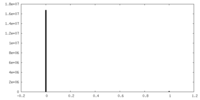
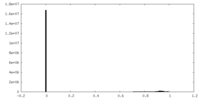
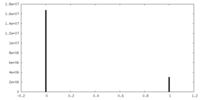
| FSC (解像度算出) |  emd_20806_fsc_1.xml emd_20806_fsc_1.xml emd_20806_fsc_2.xml emd_20806_fsc_2.xml | 11.4 KB 11.4 KB | 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20806.png emd_20806.png | 239.2 KB | ||
| マスクデータ |  emd_20806_msk_1.map emd_20806_msk_1.map emd_20806_msk_2.map emd_20806_msk_2.map | 103 MB 103 MB |  マスクマップ マスクマップ | |
| その他 |  emd_20806_additional_1.map.gz emd_20806_additional_1.map.gz emd_20806_additional_2.map.gz emd_20806_additional_2.map.gz emd_20806_additional_3.map.gz emd_20806_additional_3.map.gz emd_20806_additional_4.map.gz emd_20806_additional_4.map.gz emd_20806_additional_5.map.gz emd_20806_additional_5.map.gz emd_20806_additional_6.map.gz emd_20806_additional_6.map.gz emd_20806_additional_7.map.gz emd_20806_additional_7.map.gz emd_20806_additional_8.map.gz emd_20806_additional_8.map.gz emd_20806_half_map_1.map.gz emd_20806_half_map_1.map.gz emd_20806_half_map_2.map.gz emd_20806_half_map_2.map.gz | 95.5 MB 566.8 KB 13.9 MB 12.1 MB 173.1 KB 14.9 MB 12.6 MB 172.8 KB 15.9 MB 15.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20806 http://ftp.pdbj.org/pub/emdb/structures/EMD-20806 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20806 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20806 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6ukjMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10330 (タイトル: 3.2 Å Single-Particle Cryo-EM Reconstruction of 49 kDa Membrane-Bound PfCRT Complexed with Fab EMPIAR-10330 (タイトル: 3.2 Å Single-Particle Cryo-EM Reconstruction of 49 kDa Membrane-Bound PfCRT Complexed with FabData size: 830.4 Data #1: Unaligned and compressed multi-frame micrographs [micrographs - multiframe] Data #2: Aligned and magnification anisotropy corrected micrographs [micrographs - single frame] Data #3: Final Particle Stack with Refined Euler Angles and Shifts [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20806.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20806.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.035 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
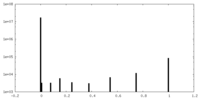
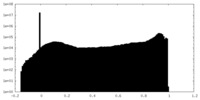
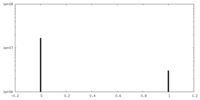
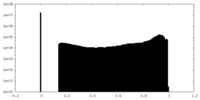
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
+マスク #1
+マスク #2
+追加マップ: Unsharpened map
+追加マップ: Local resolution map
+追加マップ: 3DFSC - PfCRT with Fab, Raw
+追加マップ: 3DFSC - PfCRT with Fab, Thresholded
+追加マップ: 3DFSC - PfCRT with Fab, Thresholded and Binarized
+追加マップ: 3DFSC - PfCRT only, Raw
+追加マップ: 3DFSC - PfCRT Only, Thresholded
+追加マップ: 3DFSC - PfCRT only, Thresholded and binarized
+ハーフマップ: Half map 1
+ハーフマップ: Half map 2
- 試料の構成要素
試料の構成要素
-全体 : Plasmodium falciparum Chloroquine Resistance Transporter (PfCRT) ...
| 全体 | 名称: Plasmodium falciparum Chloroquine Resistance Transporter (PfCRT) 7G8 Isoform Complexed with Fab |
|---|---|
| 要素 |
|
-超分子 #1: Plasmodium falciparum Chloroquine Resistance Transporter (PfCRT) ...
| 超分子 | 名称: Plasmodium falciparum Chloroquine Resistance Transporter (PfCRT) 7G8 Isoform Complexed with Fab タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 102 KDa |
-超分子 #2: Plasmodium falciparum Chloroquine Resistance Transporter (PfCRT)
| 超分子 | 名称: Plasmodium falciparum Chloroquine Resistance Transporter (PfCRT) タイプ: organelle_or_cellular_component / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Plasmodium falciparum 7G8 (マラリア病原虫) Plasmodium falciparum 7G8 (マラリア病原虫) |
| 分子量 | 理論値: 49 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK 293 F Homo sapiens (ヒト) / 組換細胞: HEK 293 F |
-超分子 #3: Antigen-binding fragment (Fab)
| 超分子 | 名称: Antigen-binding fragment (Fab) / タイプ: organelle_or_cellular_component / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 50 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: Chloroquine resistance transporter
| 分子 | 名称: Chloroquine resistance transporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Plasmodium falciparum (isolate 7G8) (マラリア病原虫) Plasmodium falciparum (isolate 7G8) (マラリア病原虫)株: isolate 7G8 |
| 分子量 | 理論値: 53.074461 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGRAKFASKK NNQKNSSKND ERYRELDNLV QEGNGSRLGG GSCLGKCAHV FKLIFKEIKD NIFIYILSII YLSVSVMNTI FAKRTLNKI GNYSFVTSET HNFICMIMFF IVYSLFGNKK GNSKERHRSF NLQFFAISML DACSVILAFI GLTRTTGNIQ S FVLQLSIP ...文字列: MGRAKFASKK NNQKNSSKND ERYRELDNLV QEGNGSRLGG GSCLGKCAHV FKLIFKEIKD NIFIYILSII YLSVSVMNTI FAKRTLNKI GNYSFVTSET HNFICMIMFF IVYSLFGNKK GNSKERHRSF NLQFFAISML DACSVILAFI GLTRTTGNIQ S FVLQLSIP INMFFCFLIL RYRYHLYNYL GAVIIVVTIA LVEMKLSFET QEENSIIFNL VLISSLIPVC FSNMTREIVF KK YKIDILR LNAMVSFFQL FTSCLILPVY TLPFLKQLHL PYNEIWTNIK NGFACLFLGR NTVVENCGLG MAKLCDDCDG AWK TFALFS FFDICDNLIT SYIIDKFSTM TYTIVSCIQG PALAIAYYFK FLAGDVVREP RLLDFVTLFG YLFGSIIYRV GNII LERKK MRNEENEDSE GELTNVDSII TQAAAGGGSG GGSENLYFQG SHHHHHHHHH HWSHPQFEK |
-分子 #2: Fab Heavy Chain
| 分子 | 名称: Fab Heavy Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.737631 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NVSYSYIHWV RQAPGKGLEW VASIYPYSGY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARYGSNYSFW YRGSSVTYAI DYWGQGTLVT VSSASTKGPS VFPLAPSSKS TSGGTAALGC L VKDYFPEP ...文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NVSYSYIHWV RQAPGKGLEW VASIYPYSGY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARYGSNYSFW YRGSSVTYAI DYWGQGTLVT VSSASTKGPS VFPLAPSSKS TSGGTAALGC L VKDYFPEP VTVSWNSGAL TSGVHTFPAV LQSSGLYSLS SVVTVPSSSL GTQTYICNVN HKPSNTKVDK KVEPKSCDKT HT C |
-分子 #3: Fab Light Chain
| 分子 | 名称: Fab Light Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.355898 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQSSTWPITF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQSSTWPITF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #4: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.56 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
詳細: Solution was filtered and degassed. | |||||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 279 K / 装置: LEICA EM GP / 詳細: Blot for 2 seconds before plunging.. | |||||||||
| 詳細 | Protein was incorporated into lipid nanodiscs and complexed with Fab. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 0.001 mm / 最大 デフォーカス(公称値): 0.0018 µm / 最小 デフォーカス(公称値): 0.0012 µm / 倍率(公称値): 215000 Bright-field microscopy / Cs: 0.001 mm / 最大 デフォーカス(公称値): 0.0018 µm / 最小 デフォーカス(公称値): 0.0012 µm / 倍率(公称値): 215000 |
| 特殊光学系 | 球面収差補正装置: Cs corrector was used. / エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-80 / 撮影したグリッド数: 1 / 実像数: 3377 / 平均露光時間: 6.0 sec. / 平均電子線量: 91.56 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 詳細 | Rosetta was used to generate an ab initio model for the PfCRT using the map. 4XMM chains H and L were used as starting point to model the Fab. | ||||||
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL | ||||||
| 得られたモデル |  PDB-6ukj: |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X