+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10335 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CIV1 of the III2-IV5B2 respiratory supercomplex from S. cerevisiae after focused refinement | |||||||||
 マップデータ マップデータ | CIV(5B)-a of III2-IV(5B)2 respiratory supercomplex from S. cerevisiae after focused refinement | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Saccharomyces cerevisiae S288C (パン酵母) Saccharomyces cerevisiae S288C (パン酵母) | |||||||||
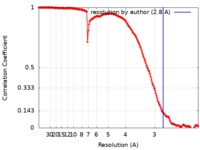
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.8 Å クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Marechal A / Hartley A / Pinotsis N | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2020 ジャーナル: Proc Natl Acad Sci U S A / 年: 2020タイトル: Rcf2 revealed in cryo-EM structures of hypoxic isoforms of mature mitochondrial III-IV supercomplexes. 著者: Andrew M Hartley / Brigitte Meunier / Nikos Pinotsis / Amandine Maréchal /   要旨: The organization of the mitochondrial electron transport chain proteins into supercomplexes (SCs) is now undisputed; however, their assembly process, or the role of differential expression isoforms, ...The organization of the mitochondrial electron transport chain proteins into supercomplexes (SCs) is now undisputed; however, their assembly process, or the role of differential expression isoforms, remain to be determined. In , cytochrome oxidase (CIV) forms SCs of varying stoichiometry with cytochrome (CIII). Recent studies have revealed, in normoxic growth conditions, an interface made exclusively by Cox5A, the only yeast respiratory protein that exists as one of two isoforms depending on oxygen levels. Here we present the cryo-EM structures of the III-IV and III-IV SCs containing the hypoxic isoform Cox5B solved at 3.4 and 2.8 Å, respectively. We show that the change of isoform does not affect SC formation or activity, and that SC stoichiometry is dictated by the level of CIII/CIV biosynthesis. Comparison of the CIV- and CIV-containing SC structures highlighted few differences, found mainly in the region of Cox5. Additional density was revealed in all SCs, independent of the CIV isoform, in a pocket formed by Cox1, Cox3, Cox12, and Cox13, away from the CIII-CIV interface. In the CIV-containing hypoxic SCs, this could be confidently assigned to the hypoxia-induced gene 1 (Hig1) type 2 protein Rcf2. With conserved residues in mammalian Hig1 proteins and Cox3/Cox12/Cox13 orthologs, we propose that Hig1 type 2 proteins are stoichiometric subunits of CIV, at least when within a III-IV SC. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10335.map.gz emd_10335.map.gz | 303.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10335-v30.xml emd-10335-v30.xml emd-10335.xml emd-10335.xml | 19.6 KB 19.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10335_fsc.xml emd_10335_fsc.xml | 15.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10335.png emd_10335.png | 69.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10335 http://ftp.pdbj.org/pub/emdb/structures/EMD-10335 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10335 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10335 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10335.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10335.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CIV(5B)-a of III2-IV(5B)2 respiratory supercomplex from S. cerevisiae after focused refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.085 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : CIV of the III2-IV(5B)2 respiratory supercomplex from S. cerevisi...
+超分子 #1: CIV of the III2-IV(5B)2 respiratory supercomplex from S. cerevisi...
+分子 #1: Cox1 Cytochrome c oxidase subunit 1
+分子 #2: Cox2 Cytochrome c oxidase subunit 2
+分子 #3: Cox3 Cytochrome c oxidase subunit 3
+分子 #4: Cox4 Cytochrome c oxidase subunit 4
+分子 #5: Cox5B Cytochrome c oxidase subunit 5B
+分子 #6: Cox6 Cytochrome c oxidase subunit 6
+分子 #7: Cox7 Cytochrome c oxidase subunit 7
+分子 #8: Cox8 Cytochrome c oxidase polypeptide VIII
+分子 #9: Cox9 Cytochrome c oxidase subunit 7A
+分子 #10: Cox12 Cytochrome c oxidase subunit 6B
+分子 #11: Cox13 Cytochrome c oxidase subunit 6A
+分子 #12: Cox26, Uncharacterized protein YDR119W-A
+分子 #13: RCF2, Respiratory supercomplex factor 2, mitochondrial
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV 詳細: 3 microliter of sample applied to negatively glow discharged grid, blot force -10; blotting time 8.5 sec.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 56.4 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー