[English] 日本語
 Yorodumi
Yorodumi- PDB-5b3s: Bovine heart cytochrome c oxidase in the carbon monoxide-bound mi... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5b3s | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Bovine heart cytochrome c oxidase in the carbon monoxide-bound mixed-valence state at 1.68 angstrom resolution (50 K) | |||||||||
 Components Components | (Cytochrome c oxidase subunit ...) x 13 | |||||||||
 Keywords Keywords | OXIDOREDUCTASE / PROTON PUMP / HEME / RESPIRATORY CHAIN | |||||||||
| Function / homology |  Function and homology information Function and homology informationComplex IV assembly / TP53 Regulates Metabolic Genes / respiratory chain complex IV assembly / Cytoprotection by HMOX1 / mitochondrial respirasome assembly / Respiratory electron transport / respiratory chain complex IV / respiratory chain complex / cytochrome-c oxidase / oxidative phosphorylation ...Complex IV assembly / TP53 Regulates Metabolic Genes / respiratory chain complex IV assembly / Cytoprotection by HMOX1 / mitochondrial respirasome assembly / Respiratory electron transport / respiratory chain complex IV / respiratory chain complex / cytochrome-c oxidase / oxidative phosphorylation / mitochondrial electron transport, cytochrome c to oxygen / cytochrome-c oxidase activity / Mitochondrial protein degradation / enzyme regulator activity / ATP synthesis coupled electron transport / aerobic respiration / central nervous system development / respiratory electron transport chain / oxidoreductase activity / mitochondrial inner membrane / copper ion binding / heme binding / mitochondrion / metal ion binding Similarity search - Function | |||||||||
| Biological species |  | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.68 Å MOLECULAR REPLACEMENT / Resolution: 1.68 Å | |||||||||
 Authors Authors | Shimada, A. / Shinzawa-Ito, K. / Yoshikawa, S. / Tsukihara, T. | |||||||||
| Funding support |  Japan, 1items Japan, 1items
| |||||||||
 Citation Citation |  Journal: To Be Published Journal: To Be PublishedTitle: Bovine heart cytochrome c oxidase in the carbon monoxide-bound mixed-valence state at 1.68 angstrom resolution (50 K) Authors: Shimada, A. / Muramoto, K. / Shinzawa-Ito, K. / Yoshikawa, S. / Tsukihara, T. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5b3s.cif.gz 5b3s.cif.gz | 1.5 MB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5b3s.ent.gz pdb5b3s.ent.gz | 1.3 MB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5b3s.json.gz 5b3s.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/b3/5b3s https://data.pdbj.org/pub/pdb/validation_reports/b3/5b3s ftp://data.pdbj.org/pub/pdb/validation_reports/b3/5b3s ftp://data.pdbj.org/pub/pdb/validation_reports/b3/5b3s | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5b1aS S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Unit cell |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS oper:
|
- Components
Components
-Cytochrome c oxidase subunit ... , 13 types, 26 molecules ANBOCPDQERFSGTHUIVJWKXLYMZ
| #1: Protein | Mass: 57093.852 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #2: Protein | Mass: 26068.404 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #3: Protein | Mass: 29725.328 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #4: Protein | Mass: 16913.367 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #5: Protein | Mass: 12083.727 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #6: Protein | Mass: 10233.528 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #7: Protein | Mass: 9532.667 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #8: Protein | Mass: 9411.600 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #9: Protein | Mass: 8537.019 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #10: Protein | Mass: 6553.546 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #11: Protein/peptide | Mass: 5442.168 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #12: Protein/peptide | Mass: 5362.319 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  #13: Protein/peptide | Mass: 4738.523 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  |
|---|
-Sugars , 1 types, 23 molecules 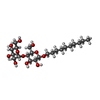
| #27: Sugar | ChemComp-DMU / |
|---|
-Non-polymers , 16 types, 2976 molecules 









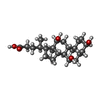




















| #14: Chemical | ChemComp-HEA / #15: Chemical | #16: Chemical | #17: Chemical | #18: Chemical | #19: Chemical | ChemComp-PGV / ( #20: Chemical | ChemComp-EDO / #21: Chemical | ChemComp-TGL / #22: Chemical | #23: Chemical | #24: Chemical | ChemComp-CHD / #25: Chemical | ChemComp-PEK / ( #26: Chemical | ChemComp-CDL / #28: Chemical | #29: Chemical | #30: Water | ChemComp-HOH / | |
|---|
-Details
| Has ligand of interest | N |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 4.06 Å3/Da / Density % sol: 69.69 % |
|---|---|
| Crystal grow | Temperature: 277 K / Method: batch mode / pH: 6.8 Details: 40mM Sodium phosphate, 0.2% decylmaltoside, 2% EG, PEG 4000 |
-Data collection
| Diffraction | Mean temperature: 50 K / Serial crystal experiment: N | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SPring-8 SPring-8  / Beamline: BL44XU / Wavelength: 0.9 Å / Beamline: BL44XU / Wavelength: 0.9 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: RAYONIX MX300HE / Detector: CCD / Date: Jun 5, 2013 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.9 Å / Relative weight: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 1.68→200 Å / Num. obs: 737777 / % possible obs: 99.9 % / Redundancy: 10.2 % / Rmerge(I) obs: 0.09 / Rpim(I) all: 0.028 / Rrim(I) all: 0.094 / Χ2: 1.582 / Net I/av σ(I): 31.952 / Net I/σ(I): 9.7 / Num. measured all: 7497834 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 5B1A Resolution: 1.68→39.624 Å / SU ML: 0.14 / Cross valid method: THROUGHOUT / σ(F): 1.33 / Phase error: 18.67 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.68→39.624 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller





















 PDBj
PDBj

























