+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0504 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of full-length chicken STING in the cGAMP-bound dimeric state | |||||||||||||||||||||
 マップデータ マップデータ | primary map | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | ER /  membrane (生体膜) / membrane (生体膜) /  adaptor / adaptor /  IMMUNE SYSTEM (免疫系) IMMUNE SYSTEM (免疫系) | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報STING mediated induction of host immune responses /  proton channel activity / 2',3'-cyclic GMP-AMP binding / cyclic-di-GMP binding / cGAS/STING signaling pathway / reticulophagy / autophagosome membrane / positive regulation of macroautophagy / proton channel activity / 2',3'-cyclic GMP-AMP binding / cyclic-di-GMP binding / cGAS/STING signaling pathway / reticulophagy / autophagosome membrane / positive regulation of macroautophagy /  autophagosome assembly / autophagosome assembly /  オートファゴソーム ...STING mediated induction of host immune responses / オートファゴソーム ...STING mediated induction of host immune responses /  proton channel activity / 2',3'-cyclic GMP-AMP binding / cyclic-di-GMP binding / cGAS/STING signaling pathway / reticulophagy / autophagosome membrane / positive regulation of macroautophagy / proton channel activity / 2',3'-cyclic GMP-AMP binding / cyclic-di-GMP binding / cGAS/STING signaling pathway / reticulophagy / autophagosome membrane / positive regulation of macroautophagy /  autophagosome assembly / autophagosome assembly /  オートファゴソーム / positive regulation of type I interferon production / positive regulation of interferon-beta production / activation of innate immune response / endoplasmic reticulum-Golgi intermediate compartment membrane / Neutrophil degranulation / protein complex oligomerization / cytoplasmic vesicle / defense response to virus / オートファゴソーム / positive regulation of type I interferon production / positive regulation of interferon-beta production / activation of innate immune response / endoplasmic reticulum-Golgi intermediate compartment membrane / Neutrophil degranulation / protein complex oligomerization / cytoplasmic vesicle / defense response to virus /  ゴルジ体 / ゴルジ体 /  自然免疫系 / endoplasmic reticulum membrane / perinuclear region of cytoplasm / protein homodimerization activity / 自然免疫系 / endoplasmic reticulum membrane / perinuclear region of cytoplasm / protein homodimerization activity /  細胞質 細胞質類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |   Gallus gallus (ニワトリ) Gallus gallus (ニワトリ) | |||||||||||||||||||||
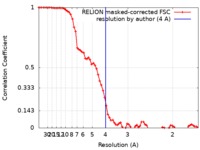
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.0 Å クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Shang G / Zhang C | |||||||||||||||||||||
| 資金援助 |  米国, 6件 米国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: Cryo-EM structures of STING reveal its mechanism of activation by cyclic GMP-AMP. 著者: Guijun Shang / Conggang Zhang / Zhijian J Chen / Xiao-Chen Bai / Xuewu Zhang /  要旨: Infections by pathogens that contain DNA trigger the production of type-I interferons and inflammatory cytokines through cyclic GMP-AMP synthase, which produces 2'3'-cyclic GMP-AMP (cGAMP) that binds ...Infections by pathogens that contain DNA trigger the production of type-I interferons and inflammatory cytokines through cyclic GMP-AMP synthase, which produces 2'3'-cyclic GMP-AMP (cGAMP) that binds to and activates stimulator of interferon genes (STING; also known as TMEM173, MITA, ERIS and MPYS). STING is an endoplasmic-reticulum membrane protein that contains four transmembrane helices followed by a cytoplasmic ligand-binding and signalling domain. The cytoplasmic domain of STING forms a dimer, which undergoes a conformational change upon binding to cGAMP. However, it remains unclear how this conformational change leads to STING activation. Here we present cryo-electron microscopy structures of full-length STING from human and chicken in the inactive dimeric state (about 80 kDa in size), as well as cGAMP-bound chicken STING in both the dimeric and tetrameric states. The structures show that the transmembrane and cytoplasmic regions interact to form an integrated, domain-swapped dimeric assembly. Closure of the ligand-binding domain, induced by cGAMP, leads to a 180° rotation of the ligand-binding domain relative to the transmembrane domain. This rotation is coupled to a conformational change in a loop on the side of the ligand-binding-domain dimer, which leads to the formation of the STING tetramer and higher-order oligomers through side-by-side packing. This model of STING oligomerization and activation is supported by our structure-based mutational analyses. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0504.map.gz emd_0504.map.gz | 27.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0504-v30.xml emd-0504-v30.xml emd-0504.xml emd-0504.xml | 12.4 KB 12.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0504_fsc.xml emd_0504_fsc.xml | 7.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0504.png emd_0504.png | 151.7 KB | ||
| Filedesc metadata |  emd-0504.cif.gz emd-0504.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0504 http://ftp.pdbj.org/pub/emdb/structures/EMD-0504 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0504 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0504 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0504.map.gz / 形式: CCP4 / 大きさ: 29.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0504.map.gz / 形式: CCP4 / 大きさ: 29.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | primary map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : full-length chicken STING
| 全体 | 名称: full-length chicken STING |
|---|---|
| 要素 |
|
-超分子 #1: full-length chicken STING
| 超分子 | 名称: full-length chicken STING / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Gallus gallus (ニワトリ) Gallus gallus (ニワトリ) |
-分子 #1: Stimulator of interferon genes protein
| 分子 | 名称: Stimulator of interferon genes protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Gallus gallus (ニワトリ) Gallus gallus (ニワトリ) |
| 分子量 | 理論値: 44.20707 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MPQDPSTRSS PARLLIPEPR AGRARHAACV LLAVCFVVLF LSGEPLAPII RSVCTQLAAL QLGVLLKGCC CLAEEIFHLH SRHHGSLWQ VLCSCFPPRW YLALLLVGGS AYLDPPEDNG HSPRLALTLS CLCQLLVLAL GLQKLSAVEV SELTESSKKN V AHGLAWSY ...文字列: MPQDPSTRSS PARLLIPEPR AGRARHAACV LLAVCFVVLF LSGEPLAPII RSVCTQLAAL QLGVLLKGCC CLAEEIFHLH SRHHGSLWQ VLCSCFPPRW YLALLLVGGS AYLDPPEDNG HSPRLALTLS CLCQLLVLAL GLQKLSAVEV SELTESSKKN V AHGLAWSY YIGYLKVVLP RLKECMEELS RTNPMLRAHR DTWKLHILVP LGCDIWDDLE KADSNIQYLA DLPETILTRA GI KRRVYKH SLYVIRDKDN KLRPCVLEFA SPLQTLCAMS QDDCAAFSRE QRLEQARLFY RSLRDILGSS KECAGLYRLI AYE EPAEPE SHFLSGLILW HLQQQQREEY MVQEELPLGT SSVELSLQVS SSDLPQPLRS DCPGIHRPDY KDDDDK UniProtKB:  Stimulator of interferon genes protein Stimulator of interferon genes protein |
-分子 #2: cGAMP
| 分子 | 名称: cGAMP / タイプ: ligand / ID: 2 / コピー数: 1 / 式: 1SY |
|---|---|
| 分子量 | 理論値: 674.411 Da |
| Chemical component information |  ChemComp-1SY: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | 前処理 - タイプ: GLOW DISCHARGE / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6nt7: |
 ムービー
ムービー コントローラー
コントローラー