+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23267 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bacterial cellulose synthase BcsB with polyalanine BcsA model | ||||||||||||
 マップデータ マップデータ | Bacterial cellulose synthase BcsB with polyalanine BcsA model | ||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報bacterial cellulose biosynthetic process /  cellulose synthase (UDP-forming) / cellulose synthase (UDP-forming) /  cellulose synthase (UDP-forming) activity / cellulose biosynthetic process / UDP-glucose metabolic process / cyclic-di-GMP binding / cellulose synthase (UDP-forming) activity / cellulose biosynthetic process / UDP-glucose metabolic process / cyclic-di-GMP binding /  hexosyltransferase activity / membrane => GO:0016020 / hexosyltransferase activity / membrane => GO:0016020 /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Escherichia coli K-12 (大腸菌) / Escherichia coli K-12 (大腸菌) /   Escherichia coli (strain K12) (大腸菌) Escherichia coli (strain K12) (大腸菌) | ||||||||||||
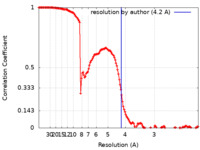
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.2 Å クライオ電子顕微鏡法 / 解像度: 4.2 Å | ||||||||||||
 データ登録者 データ登録者 | Acheson JF / Zimmer J | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2021 ジャーナル: Nat Struct Mol Biol / 年: 2021タイトル: Molecular organization of the E. coli cellulose synthase macrocomplex. 著者: Justin F Acheson / Ruoya Ho / Nicolette F Goularte / Lynette Cegelski / Jochen Zimmer /  要旨: Cellulose is frequently found in communities of sessile bacteria called biofilms. Escherichia coli and other enterobacteriaceae modify cellulose with phosphoethanolamine (pEtN) to promote host tissue ...Cellulose is frequently found in communities of sessile bacteria called biofilms. Escherichia coli and other enterobacteriaceae modify cellulose with phosphoethanolamine (pEtN) to promote host tissue adhesion. The E. coli pEtN cellulose biosynthesis machinery contains the catalytic BcsA-B complex that synthesizes and secretes cellulose, in addition to five other subunits. The membrane-anchored periplasmic BcsG subunit catalyzes pEtN modification. Here we present the structure of the roughly 1 MDa E. coli Bcs complex, consisting of one BcsA enzyme associated with six copies of BcsB, determined by single-particle cryo-electron microscopy. BcsB homo-oligomerizes primarily through interactions between its carbohydrate-binding domains as well as intermolecular beta-sheet formation. The BcsB hexamer creates a half spiral whose open side accommodates two BcsG subunits, directly adjacent to BcsA's periplasmic channel exit. The cytosolic BcsE and BcsQ subunits associate with BcsA's regulatory PilZ domain. The macrocomplex is a fascinating example of cellulose synthase specification. #1:  ジャーナル: Nat.Struct.Mol.Biol. / 年: 2021 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2021タイトル: Molecular Organization of the E. coli Cellulose Synthase Macrocomplex 著者: Acheson JF / Ho R / Goularte NF / Cegelski L / Zimmer J | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23267.map.gz emd_23267.map.gz | 229.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23267-v30.xml emd-23267-v30.xml emd-23267.xml emd-23267.xml | 19.3 KB 19.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23267_fsc.xml emd_23267_fsc.xml | 14 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23267.png emd_23267.png | 51.9 KB | ||
| マスクデータ |  emd_23267_msk_1.map emd_23267_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23267 http://ftp.pdbj.org/pub/emdb/structures/EMD-23267 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23267 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23267 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7lbyMC  7l2zC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10627 (タイトル: Poly-alanine backbone model of E. coli BcsA bound to BcsB EMPIAR-10627 (タイトル: Poly-alanine backbone model of E. coli BcsA bound to BcsBData size: 3.2 TB Data #1: Unaligned multi-frame micrographs [micrographs - multiframe] Data #2: Unaligned movie frames [micrographs - multiframe] / Data #3: Unaligned movie frames [micrographs - multiframe] / Data #4: Unaligned movie frames [micrographs - multiframe] / Data #5: Unaligned movie frames [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23267.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23267.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Bacterial cellulose synthase BcsB with polyalanine BcsA model | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23267_msk_1.map emd_23267_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Bacterial cellulose synthase BcsB with BcsA
| 全体 | 名称: Bacterial cellulose synthase BcsB with BcsA |
|---|---|
| 要素 |
|
-超分子 #1: Bacterial cellulose synthase BcsB with BcsA
| 超分子 | 名称: Bacterial cellulose synthase BcsB with BcsA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: BcsB monomer with BcsA refined using masked local refinement and signal subtraction from E coli BCS inner-membrane complex projections. |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) / 細胞中の位置: inner membrane Escherichia coli K-12 (大腸菌) / 細胞中の位置: inner membrane |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) / 組換株: C43 / 組換プラスミド: petduet_BcsA_BcsB_AdrA Escherichia coli BL21(DE3) (大腸菌) / 組換株: C43 / 組換プラスミド: petduet_BcsA_BcsB_AdrA |
| 分子量 | 理論値: 180 KDa |
-分子 #1: Cellulose synthase catalytic subunit [UDP-forming]
| 分子 | 名称: Cellulose synthase catalytic subunit [UDP-forming] / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号:  cellulose synthase (UDP-forming) cellulose synthase (UDP-forming) |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (strain K12) (大腸菌) / 株: K12 Escherichia coli (strain K12) (大腸菌) / 株: K12 |
| 分子量 | 理論値: 101.67125 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: GSILTRWLLI PPVNARLIGR YRDYRRHGAS AFSATLGCFW MILAWIFIPL EHPRWQRIRA EHKNLYPHIN ASRPRPLDPV RYLIQTCWL LIGASRKETP KPRRRAFSGL QNIRGRYHQW MNELPERVSH KTQHLDEKKE LGHLSAGARR LILGIIVTFS L ILALICVT ...文字列: GSILTRWLLI PPVNARLIGR YRDYRRHGAS AFSATLGCFW MILAWIFIPL EHPRWQRIRA EHKNLYPHIN ASRPRPLDPV RYLIQTCWL LIGASRKETP KPRRRAFSGL QNIRGRYHQW MNELPERVSH KTQHLDEKKE LGHLSAGARR LILGIIVTFS L ILALICVT QPFNPLAQFI FLMLLWGVAL IVRRMPGRFS ALMLIVLSLT VSCRYIWWRY TSTLNWDDPV SLVCGLILLF AE TYAWIVL VLGYFQVVWP LNRQPVPLPK DMSLWPSVDI FVPTYNEDLN VVKNTIYASL GIDWPKDKLN IWILDDGGRE EFR QFAQNV GVKYIARTTH EHAKAGNINN ALKYAKGEFV SIFDCDHVPT RSFLQMTMGW FLKEKQLAMM QTPHHFFSPD PFER NLGRF RKTPNEGTLF YGLVQDGNDM WDATFFCGSC AVIRRKPLDE IGGIAVETVT EDAHTSLRLH RRGYTSAYMR IPQAA GLAT ESLSAHIGQR IRWARGMVQI FRLDNPLTGK GLKFAQRLCY VNAMFHFLSG IPRLIFLTAP LAFLLLHAYI IYAPAL MIA LFVLPHMIHA SLTNSKIQGK YRHSFWSEIY ETVLAWYIAP PTLVALINPH KGKFNVTAKG GLVEEEYVDW VISRPYI FL VLLNLVGVAV GIWRYFYGPP TEMLTVVVSM VWVFYNLIVL GGAVAVSVES KQVRRSHRVE MTMPAAIARE DGHLFSCT V QDFSDGGLGI KINGQAQILE GQKVNLLLKR GQQEYVFPTQ VARVMGNEVG LKLMPLTTQQ HIDFVQCTFA RADTWALWQ DSYPEDKPLE SLLDILKLGF RGYRHLAEFA PSSVKGIFRV LTSLVSWVVS FIPPRPERSE TAQPSDQALA QQHHHHHHLE HHHHHH |
-分子 #2: Cyclic di-GMP-binding protein
| 分子 | 名称: Cyclic di-GMP-binding protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (strain K12) (大腸菌) / 株: K12 Escherichia coli (strain K12) (大腸菌) / 株: K12 |
| 分子量 | 理論値: 86.101289 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MKRKLFWICA VAMGMSAFPS FMTQATPATQ PLINAEPAVA AQTEQNPQVG QVMPGVQGAD APVVAQNGPS RDVKLTFAQI APPPGSMVL RGINPNGSIE FGMRSDEVVT KAMLNLEYTP SPSLLPVQSQ LKVYLNDELM GVLPVTKEQL GKKTLAQMPI N PLFISDFN ...文字列: MKRKLFWICA VAMGMSAFPS FMTQATPATQ PLINAEPAVA AQTEQNPQVG QVMPGVQGAD APVVAQNGPS RDVKLTFAQI APPPGSMVL RGINPNGSIE FGMRSDEVVT KAMLNLEYTP SPSLLPVQSQ LKVYLNDELM GVLPVTKEQL GKKTLAQMPI N PLFISDFN RVRLEFVGHY QDVCEKPAST TLWLDVGRSS GLDLTYQTLN VKNDLSHFPV PFFDPSDNRT NTLPMVFAGA PD VGLQQAS AIVASWFGSR SGWRGQNFPV LYNQLPDRNA IVFATNDKRP DFLRDHPAVK APVIEMINHP QNPYVKLLVV FGR DDKDLL QAAKGIAQGN ILFRGESVVV NEVKPLLPRK PYDAPNWVRT DRPVTFGELK TYEEQLQSSG LEPAAINVSL NLPP DLYLM RSTGIDMDIN YRYTMPPVKD SSRMDISLNN QFLQSFNLSS KQEANRLLLR IPVLQGLLDG KTDVSIPALK LGATN QLRF DFEYMNPMPG GSVDNCITFQ PVQNHVVIGD DSTIDFSKYY HFIPMPDLRA FANAGFPFSR MADLSQTITV MPKAPN EAQ METLLNTVGF IGAQTGFPAI NLTVTDDGST IQGKDADIMI IGGIPDKLKD DKQIDLLVQA TESWVKTPMR QTPFPGI VP DESDRAAETR STLTSSGAMA AVIGFQSPYN DQRSVIALLA DSPRGYEMLN DAVNDSGKRA TMFGSVAVIR ESGINSLR V GDVYYVGHLP WFERVWYALA NHPILLAVLA AISVILLAWV LWRLLRIISR RRLNPDNE |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: All buffers made fresh from concentrated stock. A 10% LMNG 2% DMNG 2% CHS solution was made made in milliQ water without buffer. After rocking for one day at room temp the solution was ...詳細: All buffers made fresh from concentrated stock. A 10% LMNG 2% DMNG 2% CHS solution was made made in milliQ water without buffer. After rocking for one day at room temp the solution was briefly sonicated to completely dissolve the CHS. | ||||||||||||||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER / 詳細: 2 drops amylamine added to the chamber | ||||||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: 3.5 uL applied to c-flat 1.2/1.3 300 mesh grids incubated for 30s then blotted using force 6 for 12 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.5 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 81000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.5 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 81000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 10 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 2964 / 平均電子線量: 51.0 e/Å2 / 詳細: movie mode 40 frames |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 101.45 / 当てはまり具合の基準: CC |
|---|---|
| 得られたモデル |  PDB-7lby: |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X